Pembelajaran mendalam tentang bahan baku enzim inti yang digunakan dalam Deteksi PCR SARS-CoV-2
Sejak Maret 2022, strain mutan Omicron yang licik sekali lagi menghancurkan kehidupan damai masyarakat, epidemi virus corona baru merebak di seluruh negeri dan memengaruhi 30 provinsi (daerah otonom dan kotamadya). Sebagai cara efektif pencegahan dan pengendalian epidemi SARS-CoV-2 yang tepat, deteksi asam nukleat telah menjadi cara hidup yang normal. "Apakah Anda sudah melakukan tes asam nukleat hari ini?" Hal itu juga telah menjadi sapaan sehari-hari masyarakat. Ngomong-ngomong, tahukah Anda bahan baku inti apa yang dibutuhkan dalam deteksi asam nukleat? Artikel ini akan memperkenalkan bahan baku inti yang penting dalam enzim deteksi asam nukleat.
1. Proses deteksi asam nukleat untuk SARS-CoV-2
2. Enzim inti dalam ekstraksi asam nukleat
3. Enzim inti selama RT-qPCR
4. Enzim Inti Deteksi Asam Nukleat SARS-CoV-2 dari
1. Proses deteksi asam nukleat untuk SARS-CoV-2
Enzim merupakan golongan biokatalis yang sangat penting dengan efisiensi katalitik dan spesifisitas reaksi yang tinggi. Sebagian besar reaksi biokimia memerlukan partisipasi enzim. Dalam proses deteksi asam nukleat 2019-nCoV (ditunjukkan pada Gambar 1), berbagai jenis enzim molekuler memainkan peran penting dalam berbagai tahap eksperimen seperti ekstraksi asam nukleat dan RT-qPCR. Selanjutnya, menurut berbagai kaitan eksperimen dalam deteksi asam nukleat, kami akan memilah bahan baku enzim inti yang digunakan dalam proses deteksi asam nukleat.
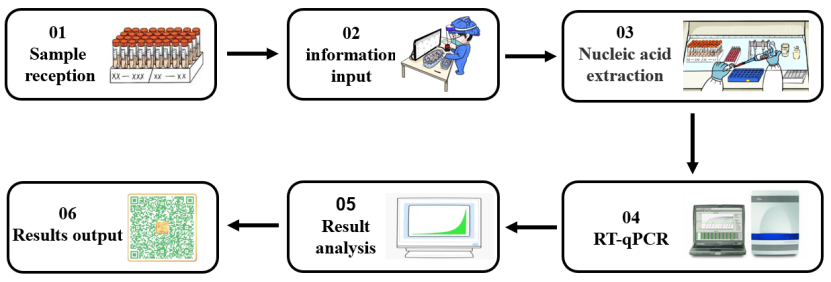
Gambar 1. Proses deteksi asam nukleat untuk SARS-CoV-2
2. Enzim inti dalam ekstraksi asam nukleat
Proses ekstraksi asam nukleat virus corona baru terutama mencakup dua langkah: lisis dan pemurnian. Lisis adalah proses penghancuran struktur sel sampel sehingga asam nukleat dalam sampel bebas dalam sistem lisis; Pemurnian adalah pemisahan lengkap asam nukleat dari komponen lain dalam sistem lisis, seperti protein, garam, dan pengotor lainnya, dan proses reaksi memerlukan partisipasi proteinase K, deoksiribonuklease I, dan inhibitor RNase.
2.1 Protease K
Proteinase K merupakan protease serin dengan aktivitas pembelahan luas, tempat pembelahannya adalah ikatan peptida karboksi-terminal dari asam amino alifatik dan aromatik (Gambar 2). Dalam proses ekstraksi asam nukleat, proteinase K dapat mendegradasi histon yang terikat erat dengan asam nukleat, mendorong pemisahan asam nukleat, dan membuat sampel asam nukleat lebih mudah diekstraksi. Selain itu, proteinase K dapat mendegradasi aktivitas RNA hidrolase (RNase) dan menghambat hidrolisis RNA templat oleh RNase.
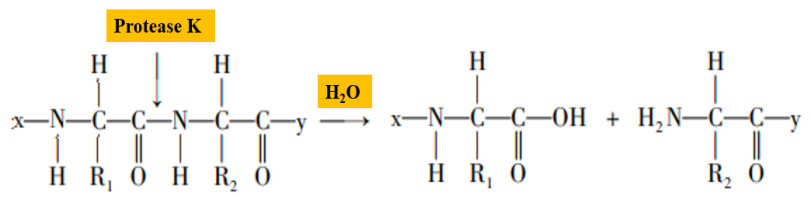
Gambar 2. Diagram skema proteinase K yang menghidrolisis ikatan peptida
2.2 Deoksiribonuklease I
Deoksiribonuklease I (DNase I) dapat mengkatalisis berbagai bentuk DNA, menargetkan pembelahan ikatan fosfodiester yang berdekatan dengan pirimidin, dan menghasilkan polinukleotida dengan gugus fosfat pada ujung 5' dan gugus hidroksil pada ujung 3', produk pencernaan rata-rata adalah politetranukleotida terkecil.Dalam proses ekstraksi asam nukleat SARS-CoV-2, DNase I terutama digunakan untuk menghilangkan kontaminasi genom dalam sampel RNA, menghindari residu DNA dalam templat RNA dan meningkatkan kemurnian templat.
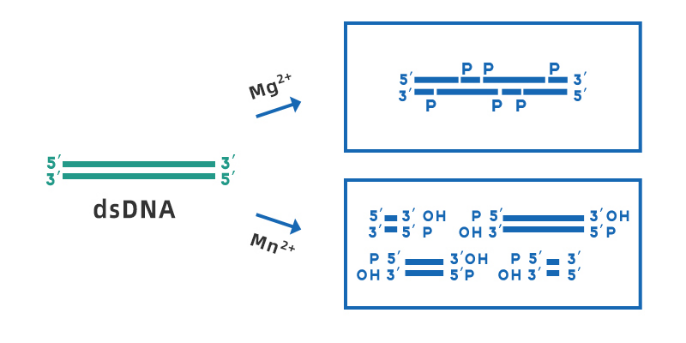
Gambar 3. Diagram skema DNase I yang membelah dsDNA dengan adanya Mg2+ dan Mn2+
2.3 Penghambat RNase
Dalam proses deteksi asam nukleat SARS-CoV-2, ekstraksi dan pemurnian sampel asam nukleat atau persiapan sistem reaksi eksperimental dapat menimbulkan kontaminasi ribonuklease (RNase), yang mengakibatkan degradasi templat RNA. Untuk menghindari kontaminasi RNase, diperlukan Inhibitor RNase.
RNase Inhibitor adalah inhibitor RNase spesifik dalam plasenta manusia, yang secara spesifik dapat mengikat RNase untuk membentuk kompleks dengan ikatan non-kovalen dan menonaktifkan RNase.
3. Enzim inti selama RT-qPCR
Setelah ekstraksi asam nukleat dari sampel SARS-CoV-2 selesai, deteksi asam nukleat dapat diselesaikan dengan RT-qPCR. Dalam proses percobaan ini, DNA polimerase, reverse transcriptase, dan urasil DNA glikosidase semuanya merupakan bahan baku enzim inti yang penting.
3.1 Transkriptase balik
Setelah ekstraksi dan pemurnian, RNA SARS-CoV-2 membutuhkan reverse transcriptase untuk mengkatalisis polimerisasi dNTP guna menghasilkan urutan cDNA yang komplementer dengan RNA cetakan (Gambar 4). Untuk reaksi RT-qPCR, reverse transcriptase yang tahan suhu tinggi harus dipilih. Saat ini, reverse transcriptase MMLV adalah yang paling banyak digunakan, karena kurangnya aktivitas endonuklease DNA dan aktivitas RNase H yang rendah, ia memiliki lebih banyak keuntungan dalam aplikasi kloning cDNA.
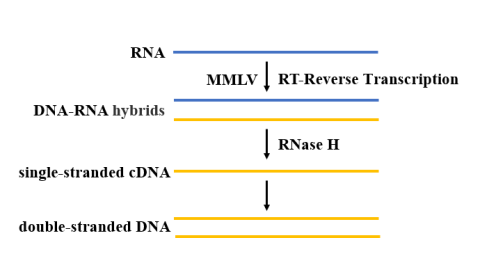
Gambar 4. Diagram skema proses transkripsi terbalik
3.2 DNA polimerase
Setelah proses transkripsi balik templat selesai dilakukan untuk menghasilkan cDNA untai ganda, DNA polimerase "pemain jiwa" dalam reaksi PCR diperlukan untuk muncul, dengan mempolimerisasi deoksiribonukleotida bebas untuk memperpanjang rantai DNA, dan sejumlah besar DNA templat diperkuat secara in vitro untuk mencapai tujuan deteksi asam nukleat virus.
DNA polimerase yang umum digunakan dalam reaksi RT-qPCR adalah DNA polimerase Taq hot-start. Jenis enzim ini tidak aktif pada suhu ruangan.Ia memiliki aktivitas polimerisasi hanya setelah hot-starting, yang dapat meminimalkan pembentukan sinyal latar belakang. Ia memecahkan masalah amplifikasi non-spesifik yang disebabkan oleh pembentukan primer-dimer atau ketidakcocokan dalam reaksi PCR konvensional. Saat ini, metode modifikasi hot-start DNA polimerase yang umum digunakan terutama mencakup modifikasi kimia, modifikasi ligan, dan modifikasi antibodi. Prinsip berbagai metode modifikasi hot-start ditunjukkan pada Gambar 5.
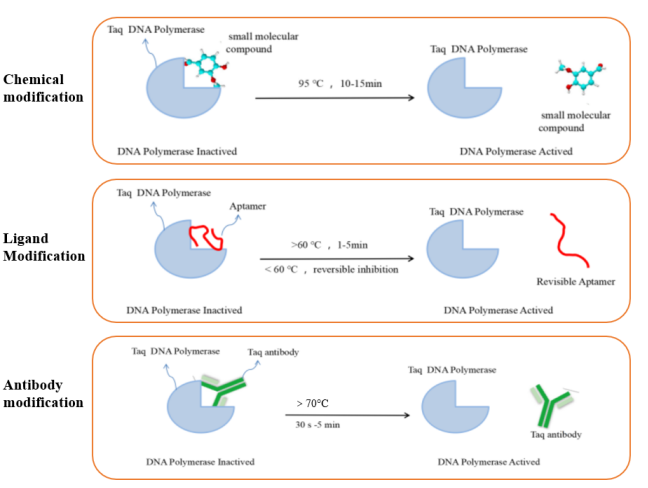
Gambar 5. Diagram skema berbagai jenis enzim hot-start yang dimodifikasi
3.3 Urasil DNA glikosidase
Dalam proses deteksi asam nukleat virus korona baru, polusi aerosol di lingkungan operasi merupakan faktor paling umum yang menyebabkan hasil PCR positif palsu. Penambahan enzim UDG (Uracil DNA Glycosylase, uracil DNA glycosylase) ke sistem amplifikasi dapat secara efektif menghilangkan polutan sisa amplifikasi (sebagian besar dalam bentuk aerosol) yang tercampur dalam sistem PCR untuk memastikan keakuratan hasil amplifikasi. Prinsip anti-polusi enzim UDG ditunjukkan pada Gambar 6.
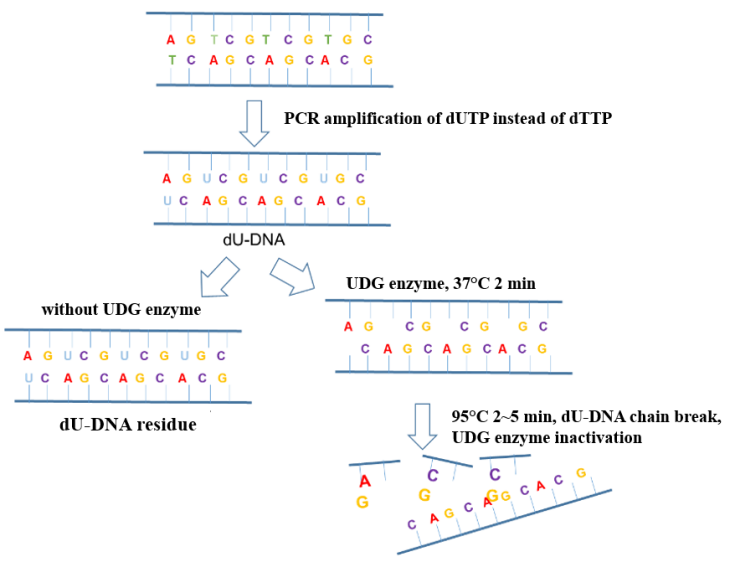
Gambar 6. Diagram skema prinsip anti polusi enzim UDG
4.Enzim Inti Deteksi Asam Nukleat SARS-CoV-2 dari Yeasen
| Proses | Keterangan | Nama Produk | SKU |
| Pengolahan sampel | Pencernaan protein | 10401ES | |
| ekstraksi RNA | Rekombinan DNAse saya (Bebas RNase) (Tanyakan) | 10325ES | |
| Penghambatan RNase | Penghambat RNase Murine (40 U/μL) | 10603ES | |
| Transkripsi terbalik | Cocok untuk RT-qPCR | Hifair™ V Transkriptase Terbalik (200 U/L) | 11300ES |
| HifairTM V Reverse Transcriptase (600U/ μL) Bebas GLyceroL (Tanyakan) | 11301ES | ||
| Amplifikasi PCR | DNA polimerase awal yang panas | 10726ES | |
| UDG Termal | Uracil DNA Glycosylase (UDG/UNG), tahan panas, 1 U/μL | 10303ES |
Mengenai membaca:
Pemilihan Transkriptase Terbalik
Inhibitor Murine RNase-Berhasil menghilangkan kontaminasi RNase dan melestarikan RNA
