คุณได้เรียนรู้วิธีสร้างไลบรารี PCR-Free แล้วหรือยัง?
ในการจัดลำดับปริมาณงานสูง การสร้างไลบรารีแบบเดิมต้องใช้การขยาย PCR ในแง่หนึ่ง มันคือการขยายตัวอย่าง DNA ร่องรอยและเพิ่มผลผลิตของไลบรารี ในอีกแง่หนึ่ง มันสามารถขยายสัญญาณฟลูออเรสเซนต์ ทำให้เครื่องจัดลำดับจับและระบุสัญญาณฟลูออเรสเซนต์ได้ง่ายขึ้นเพื่อปรับปรุงความแม่นยำของการจัดลำดับ อย่างไรก็ตาม PCR เปรียบเสมือน "ดาบสองคม" ในขณะที่แก้ปัญหาปริมาณตัวอย่างเริ่มต้นต่ำและขยายสัญญาณฟลูออเรสเซนต์ มันยังทำให้เกิดข้อผิดพลาดและอคติในการขยาย และไม่สามารถแสดง "หน้าจริง" ของลำดับจีโนมได้อย่างสมบูรณ์แบบ นอกจากนี้ โพลีเมอเรส PCR ยังมีอคติในการขยายบางอย่าง บางภูมิภาค โดยเฉพาะ GC สูงหรือ GC ต่ำ โครงสร้างรอง และภูมิภาคอื่นๆ มีประสิทธิภาพในการขยายต่ำและครอบคลุมได้ยาก นอกจากนี้ยังจะนำ InDels ที่ไม่ถูกต้องจำนวนมากเข้ามาและนำไปสู่การทำซ้ำจำนวนมาก ทำให้ปริมาณข้อมูล DNA สูญเปล่าและเพิ่มต้นทุนการจัดลำดับ ดังนั้นการนำเทคโนโลยี PCR-Free มาใช้จึงไม่เพียงแต่มีข้อดีของ PCR เท่านั้น แต่ยังช่วยแก้ไขข้อบกพร่องของ PCR ได้อีกด้วย เทคโนโลยีนี้ไม่เพียงแต่ทำให้ได้ไลบรารีคุณภาพสูงที่มีความไวสูง และสามารถตรวจจับตัวอย่างร่องรอยได้เท่านั้น แต่ยังมีความแม่นยำสูงและลดภาระงานการตรวจสอบติดตามผลสำหรับการศึกษาตัวแปรอีกด้วย แล้วไลบรารี PCR-Free คืออะไรกันแน่ และมีข้อดีอะไรบ้าง?
1. PCR-Free library คืออะไร?
2. เทคโนโลยี PCR-Free มีข้อดีอะไรบ้าง?
3. การแสดงข้อมูล PCR-Free
4. คำถามที่พบบ่อย
5. ข้อมูลผลิตภัณฑ์
6. เรื่องการอ่านหนังสือ
1. PCR-Free library คืออะไร?
เทคโนโลยีการจัดลำดับรุ่นถัดไปเป็นเทคโนโลยีที่ใช้กันทั่วไปมากที่สุดในการวิจัยการจัดลำดับที่มีปริมาณงานสูงในชีววิทยาโมเลกุลสมัยใหม่ การจัดลำดับรุ่นแรกแบบดั้งเดิมไม่สามารถตอบสนองความต้องการของนักวิจัยได้อย่างเต็มที่อีกต่อไป การจัดลำดับจีโนมใหม่ของสิ่งมีชีวิตจำลองและการจัดลำดับจีโนมของสิ่งมีชีวิตที่ไม่ใช่แบบจำลองนั้นมีค่าใช้จ่ายที่สูงกว่า เทคโนโลยีการจัดลำดับที่มีปริมาณงานต่ำและเร็วกว่า จึงทำให้เกิดเทคโนโลยีการจัดลำดับรุ่นที่สอง หลักการสำคัญของเทคโนโลยีการจัดลำดับรุ่นที่สองคือการจัดลำดับโดยการสังเคราะห์ นั่นคือ การกำหนดลำดับดีเอ็นเอโดยการรวบรวมข้อมูลพื้นฐานที่ติดฉลากปลายที่เพิ่งสังเคราะห์ขึ้น หลักการสำคัญของเทคโนโลยีการจัดลำดับรุ่นที่สองสำหรับการจัดลำดับดีเอ็นเอคือ การแยกส่วนดีเอ็นเอก่อน ซ่อมแซมปลายของดีเอ็นเอที่แยกส่วน จากนั้นจึงเพิ่มอะแดปเตอร์เฉพาะทั้งสองด้าน จากนั้นจึงใช้วิธีการต่างๆ เพื่อสร้าง PCR ที่ตรึงตามพื้นที่จำนวนล้านๆ ชิ้น สำหรับอาร์เรย์โคลน ข้อมูลการเรียงลำดับจะได้รับโดยใช้ไพรเมอร์ไฮบริดิเซชันและปฏิกิริยาการขยายด้วยเอนไซม์เพื่อสร้างภาพฉลากเรืองแสงที่รวมอยู่ในปฏิกิริยาการขยายแต่ละปฏิกิริยา
การจัดลำดับดีเอ็นเอโดยใช้การจัดลำดับรุ่นถัดไปประกอบด้วยกระบวนการหลักสองขั้นตอน ได้แก่ การเตรียมไลบรารีและการจัดลำดับบนเครื่องจักร ในระหว่างการเตรียมไลบรารี ชิ้นส่วนจีโนมที่ถูกขัดจังหวะแบบสุ่มมักจะถูกขยายโดย PCR มาตรฐาน อย่างไรก็ตาม สำหรับเทมเพลตพิเศษบางรายการ มีปัจจัยบางอย่าง เช่น โครงสร้างรองที่ซับซ้อนหรือเสถียรภาพทางความร้อนต่ำ ซึ่งส่งผลต่อความต้องการในการขยายของ PCR เทมเพลต ดังนั้นลำดับจีโนมทั้งหมดจึงไม่สามารถสะท้อนได้อย่างเท่าเทียมกันในไลบรารีการขยาย PCR โดยเฉพาะอย่างยิ่งสำหรับเทมเพลตบางรายการที่มี GC สูงหรือเนื้อหา AT สูง บางครั้งการใช้เทคนิค PCR เพื่อขยายเพื่อสร้างไลบรารีอาจเป็นเรื่องยาก ไม่มีข้อแตกต่างระหว่างไลบรารีที่ปราศจาก PCR และไลบรารี PCR ทั่วไปเมื่อจัดลำดับบนเครื่องจักร ยกเว้นว่าจะไม่ดำเนินการ PCR ในระหว่างกระบวนการสร้างไลบรารี ไลบรารีที่ปราศจาก PCR สามารถปรับปรุงการกระจายของการอ่านข้อมูลในทางทฤษฎีและสร้างการครอบคลุมจีโนมที่สม่ำเสมอมากขึ้นไลบรารีที่ปราศจาก PCR เป็นส่วนเสริมของวิธีการสร้างไลบรารี เนื่องจากไลบรารีนี้ถูกจัดเตรียมโดยตรงในไลบรารีออนบอร์ดโดยไม่ต้องขยาย PCR จึงสามารถปรับปรุงการครอบคลุมของภูมิภาค GC สูงหรือ AT สูงบางส่วนได้ จึงลดฐานข้อผิดพลาดที่เกิดจาก PCR อคติของข้อมูล และการทำซ้ำลำดับ
ขั้นตอนหลักของการสร้างไลบรารี NGS ทั่วไป ได้แก่ การแบ่งส่วนของจีโนม การซ่อมแซมปลาย การเพิ่มอะแดปเตอร์ PCR และการขยายสัญญาณก่อนการจัดลำดับ แล้วการสร้างไลบรารีที่ไม่ใช้ PCR ล่ะ? ตามชื่อที่บ่งบอก นี่คือกระบวนการสร้างไลบรารีที่ไม่ต้องใช้ PCR ขั้นตอนหลักของการสร้างไลบรารี ได้แก่ การแบ่งส่วนของจีโนม การปรับเปลี่ยนปลาย การเพิ่มลิงก์เกอร์ และการควบคุมคุณภาพไลบรารี แต่ในความเป็นจริง ขั้นตอนเหล่านี้สามารถถือได้ว่าไม่ใช้ PCR ในกระบวนการสร้างไลบรารีเท่านั้น การไม่ใช้ PCR ที่แท้จริงควรเป็นกระบวนการตั้งแต่การสร้างไลบรารีไปจนถึงการจัดลำดับโดยไม่ต้องขยายไลบรารี (เช่น เทคโนโลยีการจัดลำดับโมเลกุลเดี่ยว) หรือเทคโนโลยีการจัดลำดับที่ไม่มีการสะสมข้อผิดพลาดในการขยาย PCR (เช่น DNBseq ของ MGI)

รูปที่ 1 แผนผังกระบวนการสร้างห้องสมุดแบบปกติและการสร้างห้องสมุดแบบปลอด PCR
2. เทคโนโลยี PCR-Free มีข้อดีอะไรบ้าง?
ในกระบวนการสร้างห้องสมุดตามปกติ เอนไซม์ขยายอาจก่อให้เกิดข้อผิดพลาดได้ การขยายแบบวนรอบจะทำให้ข้อผิดพลาดเหล่านี้สะสมและขยายเพิ่มขึ้น ทำให้ความแม่นยำของการจำลองลำดับดีเอ็นเอลดลง นอกจากนี้ DNA โพลิเมอเรสยังมีอคติในการขยายในระดับหนึ่ง โดยเฉพาะอย่างยิ่งสำหรับบริเวณที่มีเนื้อหา GC หรือโครงสร้างรองแตกต่างกันมาก ส่งผลให้ประสิทธิภาพในการขยายต่ำและครอบคลุมข้อมูลได้ไม่ดี ในขณะที่นำ InDels ที่ไม่ถูกต้องเข้ามา ก็ยังทำให้เกิดการทำซ้ำจำนวนมากซึ่งส่งผลให้ปริมาณข้อมูลดีเอ็นเอสูญเปล่าและเพิ่มต้นทุนการจัดลำดับ เทคโนโลยี PCR-Free สามารถหลีกเลี่ยงปัญหาที่กล่าวถึงข้างต้นที่เกิดจากการขยาย PCR ในการสร้างห้องสมุดแบบเดิมได้เป็นอย่างดี ในกระบวนการสร้างห้องสมุดและจัดลำดับ หากมีกระบวนการขยาย PCR ก็จะมีผลกระทบต่อความสม่ำเสมอของการครอบคลุมจีโนมมากขึ้น สำหรับเทมเพลตดีเอ็นเอ บางส่วนมีโครงสร้างรองที่ซับซ้อน และบางส่วนมีความแตกต่างกันมากในเสถียรภาพทางความร้อน ปัจจัยเหล่านี้จะส่งผลต่อประสิทธิภาพของการขยาย PCR ดังนั้น ในกระบวนการขยาย PCR จึงไม่สามารถรับประกันได้ว่าชิ้นส่วนจีโนมทั้งหมดจะได้รับประสิทธิภาพการขยายเท่ากัน แต่มีความลำเอียงในการขยายที่ชัดเจน เช่น การครอบคลุมต่ำในบริเวณ GC สูงหรือบริเวณ GC ต่ำของจีโนม อย่างไรก็ตาม ไม่มี PCR ในกระบวนการจัดลำดับแบบ PCR-Free ทั้งหมด เมื่อเปรียบเทียบกับการสร้างไลบรารี PCR การครอบคลุมบริเวณ GC สูงและบริเวณซ้ำ AT/TA ของจีโนมจะได้รับการปรับปรุง ข้อดีเฉพาะมีดังต่อไปนี้
2.1 กระบวนการเตรียมห้องสมุดที่คล่องตัว ประหยัดเวลา
การเตรียมห้องสมุดที่ปราศจาก PCR ช่วยขจัดความจำเป็นในการขยาย PCR และขั้นตอนอื่นๆ ทำให้กระบวนการเตรียมห้องสมุดมีประสิทธิภาพขึ้นและประหยัดเวลา
2.2 ลดต้นทุนการจัดเตรียมห้องสมุด
ในกระบวนการเตรียมห้องสมุดแบบเดิม เอนไซม์คุณภาพสูงราคาแพงจะถูกใช้สำหรับการขยาย PCR เพื่อให้แน่ใจว่าลำดับมีความเที่ยงตรง ในขณะเดียวกัน PCR-Free จะขจัดขั้นตอนการขยาย PCR ซึ่งช่วยลดต้นทุนการเตรียมห้องสมุด
2.3 ลดความลำเอียงในการขยายสัญญาณ
เอนไซม์ขยาย DNA มีอคติในการขยายบางอย่าง โดยเฉพาะอย่างยิ่งสำหรับเทมเพลตที่มีเนื้อหา GC หรือโครงสร้างรองที่แตกต่างกันอย่างมีนัยสำคัญ ดังนั้น PCR-Free จึงสามารถหลีกเลี่ยงอคติในการขยายที่เกิดจากโพลิเมอเรสได้อย่างมีประสิทธิภาพ
2.4 ไม่มีการจำลอง PCR
PCR-Free สามารถลดการอ่านซ้ำและเพิ่มอัตราการทำ Unique Mapping และการใช้ข้อมูลที่มีประสิทธิภาพ
2.5 ลดข้อผิดพลาด
การขยาย PCR มีแนวโน้มที่จะทำให้เกิดข้อผิดพลาดในการจำลองของการแทรกและการลบ ส่งผลให้มีอัตราข้อผิดพลาดในการจัดลำดับ Indel ที่สูงขึ้น ในขณะที่ PCR-Free สามารถหลีกเลี่ยงการสร้างและการสะสมของข้อผิดพลาดดังกล่าวได้อย่างมีประสิทธิภาพ
3. การแสดงข้อมูล PCR-Free
ชุดเตรียมไลบรารี DNA ฟรี Hieff NGS™ Ultima Pro PCR V2 (Cat# 12196ES) มีอัตราการแปลงไลบรารีที่ดีกว่าผลิตภัณฑ์คู่แข่ง
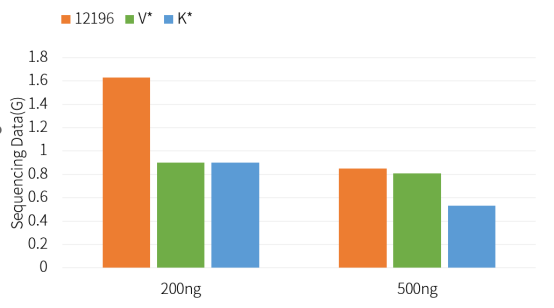
รูปที่ 2 ปริมาณข้อมูลการจัดลำดับนอกเครื่องจักรสำหรับการสร้างห้องสมุด PCR-Free
4. คำถามที่พบบ่อย
ถาม: ประเภทตัวอย่างใดบ้างที่เหมาะกับห้องสมุดที่ปราศจาก PCR?
A: ตัวอย่างบางส่วนที่มีโครงสร้างรองที่ซับซ้อน GC สูง และความต้องการ PCR สามารถเลือกสร้างไลบรารีที่ปราศจาก PCR ได้
ถาม: ขนาดชิ้นส่วนทั่วไปของไลบรารีที่ปราศจาก PCR คือเท่าใด
A: ไลบรารีที่ปราศจาก PCR นั้นไม่มีข้อกำหนดพิเศษเกี่ยวกับขนาดของชิ้นส่วน ไลบรารีผลิตภัณฑ์ PCR นั้นถูกสร้างขึ้นตามขนาดของผลิตภัณฑ์ PCR สำหรับไลบรารีจีโนมนั้น แนะนำให้ใช้ขนาดประมาณ 350bp เพื่อให้คุณภาพของข้อมูลไลบรารีนั้นค่อนข้างดี
5. ข้อมูลผลิตภัณฑ์
ผลิตภัณฑ์ที่
ตารางที่ 1 ข้อมูลสินค้า
| ผลิตภัณฑ์ ชื่อ | หมายเลขแคท | ขนาด |
| ชุดเตรียมไลบรารี DNA ฟรี Hieff NGS™ Ultima Pro PCR V2 | 12196ES24/96 | 24/96ที |
6. เรื่องการอ่านหนังสือ
โซลูชันสำหรับการเตรียมไลบรารี NGS จากตัวอย่าง DNA
คุณมีความรู้เกี่ยวกับเทคโนโลยีที่เกี่ยวข้องกับ NGS มากเพียงใด?
