คำอธิบาย
ลูกปัดคัดเลือก DNA ของ Hieff NGS™ ได้รับการเตรียมขึ้นโดยยึดตามหลักการ SPRI (Solid Phase Reverse Immobilization) และสามารถใช้สำหรับการทำให้บริสุทธิ์ DNA และการเลือกขนาดในระหว่างการเตรียมไลบรารีการจัดลำดับรุ่นถัดไป (NGS) ลูกปัดคัดเลือก DNA ของ Hieff NGS™ สามารถใช้งานร่วมกับชุดเตรียมไลบรารี DNA และ RNA ต่างๆ ได้ และเป็นทางเลือกที่ดี ลูกปัด AMPure-
ส่วนประกอบ
| ส่วนประกอบ หมายเลข | ชื่อ | 12601ES08 | 12601ES56 | 12601ES75 |
| 12601 | ลูกปัดคัดแยก DNA Hieff NGS™ | 5 มล. | 60 มล. | 450 มล. |
ข้อมูลจำเพาะ
| สายผลิตภัณฑ์ | DNA สะอาดและคัดเลือกลูกปัด |
| วัสดุเริ่มต้น | ดีเอ็นเอ |
| ความเข้ากันได้ | ดีเอ็นเอ |
| เทคโนโลยีการแยกตัว | ลูกปัดแม่เหล็ก |
| ประเภทผลิตภัณฑ์ขั้นสุดท้าย | ดีเอ็นเอ |
| สำหรับใช้กับ (แอปพลิเคชั่น) | การเลือกขนาด DNA |
การขนส่งและการเก็บรักษา
ลูกปัดจะถูกจัดส่งพร้อมกับถุงน้ำแข็งและสามารถเก็บไว้ที่อุณหภูมิ 2°C-8°C ได้นานหนึ่งปี
คำแนะนำ
- 1. การเตรียมพร้อม
ให้ปรับสมดุลลูกปัดคัดแยกที่อุณหภูมิห้องเป็นเวลาอย่างน้อย 30 นาที ก่อนใช้งาน
- 2. การเลือกขนาด DNA
ขั้นตอนการดำเนินการเลือกขนาดแสดงอยู่ในรูปที่ 1 และโปรโตคอลเป็นดังต่อไปนี้
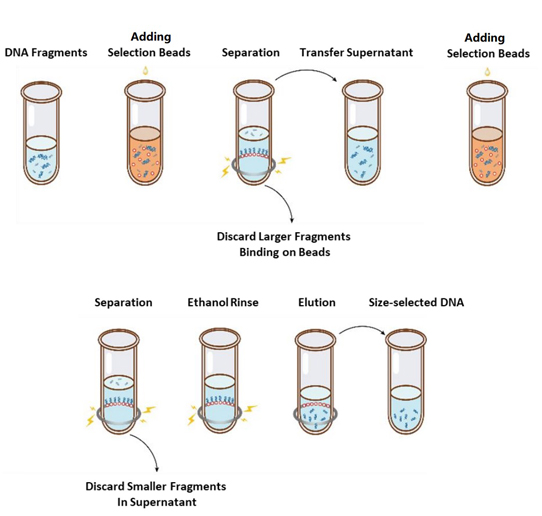
รูปที่ 1 แผนผังขั้นตอนการเลือกขนาด DNA
2.1 ผสมลูกปัดให้เข้ากันด้วยการเขย่าหรือปิเปตขึ้นลงทุกครั้งก่อนใช้งาน
2.2 เติมลูกปัดคัดเลือกรอบแรกลงในตัวอย่าง (ดูตารางที่ 1) ผสมให้เข้ากันโดยใช้เครื่องวอร์เท็กซ์หรือปิเปตขึ้นลงอย่างน้อย 10 ครั้ง
2.3 ฟักที่อุณหภูมิห้องเป็นเวลา 5 นาที
2.4 หมุนหลอดลงสักครู่แล้ววางบนฐานแม่เหล็ก เมื่อสารละลายใส (ประมาณ 5 นาที) ให้ย้ายส่วนที่เป็นของเหลวใสกว่าลงในหลอด PCR หลอดใหม่
2.5 เติมลูกปัดคัดรอบที่ 2 ลงในตัวอย่างจากขั้นตอน 2.4 ตามตารางที่ 1 ผสมให้เข้ากันโดยใช้การเขย่าหรือปิเปตขึ้นและลงอย่างน้อย 10 ครั้ง
2.6 ฟักที่อุณหภูมิห้องเป็นเวลา 5 นาที
2.7 หมุนหลอดลงสักครู่แล้ววางบนฐานแม่เหล็ก เมื่อสารละลายใส (ประมาณ 5 นาที) ให้ดูดของเหลวส่วนบนออกแล้วทิ้งไป
2.8 วางหลอดทดลองบนฐานแม่เหล็ก แล้วเติมเอธานอล 80% ที่เตรียมใหม่ 200 μL ลงไปโดยไม่รบกวนลูกปัด ฟักที่อุณหภูมิห้องเป็นเวลา 30 วินาที ดูดเอธานอลออกแล้วทิ้งไป
2.9 ทำซ้ำขั้นตอน 2.8 หนึ่งครั้งเพื่อซักรวมสองครั้ง
2.10 กำจัดเอธานอลที่เหลือด้วยปลายปิเปตขนาด 10 µL วางหลอดทดลองบนขาตั้งแม่เหล็ก ปล่อยให้ลูกปัดที่เลือกแห้งโดยเปิดฝาทิ้งไว้จนกระทั่งมีรอยแตกร้าวปรากฏขึ้น (ประมาณ 5 นาที)
หมายเหตุ: อย่าทำให้ลูกปัดที่เลือกแห้งเกินไป เพราะอาจทำให้เป้าหมาย DNA ที่ได้รับการฟื้นฟูลดลง
2.11 ถอดหลอดออกจากฐานแม่เหล็ก เติม ddH2O (≥20 µL) ในปริมาณที่เหมาะสม และผสมให้เข้ากันโดยใช้เครื่องวอร์เท็กซ์หรือปิเปตขึ้นลงอย่างน้อย 10 ครั้ง
2.12 ฟักที่อุณหภูมิห้องเป็นเวลา 5 นาที
หมุนหลอดลงสักครู่แล้ววางบนฐานแม่เหล็ก เมื่อสารละลายใส (ประมาณ 5 นาที) ให้ถ่ายของเหลวส่วนบน 20 μL ลงในหลอดใหม่
- 3.เงื่อนไขที่แนะนำสำหรับการเลือกขนาด DNA
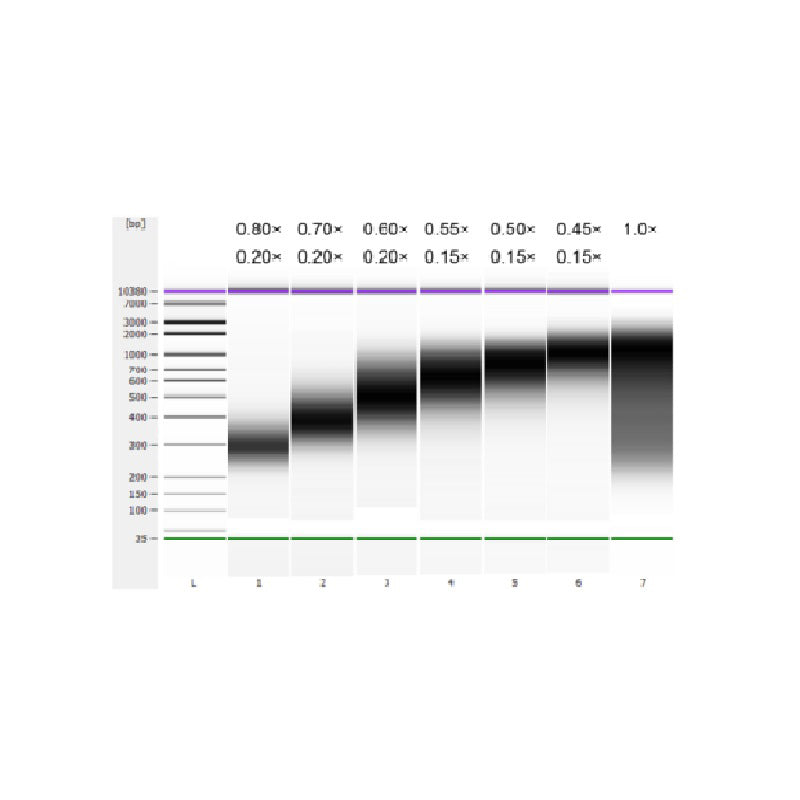
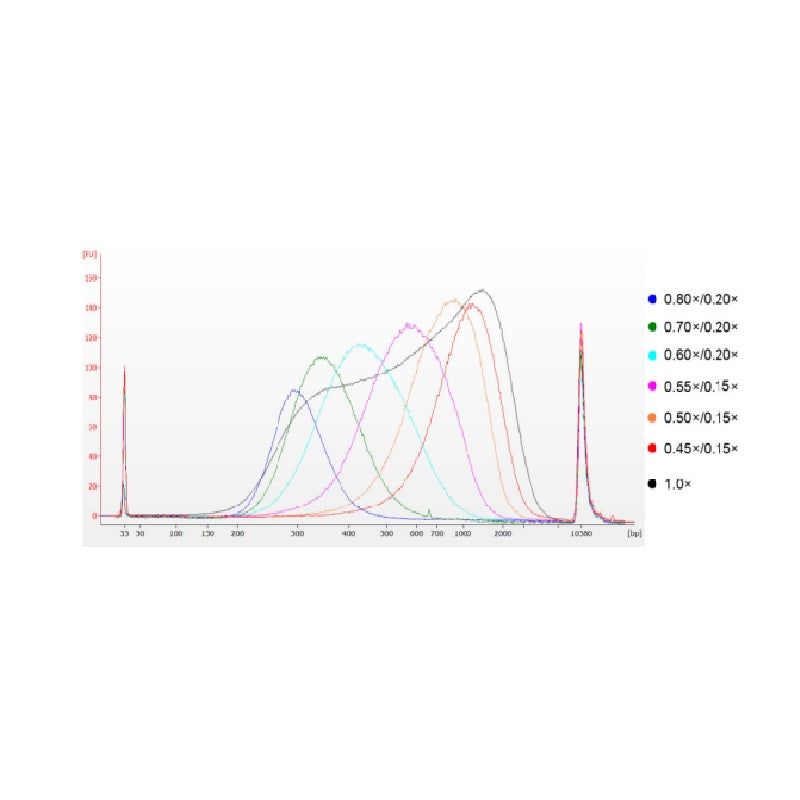
ดีเอ็นเอจากต่อมไทมัสของลูกวัวถูกแบ่งส่วนโดยใช้คลื่นเสียงความถี่สูงเพื่อเตรียมชิ้นส่วนขนาด 100-1,000 คู่เบส และทำการคัดเลือกขนาดสองรอบตามตารางที่ 1 จากนั้นวิเคราะห์ผลลัพธ์โดยใช้เครื่อง Agilent 2100 Bioanalyzer (รูปที่ 2)
ตารางที่ 1 เงื่อนไขที่แนะนำสำหรับการเลือกขนาด DNA
| ความยาวของชิ้นส่วน DNA | 250-350 บ. | 320-420 บ. | 450-550 บ. | 550-700 บ. | 700-900 บ. | 800-1,000 คู่ |
| อัตราส่วนของลูกปัด: ดีเอ็นเอรอบที่ 1 | 0.80× | 0.70× | 0.60× | 0.55× | 0.50× | 0.45× |
| อัตราส่วนของลูกปัด: ดีเอ็นเอสำหรับครั้งที่ 2 กลม | 0.20× | 0.20× | 0.20× | 0.15× | 0.15× | 0.15× |
หมายเหตุ: "×" ในตารางระบุปริมาตรของตัวอย่าง DNA ตัวอย่างเช่น หากความยาวของส่วนที่แทรกในคลังข้อมูลคือ 250 bp และปริมาตรของตัวอย่าง DNA คือ 100 μL ปริมาตรของลูกปัดแม่เหล็กที่ใช้ในการคัดแยกรอบแรกคือ 0.80×100 μL=80 μL ปริมาตรของลูกปัดแม่เหล็กที่ใช้ในการคัดแยกรอบที่สองคือ 0.20×100 μL=20 μL
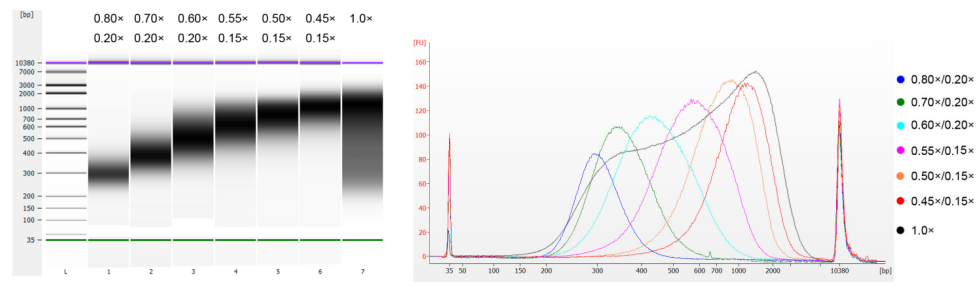
รูปที่ 2 อิเล็กโทรเฟอโรแกรมของชิป DNA ความไวสูง Agilent 2100
หมายเหตุ:
1. เพื่อความปลอดภัยและสุขภาพของคุณ โปรดสวมเสื้อคลุมแล็บและถุงมือแบบใช้แล้วทิ้งขณะผ่าตัด

อ้างจาก "การรวมลำดับเฉพาะโดยตระกูล 1 casposase จาก Candidatus Nitrosopumilus koreensis AR1. กรดนิวคลีอิก Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725"

อ้างจาก "การติดเชื้อ Wolbachia เมื่อไม่นานนี้ทำให้ชุมชนจุลินทรีย์ในประชากร Laodelphax striatellus ป่าเปลี่ยนแปลงไป Microbiome 2020;8(1):104 เผยแพร่เมื่อวันที่ 2 กรกฎาคม 2020 doi:10.1186/s40168-020-00878-x"
[1] Wang X, Yuan Q, Zhang W และคณะ การรวมลำดับเฉพาะโดยแฟมิลี 1 แคสโปเซสจาก Candidatus Nitrosopumilus koreensis AR1. กรดนิวคลีอิก Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725(IF:16.971)
[2] Duan XZ, Sun JT, Wang LT และคณะ การติดเชื้อ Wolbachia เมื่อเร็ว ๆ นี้ทำให้ชุมชนจุลินทรีย์ในประชากร Laodelphax striatellus เปลี่ยนแปลงไป ไมโครไบโอม 2020;8(1):104 เผยแพร่เมื่อวันที่ 2 กรกฎาคม 2020 doi:10.1186/s40168-020-00878-x(IF:11.607)
[3] เพลง B, Almatrafi E, Sang F, และคณะการจัดการตะกอนที่ผ่านการบำบัดด้วยเฟนตันด้วยไบโอชาร์และปุ๋ยหมักมูลแกะ: ผลกระทบต่อลักษณะวิวัฒนาการของชุมชนแบคทีเรีย J Environ Manage 2022;316:115218 doi:10.1016/j.jenvman.2022.115218(IF:6.789)
[4] Huang C, Mei Q, Lou L และคณะ Ulcerative Colitis in Response to Fecal Microbiota Transplantation via Modulation of Gut Microbiota and Th17/Treg Cell Balance. Cells. 2022;11(11):1851. เผยแพร่เมื่อวันที่ 5 มิถุนายน 2022 doi:10.3390/cells11111851(IF:6.600)
[5] Ghosh S, Yang X, Wang L, Zhang C, Zhao L. การให้อาหารพรีไบโอติกแบบแอคทีฟเฟสทำให้จุลินทรีย์ในลำไส้เปลี่ยนแปลง กระตุ้นให้เกิดการบรรเทาภาวะไขมันเกาะตับและคอเลสเตอรอลในซีรั่มที่ไม่ขึ้นกับน้ำหนักในหนูที่ได้รับอาหารไขมันสูง Comput Struct Biotechnol J. 2020;19:448-458. เผยแพร่เมื่อวันที่ 24 ธันวาคม 2020 doi:10.1016/j.csbj.2020.12.011(IF:6.018)
[6] Gao X, Yu B, Yu J และคณะ Developmental Profiling of Dietary Carbohydrate Digestion in Piglets. Front Microbiol. 2022;13:896660. เผยแพร่เมื่อวันที่ 29 เมษายน 2022 doi:10.3389/fmicb.2022.896660(IF:5.640)
[7] Li P, Zhang Y, Yan F, Zhou X. ลักษณะของแบคทีเรียโฟจ vB_Kox_ZX8 ที่แยกได้จาก Klebsiella oxytoca ทางคลินิกและผลการรักษาต่อภาวะแบคทีเรียในกระแสเลือดของหนู Front Microbiol. 2021;12:763136. เผยแพร่เมื่อวันที่ 3 ธันวาคม 2021 doi:10.3389/fmicb.2021.763136(IF:5.640)
[8] Lin Z, Luo P, Huang D, Wu Y, Li F, Liu H. กลยุทธ์ที่ใช้มัลติโอมิกส์สำหรับการวิเคราะห์ความเป็นพิษของอะคริลาไมด์ในแบบจำลอง Saccharomyces cerevisiae Chem Biol Interact 2021;349:109682 doi:10.1016/j.cbi.2021.109682(IF:5.194)
[9] ซัน X, Lv W, วัง Y และคณะ ยีน Mrgprb2 มีบทบาทในปฏิกิริยาแอนาฟิแลคตอยด์ที่เกิดจากการฉีด Houttuynia cordata เจ เอทโนฟาร์มาคอล. 2022;289:115053. ดอย:10.1016/j.jep.2022.115053(IF:4.360)
[10] Ma H, Lai B, Zan C, Di X, Zhu X, Wang K. GLO1 มีส่วนสนับสนุนต่อการดื้อยาของ Escherichia coli ผ่านการเหนี่ยวนำให้เกิด β-Lactamases สเปกตรัมขยายประเภท PER Infect Drug Resist. 2022;15:1573-1586. เผยแพร่เมื่อวันที่ 5 เมษายน 2022 doi:10.2147/IDR.S358578(IF:4.003)
[11] Zhong Y, Zhao W, Tang Z และคณะ การวิเคราะห์ทรานสคริปโตมิกส์เชิงเปรียบเทียบของระยะการพัฒนาที่แตกต่างกันของรังไข่ในกุ้งน้ำจืดหนองแดง Procambarus clarkii BMC Genomics 2021;22(1):199 เผยแพร่เมื่อวันที่ 21 มีนาคม 2021 doi:10.1186/s12864-021-07537-x(IF:3.969)
[12] Lian C, Yang H, Lan J และคณะ การวิเคราะห์เปรียบเทียบจีโนมคลอโรพลาสต์เผยให้เห็นความสัมพันธ์เชิงวิวัฒนาการและความหลากหลายภายในสายพันธุ์ในพืชสมุนไพร Isodon rubescens PLoS One 2022;17(4):e0266546 เผยแพร่เมื่อวันที่ 6 เมษายน 2022 doi:10.1371/journal.pone.0266546(IF:3.240)
(13) Diao G, Huang J, Zheng X และคณะ พรอสตาแกลนดิน E2 ทำหน้าที่สองอย่างในการควบคุมการเคลื่อนที่ของเซลล์เดนไดรต์ Int J Mol Med. 2021;47(1):207-218. doi:10.3892/ijmm.2020.4801(IF:3.098)
[14] Bing XL, Zhao DS, Peng CW, Huang HJ, Hong XY. ความคล้ายคลึงและการเปลี่ยนแปลงเชิงพื้นที่ของชุมชนแบคทีเรียและเชื้อราในประชากรเพลี้ยกระโดดในนาข้าว (Hemiptera: Delphacidae) Insect Sci. 2020;27(5):947-963. doi:10.1111/1744-7917.12782(IF:2.791)
[15] Li X, Zhou S, Zhang J, Zhou Z, Xiong Q. Directional Changes in the Intestinal Bacterial Community in Black Soldier Fly (Hermetia illucens) Larvae. Animals (Basel). 2021;11(12):3475. เผยแพร่เมื่อวันที่ 6 ธันวาคม 2021 doi:10.3390/ani11123475(IF:2.752)
[16] Yang J, Peng Y, Kong W. การระบุและลำดับจีโนมที่สมบูรณ์ของไวรัสมัลเบอร์รี่คริปติก 1 Arch Virol. 2022;167(2):687-690. doi:10.1007/s00705-021-05350-1(IF:2.574)
[17] Chang Y, Xia X, Sui L และคณะ การตั้งอาณานิคมของเชื้อราก่อโรคในแมลงแบบเอ็นโดไฟต์ช่วยเพิ่มความต้านทานโรคพืชโดยการเปลี่ยนแปลงชุมชนแบคทีเรียในเอ็นโดไฟต์ J Basic Microbiol. 2021;61(12):1098-1112. doi:101002/งานม.202100494(IF:2.281)
[18] Ding CY, Ma YM, Li B และคณะ การระบุและวิเคราะห์การทำงานของยีนที่แสดงออกแตกต่างกันใน Myzus persicae (Hemiptera: Aphididae) ในการตอบสนองต่อ Trans-anethole J Insect Sci. 2022;22(1):3. doi:10.1093/jisesa/ieab094(IF:1.857)
การชำระเงินและความปลอดภัย
ข้อมูลการชำระเงินของคุณได้รับการดำเนินการอย่างปลอดภัย เราไม่เก็บรายละเอียดบัตรเครดิตและไม่สามารถเข้าถึงข้อมูลบัตรเครดิตของคุณได้
การสอบถาม
คุณอาจชอบ
คำถามที่พบบ่อย
ผลิตภัณฑ์นี้มีวัตถุประสงค์เพื่อการวิจัยเท่านั้น และไม่ได้มีวัตถุประสงค์เพื่อใช้ในการรักษาหรือวินิจฉัยโรคในมนุษย์หรือสัตว์ ผลิตภัณฑ์และเนื้อหาได้รับการคุ้มครองโดยสิทธิบัตร เครื่องหมายการค้า และลิขสิทธิ์ที่เป็นของ
แอปพลิเคชั่นบางตัวอาจต้องใช้สิทธิ์ในทรัพย์สินทางปัญญาของบุคคลที่สามเพิ่มเติม