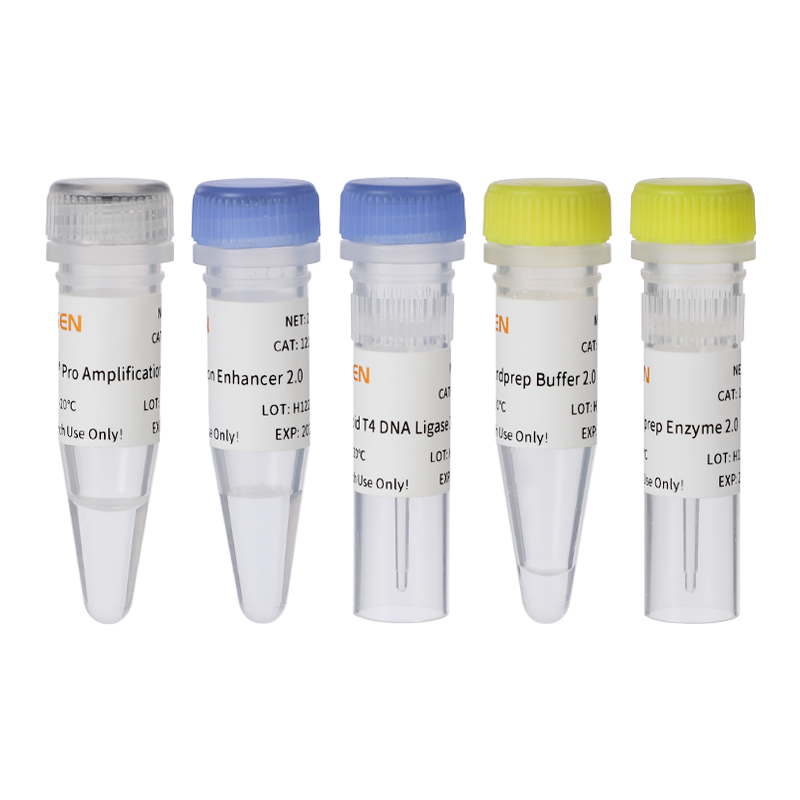
คำอธิบาย
ไฮฟ์ เอ็นจีเอสทีเอ็ม DNA Library Prep Kit คือชุดสร้างห้องสมุดรุ่นใหม่ที่ได้รับการพัฒนาและออกแบบมาโดยเฉพาะสำหรับ อิลลูมิน่า® และเอ็มจีไอ® แพลตฟอร์มการเรียงลำดับผลิตภัณฑ์นี้แสดงให้เห็นถึงประสิทธิภาพที่สูงขึ้นในการซ่อมแซมปลายสาย การต่อ dA และการผูกอะแดปเตอร์เมื่อเทียบกับรุ่นก่อนหน้า โดยอาศัยชุดสร้างห้องสมุดรุ่นก่อนหน้า เอนไซม์ที่มีความเที่ยงตรงสูงช่วยปรับปรุงความสม่ำเสมอและความเที่ยงตรงของการขยายพันธุ์ได้อย่างมาก ชุดนี้สามารถใช้งานร่วมกับตัวอย่าง DNA ส่วนใหญ่ได้ รวมถึง DNA จีโนมมาตรฐานจากสัตว์/พืช/จุลินทรีย์ ตัวอย่าง FFPE, cfDNA และ ChIP DNA
ข้อมูลจำเพาะ
| เลขที่แมว | 12927ES08 / 12927ES24 / 12927ES96 |
| ขนาด | 8 รเอ็กซ์เอ็น / 24 รเอ็กซ์เอ็น / 96 รเอ็กซ์เอ็น |
ส่วนประกอบ
| ส่วนประกอบ หมายเลข | ชื่อ | 12927ES08 | 12927อีเอส24 | 12927ES96 |
| 12927-ก | 56 ไมโครลิตร | 168 ไมโครลิตร | 672 ไมโครลิตร | |
| 12927-บี | เอ็นด์เพรพ เอ็นไซม์ | 24 ไมโครลิตร | 72 ไมโครลิตร | 288 ไมโครลิตร |
| 12927-ซี | สารเพิ่มประสิทธิภาพการรัด | 240 ไมโครลิตร | 720 ไมโครลิตร | 3×960 ไมโครลิตร |
| 12927-ดี | รวดเร็ว ที4 ดีเอ็นเอ ไลเกส | 80 ไมโครลิตร | 240 ไมโครลิตร | 2×480 ไมโครลิตร |
| 12927-อี | คานาเซ่ทีเอ็ม มิกซ์ขยายเสียงระดับมืออาชีพ | 200 ไมโครลิตร | 600 ไมโครลิตร | 3×800 ไมโครลิตร |
พื้นที่จัดเก็บ
ควรเก็บผลิตภัณฑ์นี้ไว้ที่ -25~-15℃ เป็นเวลา 1 ปี.
หมายเหตุ
1. เกี่ยวกับการดำเนินงาน
1. โปรดปฏิบัติงานโดยสวมเสื้อคลุมแล็บและถุงมือแบบใช้แล้วทิ้ง-เพื่อความปลอดภัยของคุณ
2. ละลายส่วนประกอบที่อุณหภูมิห้อง หลังการละลายน้ำแข็งผสมให้เข้ากันด้วยการปั่น หมุนหลอดสั้นๆ แล้ววางลงบนน้ำแข็งเพื่อใช้ในภายหลัง
3. เมื่อเตรียมสารละลายปฏิกิริยาในแต่ละขั้นตอน แนะนำให้ใช้ปิเปตเพื่อผสมให้เข้ากันหรือเขย่าเบาๆ การเขย่าแรงๆ อาจทำให้ปริมาณสารในห้องสมุดลดลง
4. ขอแนะนำให้ใช้ปลายปิเปตแบบกรองเพื่อหลีกเลี่ยงการปนเปื้อนข้าม ควรเปลี่ยนปลายปิเปตเมื่อประมวลผลตัวอย่างที่แตกต่างกัน
5. การดำเนินการที่ไม่เหมาะสมอาจทำให้เกิดการปนเปื้อนของละอองลอย ซึ่งส่งผลต่อความแม่นยำของผลลัพธ์ แนะนำให้แยกบริเวณการผสมปฏิกิริยา PCR และบริเวณการทดสอบการทำให้บริสุทธิ์ผลิตภัณฑ์ PCR ออกจากกันทางกายภาพ ติดตั้งอุปกรณ์ เช่น ปิเปตเฉพาะทางสำหรับการสร้างห้องสมุดทำความสะอาดตามปกติสำหรับแต่ละพื้นที่โดยเช็ดพื้นผิวด้วยโซเดียมไฮโปคลอไรต์ 0.5% หรือสารฟอกขาว 10%
6. ผลิตภัณฑ์นี้ใช้เพื่อการวิจัยเท่านั้น
2. การแตกตัวของดีเอ็นเอ
1. ชุดนี้สามารถใช้งานได้กับ DNA ที่แตกกระจายโดยกลไก หรือ DNA ที่แตกกระจายโดยเอนไซม์
2. ชุดทดสอบนี้รองรับ DNA อินพุต 100 pg - 1000 ng ขอแนะนำให้ใช้ DNA อินพุตคุณภาพสูงกับ A260/A280 = 1.8-2.0 ตารางที่ 1 แสดงปริมาณ DNA อินพุตที่แนะนำ
ตารางที่ 1 ปริมาณ DNA อินพุตที่แนะนำ
| แอปพลิเคชัน | ประเภทตัวอย่าง | อินพุตดีเอ็นเอ |
| ดับเบิ้ลยูจีเอส | จีโนมที่ซับซ้อน | 50 นาโนกรัม1000 ง |
| การเรียงลำดับการจับภาพแบบกำหนดเป้าหมาย | จีโนมที่ซับซ้อน | 10 นาโนกรัม1000 ง |
| WGS, การจัดลำดับเป้าหมาย | ดีเอ็นเอ FFPE | 50 นาโนกรัม1000 ง |
| การจัดลำดับเป้าหมาย | cfDNA/ctDNA | ≥500 หน้า |
| ดับเบิ้ลยูจีเอส | จีโนมของจุลินทรีย์ | ≥1 นาโนกรัม |
| WGS (ปราศจาก PCR) | DNA อินพุตคุณภาพสูง | ≥50 นาโนกรัม |
บันทึก:เมื่อ DNA อินพุตมีคุณภาพต่ำหรือต้องเลือกขนาด DNA ควรเพิ่มจำนวน DNA อินพุตตามนั้น
3. “DNA อินพุต” หมายถึงตัวอย่าง DNA ที่พร้อมสำหรับการซ่อมแซมส่วนปลาย/การหาง dA โดยเฉพาะ
4. แนะนำให้ทำขั้นตอนการทำให้บริสุทธิ์/เลือกขนาดลูกปัดหลังจากการแตกตัวหากตัวอย่าง DNA ที่ป้อนมีเกลือที่มีความเข้มข้นสูง เช่น สารคีเลตโลหะ เกลืออาจส่งผลกระทบต่อประสิทธิภาพของปฏิกิริยาต่อไปนี้ รวมถึงการซ่อมแซมปลายและการแยกตัวของ dAโปรดชะตัวอย่าง DNA ในบัฟเฟอร์ TE แทนน้ำบริสุทธิ์ที่ผ่านการฆ่าเชื้อแล้วสำหรับการแตกตัวหากใช้วิธีการแตกตัวทางกล หากใช้วิธีการแตกตัวด้วยเอนไซม์โดยไม่ทำความสะอาดลูกปัดหรือเลือกขนาดก่อนดำเนินการเตรียมห้องสมุด โปรดให้แน่ใจว่าบัฟเฟอร์หยุดที่ใช้ไม่มีสารคีเลตโลหะเกินปริมาณ มิฉะนั้น โปรดทำความสะอาดหรือเลือกขนาดตัวอย่างที่แตกตัวแล้วชะในบัฟเฟอร์ TE หรือน้ำบริสุทธิ์ที่ผ่านการฆ่าเชื้อแล้ว (≤50 μL) ก่อนดำเนินการเตรียมห้องสมุด
3. การเชื่อมต่ออะแดปเตอร์
1. ชุด Illumina หรือ MGI Long Adapter (Barcoded Adapter) และชุด Short Adapter มีให้เลือกใช้ตามความต้องการในการทดลองของพวกเขา
2. ขอแนะนำให้เลือกใช้อะแดปเตอร์เชิงพาณิชย์ที่มีคุณภาพสูง หากเลือกอะแดปเตอร์ที่ผลิตเอง โปรดฝากบริษัทที่มีประสบการณ์ในการสังเคราะห์ไพรเมอร์ NGS และระบุถึงความจำเป็นในการควบคุมการปนเปื้อนอย่างเข้มงวด นอกจากนี้ ขอแนะนำให้เตรียมสารละลายแอนนีลลิ่ง DNA ในม้านั่งที่สะอาด และใช้อะแดปเตอร์เพียงชนิดเดียวในแต่ละครั้งเพื่อป้องกันการปนเปื้อนข้ามกัน
3. โปรดละลายอะแดปเตอร์บนน้ำแข็งหรือที่อุณหภูมิ 4°C เมื่อใช้งานที่อุณหภูมิห้อง อุณหภูมิในห้องปฏิบัติการไม่ควรเกิน 25°C เพื่อป้องกันไม่ให้อะแดปเตอร์เสียสภาพ
4. คุณภาพและความเข้มข้นของอะแดปเตอร์จะส่งผลโดยตรงต่อประสิทธิภาพการเชื่อมต่อและผลผลิตของไลบรารี ความเข้มข้นของอะแดปเตอร์ที่สูงเกินไปจะเอื้อต่อการก่อตัวของไดเมอร์ของอะแดปเตอร์ ในขณะที่อะแดปเตอร์ที่น้อยเกินไปจะลดอัตราการเชื่อมต่อและผลผลิตของไลบรารี การเจือจางที่สอดคล้องกับบัฟเฟอร์ TE ตามปริมาณ DNA อินพุตเมื่อใช้อะแดปเตอร์ ตารางที่ 2 -5 แสดงรายการวิธีการเจือจางอะแดปเตอร์ที่แนะนำสำหรับปริมาณ DNA อินพุตที่แตกต่างกันโดยใช้ชุดนี้
ตารางที่ 2 Illumina™ ที่แนะนำ จำนวนอะแดปเตอร์สำหรับอินพุต DNA ที่แตกต่างกัน
| อินพุตดีเอ็นเอ | การเจือจางอะแดปเตอร์ (ปริมาตรของอะแดปเตอร์ : ปริมาตรรวม) | ความเข้มข้น |
| 0.1นาโนกรัม ~ 1นาโนกรัมจี | 150 เท่า (1 : 150) | 0.1 ไมโครโมลาร์ |
| 1 ง ~ 10 นาโนกรัม | 75 เท่า (1 : 75) | 0.2 ไมโครโมลาร์ |
| 10 ง ~ 25 นาโนกรัม | 15 เท่า (1 : 15) | 1 ไมโครโมลาร์ |
| 25 นาโนกรัม ~ 100 นาโนกรัม | 7.5 เท่า (1 : 7.5) | 2 ไมโครโมลาร์ |
| 100 นาโนกรัม ~ 1,000 นาโนกรัม | 3 พับ (1 : 3) | 5 ไมโครโมลาร์ |
ตารางที่ 3 MGI™ ที่แนะนำ จำนวนอะแดปเตอร์สำหรับอินพุต DNA ที่แตกต่างกัน
| อินพุตดีเอ็นเอ | การเจือจางอะแดปเตอร์ (ปริมาตรของอะแดปเตอร์ : ปริมาตรรวม) | ความเข้มข้น |
| 0.1นาโนกรัม ~ 1นาโนกรัมจี | 100-พับ (1 : 100) | 0.1 ไมโครโมลาร์ |
| 1 ง ~ 10 นาโนกรัม | 50-พับ (1 : 50- | 0.2 ไมโครโมลาร์ |
| 10 ง ~ 25 นาโนกรัม | 10-พับ (1 : 10- | 1 ไมโครโมลาร์ |
| 25 นาโนกรัม ~ 100 นาโนกรัม | 5 พับ (1 : 5) | 2 ไมโครโมลาร์ |
| 100 นาโนกรัม ~ 1,000 นาโนกรัม | 2-พับ (1 : 2- | 5 ไมโครโมลาร์ |
ตารางที่ 4 Illumina™ ที่แนะนำ จำนวนอะแดปเตอร์ UMI สำหรับ DNA อินพุตที่แตกต่างกัน
| อินพุตดีเอ็นเอ | การเจือจางอะแดปเตอร์ (ปริมาตรของอะแดปเตอร์ : ปริมาตรรวม) | ความเข้มข้น |
| 0.1นาโนกรัม ~ 1นาโนกรัมจี | 150 เท่า (1 : 150) | 0.1 ไมโครโมลาร์ |
| 1 ง ~ 10 นาโนกรัม | 75 เท่า (1 : 75) | 0.2 ไมโครโมลาร์ |
| 10 ง ~ 25 นาโนกรัม | 15 เท่า (1 : 15) | 1 ไมโครโมลาร์ |
| 25 นาโนกรัม ~ 100 นาโนกรัม | 7.5 พับ (1 : 7.5) | 2 ไมโครโมลาร์ |
| 100 นาโนกรัม ~ 1,000 นาโนกรัม | 3 พับ (1 : 3) | 5 ไมโครโมลาร์ |
ตารางที่ 5 MGI™ ที่แนะนำ จำนวนอะแดปเตอร์ UMI สำหรับ DNA อินพุตที่แตกต่างกัน
| อินพุตดีเอ็นเอ | การเจือจางอะแดปเตอร์ (ปริมาตรของอะแดปเตอร์ : ปริมาตรรวม) | ความเข้มข้น |
| 5 ง ~ 25 นจี | 50-พับ (1 : 50) | 0.2 ไมโครโมลาร์ |
| 25 นาโนกรัม ~ 100 นาโนกรัม | 10-พับ (1 : 10- | 1 ไมโครโมลาร์ |
| 100 นาโนกรัม ~ 1,000 นาโนกรัม | 4-พับ (1 : 4- | 2.5 ไมโครโมลาร์ |
4. การทำความสะอาด DNA และการเลือกขนาดตามลูกปัด
1. การเลือกขนาด DNA สามารถทำได้ก่อนการซ่อมแซมปลาย/การหาง dA หลังการผูกอะแดปเตอร์ หรือหลังการขยาย
2. ขอแนะนำให้ดำเนินการเลือกขนาดทันทีหลังจากการผูกอะแดปเตอร์หากปริมาณ DNA อินพุตมากกว่า 50 นาโนกรัม- มิฉะนั้นกรุณาเลือกขนาดหลังจากขยาย
3. สารเพิ่มประสิทธิภาพการรัดท่อประกอบด้วย PEG ที่มีความเข้มข้นสูง ซึ่งอาจส่งผลกระทบอย่างมีนัยสำคัญต่อการเลือกขนาดที่แม่นยำ ดังนั้น หากต้องเลือกขนาดทันทีหลังการรัดท่อด้วยอะแดปเตอร์ ขอแนะนำอย่างยิ่งให้เพิ่มขั้นตอนการทำความสะอาดเม็ดบีดก่อนการเลือกขนาด ขั้นตอนการเลือกขนาดสามารถดำเนินการได้โดยตรงหากดำเนินการก่อนการซ่อมแซมปลายท่อ/การต่อ dA หรือหลังจาก การขยายห้องสมุด
4. ควรปรับสมดุลลูกปัดแม่เหล็กที่อุณหภูมิห้องก่อนใช้งาน มิฉะนั้น ผลผลิตจะลดลง และผลการเลือกขนาดจะได้รับผลกระทบ
5. ควรผสมลูกปัดแม่เหล็กให้เข้ากันโดยใช้วิธีวอร์เท็กซ์หรือปิเปตก่อนใช้งาน
6. ห้ามดูดลูกปัดออกเมื่อถ่ายโอนของเหลวส่วนบน แม้ว่าจะมีลูกปัดเพียงเล็กน้อยก็อาจส่งผลต่อปฏิกิริยาต่อไปนี้ได้
7. ควรเตรียมเอธานอล 80% ใหม่ๆ มิฉะนั้น จะส่งผลต่อประสิทธิภาพการกู้คืน
8. เพื่อให้เลือกขนาดได้แม่นยำ ขอแนะนำให้เริ่มด้วยปริมาตรมากกว่า 100 μL หากน้อยกว่านี้ ขอแนะนำให้เพิ่มปริมาตรเป็น 100 μL ด้วยน้ำบริสุทธิ์พิเศษ
9. ควรทำให้ลูกปัดแม่เหล็กแห้งที่อุณหภูมิห้องก่อนทำการชะผลิตภัณฑ์ออก หากผลิตภัณฑ์แห้งไม่เพียงพอ อาจทำให้เอธานอลที่เหลือส่งผลต่อปฏิกิริยาที่เกิดขึ้นในภายหลังได้ง่าย หากแห้งมากเกินไป ลูกปัดแม่เหล็กจะแตกร้าวและลดปริมาณการฟอกให้บริสุทธิ์ โดยปกติแล้ว ควรทำให้ลูกปัดแห้งที่อุณหภูมิห้องเป็นเวลา 3-5 นาทีก็เพียงพอที่จะทำให้ลูกปัดแห้งสนิท
10. หากจำเป็น ตัวอย่าง DNA ที่ผ่านการทำให้บริสุทธิ์หรือเลือกขนาดจะถูกชะออกมา 0.1× สามารถเก็บบัฟเฟอร์ TE ที่อุณหภูมิ 4°C ได้นาน 1-2 สัปดาห์ หรือที่อุณหภูมิ -20°C ได้นาน 1 เดือน
5. การขยายเสียงห้องสมุด
1. การขยายไลบรารีหรือไม่นั้นขึ้นอยู่กับปริมาณอินพุตของ DNA ประเภทของอะแดปเตอร์ แอปพลิเคชันข้อมูลการจัดลำดับ ฯลฯ จำเป็นต้องทำขั้นตอนการขยายหากใช้อะแดปเตอร์บางส่วน เมื่อใช้อะแดปเตอร์แบบเต็มความยาว หากอินพุต DNA < 200 นาโนกรัม ขอแนะนำให้ทำการขยาย มิฉะนั้น ไม่จำเป็นต้องทำการขยาย
2. จำนวนรอบการขยายสัญญาณควรได้รับการควบคุมอย่างเคร่งครัด การขยายสัญญาณที่ไม่เพียงพออาจส่งผลให้ผลผลิตของไลบรารีลดลง การขยายสัญญาณมากเกินไปอาจทำให้เกิดอคติ ข้อผิดพลาด การอ่านซ้ำ และผลิตภัณฑ์ไคเมอริกที่เพิ่มขึ้น ตารางที่ 6 แสดงรายการหมายเลขรอบที่แนะนำโดยตั้งเป้าผลผลิตของห้องสมุดที่ 1 μg
ตารางที่ 6 จำนวนรอบที่แนะนำในการสร้าง ผลผลิตห้องสมุด 1,000 นาโนกรัม
| อินพุตดีเอ็นเอ | จำนวนรอบที่จำเป็นในการสร้างผลผลิตห้องสมุด 1 μg |
| 1,000 นาโนกรัม | 2 - 4 |
| 500 นาโนกรัม | 2 - 4 |
| 250 นาโนกรัม | 4 - 6 |
| 100 นาโนกรัม | 5 - 7 |
| 50 นาโนกรัม | 7 - 9 |
| 10 ง | 9 - 11 |
| 5 ง | 10 - 12 |
| 1 ง | 12 - 15 |
| 100 หน้า | 16 - 18 |
บันทึก-
1.ตาราง 6 แสดงจำนวนพารามิเตอร์ลูปโดยใช้การทดสอบ DNA อินพุตคุณภาพสูงประมาณ 200 bp คุณภาพของ DNA FFPE แตกต่างกันอย่างมาก และเมื่อคุณภาพของ DNA ไม่ดีหรือความยาวของไลบรารียาว จำนวนรอบจะต้องเพิ่มขึ้นอย่างเหมาะสมเพื่อให้ได้ไลบรารีที่เพียงพอ
2.ฉันจำเป็นต้องเลือกขนาด f ในระหว่างกระบวนการสร้างห้องสมุด ขอแนะนำให้เลือกจำนวนรอบที่สูงกว่าสำหรับการขยายห้องสมุด มิฉะนั้น ขอแนะนำให้เลือกจำนวนรอบที่น้อยกว่า
3.ฉันหากใช้อะแดปเตอร์ที่ไม่สมบูรณ์ จะต้องขยายอย่างน้อย 2 วงจรเพื่อสร้างอะแดปเตอร์ที่สมบูรณ์
6. การวิเคราะห์คุณภาพห้องสมุด
1. คุณภาพของห้องสมุดที่สร้างขึ้นโดยทั่วไปจะได้รับการวิเคราะห์โดยการวัดความเข้มข้นและการกระจายขนาด
2. สามารถวัดความเข้มข้นของห้องสมุดได้ด้วยวิธีที่ใช้ฟลูออเรสเซนต์ เช่น Qubit และ PicoGreen หรือ qPCR
3. ไม่แนะนำให้ใช้การวัดปริมาณตามการดูดกลืนแสง เช่น NanoDrop
4. แนะนำให้ใช้วิธี qPCR สำหรับการวัดปริมาณไลบรารี: วิธีที่ใช้ฟลูออเรสเซนต์ เช่น Qubit และ PicoGreen ไม่สามารถแยกความแตกต่างระหว่างโครงสร้าง dsDNA ที่ไม่สมบูรณ์ (ส่วนที่แทรกโดยไม่มีอะแดปเตอร์หรือส่วนที่ปลายด้านใดด้านหนึ่งถูกผูกด้วยอะแดปเตอร์) จากไลบรารีที่สมบูรณ์ได้ วิธี qPCR จะขยายและวัดไลบรารีที่สมบูรณ์โดยที่ปลายทั้งสองข้างถูกผูกด้วยอะแดปเตอร์เท่านั้น (ไลบรารีที่เรียงลำดับได้) ซึ่งจะทำให้การวัดปริมาณโหลดมีความแม่นยำมากขึ้น
5.การกระจายขนาดของห้องสมุดสามารถวิเคราะห์ได้โดยใช้ Agilent Bioanalyzer หรืออุปกรณ์อื่นๆ ที่ใช้หลักการของอิเล็กโทรโฟรีซิสแบบเส้นเลือดฝอยหรือไมโครฟลูอิดิกส์
7. วัสดุอื่น ๆ
1. ลูกปัดแม่เหล็กทำความสะอาด DNA: Hieff NGSทีเอ็ม ลูกปัดคัดแยกดีเอ็นเอ (
2. อะแดปเตอร์: อะแดปเตอร์ครบชุดสำหรับ Illumina-
3. การวิเคราะห์คุณภาพห้องสมุด: Agilent 2100 Bioanalyzer DNA 1000 Chip/High Sensitivity Chip หรือผลิตภัณฑ์เทียบเท่าอื่น ๆ; สารเคมีเชิงปริมาณในห้องสมุด
4. วัสดุอื่นๆ: เอธานอลบริสุทธิ์ น้ำบริสุทธิ์ที่ผ่านการฆ่าเชื้อ ปลายปิเปตที่มีการกักเก็บต่ำ หลอด PCR ขาตั้งแม่เหล็ก เครื่องปั่นความร้อน ฯลฯ
คำแนะนำ
ขั้นตอนที่ 1. การซ่อมแซมส่วนปลาย/การขัดผิว
1. ละลาย สารเคมีที่ระบุในตาราง 7 พลิกกลับเพื่อผสมสารเคมีให้เข้ากันแล้ววางบนน้ำแข็งเพื่อใช้ในภายหลัง
2. ประกอบสารเคมีตามตาราง 7 บนน้ำแข็ง
โต๊ะ 7 ระบบซ่อมแซมปลายท่อ/ ระบบปฏิกิริยา dA-Tailing
| ส่วนประกอบ | ปริมาตร (μL) |
| ดีเอ็นเอที่แตกเป็นชิ้นเล็กชิ้นน้อย | เอ็กซ์ |
| บัฟเฟอร์เอ็นด์เพรพ | 7 |
| เอ็นด์เพรพ เอ็นไซม์ | 3 |
| ดีดีเอช2โอ้ | สูงถึง 60 |
3. ผสมเบาๆ ด้วยการปิเปตหรือเขย่า ปั่นเหวี่ยงสั้นๆ เพื่อให้ได้สารละลายลงไป
4. วางหลอดในเครื่องเทอร์โมไซเคิลและตั้งโปรแกรมตามตารางที่ 8
ตารางที่ 8 การซ่อมแซมส่วนปลาย/การซ่อมบำรุง โปรแกรมตอบสนอง
| อุณหภูมิ | เวลา |
| ปิดฝาให้ร้อน 105 องศาเซลเซียส | บน |
| 30 องศาเซลเซียส | 30 นาที |
| 72 องศาเซลเซียส | 30 นาที |
| 4 องศาเซลเซียส | ถือ |
ขั้นตอนที่ 2. อะแดปเตอร์ผูก
1. เจือจางอะแดปเตอร์ให้มีความเข้มข้นที่เหมาะสมตามตารางที่ 2-5.
2. ละลาย สารเคมีที่ระบุในตาราง 9 พลิกกลับเพื่อผสมสารเคมีให้เข้ากันแล้ววางบนน้ำแข็งเพื่อใช้ในภายหลัง
3. ประกอบสารเคมีตามตาราง 9 บนน้ำแข็ง
ตารางที่ 9 อะแดปเตอร์ผูก ระบบการตอบสนอง
| ส่วนประกอบ | ปริมาตร (μL) |
| ดีเอ็นเอหาง dA-สินค้าจากขั้นตอนที่ 1- | 60 |
| สารเพิ่มประสิทธิภาพการรัด | 30* |
| อะแดปเตอร์ดีเอ็นเอ | 5** |
| รวดเร็ว ที4 ดีเอ็นเอ ไลเกส | 10 |
| ดีดีเอชทูโอ | 5 |
| ทั้งหมด | 110 |
บันทึก:*Ligation Enhancer มีความหนืด โปรดผสมให้เข้ากันโดยการพลิกกลับหรือทำเป็นมุมฉาก ปั่นเหวี่ยงสั้นๆ ก่อนใช้งาน-
-ความเข้มข้นเดิมของ Illuminaทีเอ็ม อะแดปเตอร์ของ YEASE คือ 15 μM โปรดเจือจางอะแดปเตอร์ตามปริมาณอินพุต และ ให้กำหนดปริมาตรของอะแดปเตอร์ให้คงที่ที่ 5 μL
-ความเข้มข้นเดิมของ MGIทีเอ็ม อะแดปเตอร์ของ YEASE คือ 10 μM กรุณาเจือจางอะแดปเตอร์ตามปริมาณอินพุต และ ให้กำหนดปริมาตรของอะแดปเตอร์ให้คงที่ที่ 5 μL
4. ผสมให้เข้ากันโดยค่อยๆ ปิเปตขึ้นและลง และปั่นลงมาสั้นๆ เพื่อรวบรวมของเหลวทั้งหมดจากด้านข้างของหลอด
5. ฟักตัวอย่างในเครื่องปั่นความร้อนที่อุ่นไว้ล่วงหน้าตามที่แสดงในตารางที่ 10 และดำเนินการตอบสนองการเชื่อมต่ออะแดปเตอร์
ตารางที่ 10 โปรแกรมการตอบสนองการเชื่อมต่ออะแดปเตอร์
| อุณหภูมิ | เวลา |
| ตั้งอุณหภูมิฝาให้ร้อนถึง 105°C | ปิด |
| 20 องศาเซลเซียส | 15 นาที |
| 4°C | ถือ |
ขั้นตอนที่ 3. การทำความสะอาดหรือการเลือกขนาดหลังการเชื่อมต่ออะแดปเตอร์
ขั้นตอนนี้คือการฟอกหรือเลือกขนาดผลิตภัณฑ์ในขั้นตอนที่ 2 ด้วยลูกปัดแม่เหล็ก การทำให้บริสุทธิ์สามารถกำจัดอะแดปเตอร์ที่เหลือ ไดเมอร์อะแดปเตอร์ หรือผลิตภัณฑ์อื่น ๆ ที่ใช้ไม่ได้
เคลียน ขึ้นของอะแดปเตอร์ที่เชื่อมดีเอ็นเอ
1. การเตรียมตัว: รับประทานยา Hieff NGSทีเอ็ม นำลูกปัด DNA Selection ออกจากตู้เย็น แล้วปรับสมดุลที่อุณหภูมิห้องเป็นเวลาอย่างน้อย 30 นาที เตรียมเอธานอล 80% สดๆ
2. ผสมลูกปัดให้ทั่วโดยการกลับด้านหรือทำเป็นทรงจุดยอด
3. เพิ่ม 88 ไมโครลิตร ไฮฟ์ เอ็นจีเอสทีเอ็ม ลูกปัดคัดเลือก DNA (0.8×, ลูกปัด : DNA = 0.8:1) ลงในหลอดที่มีผลิตภัณฑ์ที่ต่อเข้ากับอะแดปเตอร์ ปิดและผสมให้เข้ากัน แล้วฟักที่อุณหภูมิห้องเป็นเวลา 5 นาที
4. ปั่นเหวี่ยงสักครู่เพื่อให้สารละลายไหลลงมา จากนั้นวางหลอดปั่นเหวี่ยงบนแท่นแม่เหล็ก เมื่อเม็ดแม่เหล็กถูกดูดซับจนหมด (ประมาณ 5 นาที) ให้ค่อยๆ ดึงของเหลวออก
5. วางหลอดไว้บนขาตั้งแม่เหล็ก เติมเอธานอล 80% ที่เตรียมสดใหม่ 200 μL ลงในหลอดโดยตรง ฟักที่อุณหภูมิห้องเป็นเวลา 30 วินาที แล้วค่อยๆ เอาของเหลวออก
6. ทำซ้ำ ขั้นตอนที่ 5 อีกครั้ง
7. วางหลอดไว้บนแท่นแม่เหล็ก เปิดฝาแล้วเช็ดลูกปัดให้แห้งจนกระทั่งลูกปัดแตกเล็กน้อย (ไม่เกิน 5 นาที)
8. ถอดหลอดออกจากขาตั้งแม่เหล็กเพื่อทำการชะล้าง และชะล้างดีเอ็นเอออกไป
1). หากไม่จำเป็นต้องเลือกขนาดผลิตภัณฑ์ ให้เพิ่ม ddH 21 μL2O โดยตรง ผสมให้เข้ากันโดยใช้เครื่องวอร์เท็กซ์หรือปิเปตขึ้นลง 10 ครั้ง ฟักที่อุณหภูมิห้องเป็นเวลา 5 นาที หมุนหลอดเป็นเวลาสั้นๆ แล้ววางบนแท่นแม่เหล็ก เมื่อสารละลายใส (ประมาณ 5 นาที) ให้ถ่ายของเหลวส่วนบน 20 μL ลงในหลอด PCR ใหม่โดยระวังอย่าให้สัมผัสลูกปัดแม่เหล็ก
2). หากจำเป็นต้องเลือกขนาดผลิตภัณฑ์ ให้เพิ่ม 102 μL ddH2O โดยตรง ผสมให้เข้ากันโดยใช้เครื่องวอร์เท็กซ์หรือปิเปตขึ้นลง 10 ครั้ง ฟักที่อุณหภูมิห้องเป็นเวลา 5 นาที หมุนหลอดทดลองสักครู่แล้ววางบนฐานแม่เหล็ก เมื่อสารละลายใส (ประมาณ 5 นาที) ให้ถ่ายของเหลวส่วนบน 100 μL ลงในหลอด PCR ใหม่โดยระวังอย่าให้สัมผัสลูกปัดแม่เหล็ก
บันทึก:หากจำเป็นต้องจัดเก็บผลิตภัณฑ์ที่ผ่านการทำให้บริสุทธิ์ สามารถชะออกได้ด้วย TE Buffer
การเลือกขนาดของ DNA ที่เชื่อมต่อด้วยอะแดปเตอร์
1.การเตรียมตัว: รับประทานยา Hieff NGSทีเอ็ม นำลูกปัด DNA Selection ออกจากตู้เย็น แล้วปรับสมดุลที่อุณหภูมิห้องเป็นเวลาอย่างน้อย 30 นาที เตรียมเอธานอล 80% สดๆ
2. ผสมลูกปัดให้ทั่วโดยการกลับด้านหรือทำเป็นทรงจุดยอด
3. ตามขนาดเป้าหมาย ให้เพิ่มลูกปัดรอบแรกลงในเทมเพลต DNA ที่บริสุทธิ์ 100 μL ตามตาราง 11ผสมให้เข้ากันโดยใช้การวอร์เท็กซ์หรือปิเปต 10 ครั้ง
โต๊ะ 11 ลูกปัดที่แนะนำ: อัตราส่วน DNA สำหรับการเลือกขนาดลูกปัด
| ขนาดไลบรารี DNA ที่แทรกไว้ | 150 - 250 คู่ | 200-300 บ. | 300-400 คู่ | 400-500 คู่ |
| ขนาดห้องสมุด DNA สุดท้าย | 250-350 บ. | 350-450 บ. | 450-550 บ. | 550-650 บ. |
| อัตราส่วนปริมาตรใน 1 เซนต์ ทรงกลม (ลูกปัด:DNA) | 0.80× | 0.70× | 0.60× | 0.55× |
| อัตราส่วนปริมาตรใน 2 และ ทรงกลม (ลูกปัด:DNA) | 0.20× | 0.20× | 0.20× | 0.15× |
บันทึก:“×” ในตารางระบุปริมาตรของตัวอย่าง DNA ตัวอย่างเช่น หากความยาวของส่วนที่แทรกในคลังข้อมูลคือ 250 bp และปริมาตรของตัวอย่าง DNA คือ 100 μL ปริมาตรของลูกปัดแม่เหล็กที่ใช้ในการคัดแยกรอบแรกคือ 0.7×100 μL=70 μL ปริมาตรของลูกปัดแม่เหล็กที่ใช้ในการคัดแยกรอบที่สองคือ 0.20×100 μL=20 μL ปริมาตรลูกปัดที่แนะนำในตารางนี้ใช้สำหรับ DNA ที่ต่อกับอะแดปเตอร์ หากดำเนินการตามขั้นตอนการเลือกขนาดก่อนการต่อ โปรดดูโปรโตคอลการผลิตของ Hieff NGSทีเอ็ม ลูกปัดคัดแยก DNA (Cat#12601)
4. ฟักที่อุณหภูมิห้องประมาณ 5 นาที
5. หมุนหลอดเป็นเวลาสั้นๆ แล้ววางบนฐานแม่เหล็ก เมื่อสารละลายใส (ประมาณ 5 นาที) ให้ย้ายส่วนที่เป็นของเหลวใสกว่าไปยังหลอด PCR หลอดใหม่
6. เพิ่มลูกปัดคัดเลือกรอบที่สองลงในตัวอย่างจากขั้นตอนที่ 5 ตามตาราง 11ผสมให้เข้ากันโดยการเขย่าหรือปิเปตขึ้นลงอย่างน้อย 10 ครั้ง
7. ฟักที่อุณหภูมิห้องประมาณ 5 นาที
8. ปั่นเหวี่ยงสักครู่เพื่อเอาสารละลายออกมา แล้ววางหลอดปั่นเหวี่ยงบนแท่นแม่เหล็ก เมื่อเม็ดแม่เหล็กถูกดูดซับจนหมด (ประมาณ 5 นาที) ให้ค่อยๆ เอาของเหลวออก
9. วางหลอดไว้บนขาตั้งแม่เหล็ก เติมเอธานอล 80% ที่เตรียมสดใหม่ 200 μL ลงในหลอดโดยตรง ฟักที่อุณหภูมิห้องเป็นเวลา 30 วินาที แล้วค่อยๆ เอาของเหลวออก
10. ทำซ้ำ ขั้นตอนที่ 9 อีกครั้ง.
11. วางหลอดไว้บนแท่นแม่เหล็ก เปิดฝาแล้วเช็ดลูกปัดให้แห้งจนกระทั่งลูกปัดแตกเล็กน้อย (ไม่เกิน 5 นาที)
12. ถอดหลอดออกจากขาตั้งแม่เหล็กเพื่อทำการชะออก แล้ว เพิ่ม ddH 21 μL โดยตรง2O. ผสมให้เข้ากันโดยใช้เครื่องวอร์เท็กซ์หรือปิเปตขึ้นลง และฟักที่อุณหภูมิห้องเป็นเวลา 5 นาที (หมายเหตุ: หากจำเป็นต้องเก็บผลิตภัณฑ์ที่บริสุทธิ์ โปรดชะออกในบัฟเฟอร์ TE) หมุนหลอดลงมาสั้นๆ แล้ววางบนแท่นแม่เหล็กจนกว่าของเหลวจะใส (ประมาณ 5 นาที) ถ่ายของเหลวส่วนบน 20 μL ลงในหลอด PCR ใหม่อย่างระมัดระวัง โดยไม่สัมผัสลูกปัด
ขั้นตอนที่ 4 การขยายเสียงห้องสมุด
ขั้นตอนนี้สามารถเพิ่มความเข้มข้นของผลิตภัณฑ์ที่ผ่านการบริสุทธิ์หรือเลือกขนาดด้วยการขยาย PCR
1. ละลายรายการสารเคมีในตาราง 12 คว่ำลงและผสมให้เข้ากัน แล้ววางไว้บนน้ำแข็งเพื่อใช้ในภายหลัง
2. ประกอบปฏิกิริยาต่อไปนี้ในหลอด PCR ที่ผ่านการฆ่าเชื้อแล้ว
ตารางที่ 12 ปฏิกิริยา PCR ดีเอ็นเอแบบอะแดปเตอร์-ลิเกท ระบบ
| ส่วนประกอบ | ปริมาตร (μL) |
| อะแดปเตอร์เชื่อมต่อดีเอ็นเอ | 20 |
| คานาเซ่ทีเอ็ม มิกซ์ขยายเสียงระดับมืออาชีพ | 25 |
| ไพรเมอร์มิกซ์* | 5 |
| ทั้งหมด | 50 |
[บันทึก]: * หากใช้อะแดปเตอร์แบบครบชุด ไฮฟ์ เอ็นจีเอสทีเอ็ม ไพรเมอร์มิกซ์ -
3. ผสมเบาๆ ด้วยการปิเปตหรือเขย่า แล้วปั่นเป็นเวลาสั้นๆ เพื่อให้สารละลายอยู่ด้านล่าง
4. ใส่หลอดเข้าไปในเครื่อง Thermocycler และตั้งค่าโปรแกรมตามตารางที่ 13 เพื่อเริ่มการขยายเสียง
ตารางที่ 13 โปรแกรมปฏิกิริยาการขยาย PCR
| อุณหภูมิ | เวลา | วงจร |
| ตั้งอุณหภูมิฝาให้ร้อนถึง 105°C | บน | - |
| 98 องศาเซลเซียส | 45 วินาที | 1 |
| 98 องศาเซลเซียส | 15 วินาที | ดูตารางที่ 6 |
| 60 องศาเซลเซียส | 30 วินาที | |
| 72 องศาเซลเซียส | 30 วินาที | |
| 72 องศาเซลเซียส | 1 นาที | 1 |
| 4°C | ถือ | - |
ขั้นตอนที่ 5 การทำความสะอาดหลังการขยายสัญญาณ/การเลือกขนาด
ความสะอาด ขั้นขึ้นไปหมายถึง ก้าว 3. ฮิฟ เอ็นจีเอสทีเอ็ม ลูกปัดคัดเลือก DNA (0.9×, ลูกปัด:DNA=0.9:1) ใช้ในการทำให้ผลิตภัณฑ์ PCR บริสุทธิ์ หากจำเป็นต้องเลือกขนาด โปรดดูที่ ก้าว 3-
ขั้นตอนที่ 6 การควบคุมคุณภาพของห้องสมุดขั้นสุดท้าย
โดยทั่วไปคุณภาพของห้องสมุดที่สร้างขึ้นจะได้รับการประเมินโดยการวัดความเข้มข้นและการกระจายขนาด สำหรับรายละเอียด โปรดดูหมายเหตุ 6
การชำระเงินและความปลอดภัย
ข้อมูลการชำระเงินของคุณได้รับการดำเนินการอย่างปลอดภัย เราไม่เก็บรายละเอียดบัตรเครดิตและไม่สามารถเข้าถึงข้อมูลบัตรเครดิตของคุณได้
การสอบถาม
คุณอาจชอบ
คำถามที่พบบ่อย
ผลิตภัณฑ์นี้มีวัตถุประสงค์เพื่อการวิจัยเท่านั้น และไม่ได้มีวัตถุประสงค์เพื่อใช้ในการรักษาหรือวินิจฉัยโรคในมนุษย์หรือสัตว์ ผลิตภัณฑ์และเนื้อหาได้รับการคุ้มครองโดยสิทธิบัตร เครื่องหมายการค้า และลิขสิทธิ์ที่เป็นของ
แอปพลิเคชั่นบางตัวอาจต้องใช้สิทธิ์ในทรัพย์สินทางปัญญาของบุคคลที่สามเพิ่มเติม