คำอธิบาย
ไฮเอฟเอ็นจีเอส™ OnePot Pro DNA Library Prep Kit V4 เป็นชุดเตรียมไลบรารีเอนไซม์รุ่นใหม่ที่ออกแบบมาเพื่อใช้กับ Illumina และแพลตฟอร์มการจัดลำดับข้อมูลปริมาณสูงของ MGI เมื่อเปรียบเทียบกับวิธีการเตรียมไลบรารีแบบเดิม ชุดนี้ใช้เอนไซม์การแยกส่วนที่มีคุณภาพสูง จึงไม่จำเป็นต้องใช้กระบวนการโซนิเคชั่นที่ยุ่งยาก โมดูลการแยกส่วนและการซ่อมแซมปลายจะรวมกันเป็นขั้นตอนเดียว ซึ่งช่วยลดทั้งเวลาและต้นทุนในการเตรียมไลบรารีได้อย่างมาก ชุดนี้เหมาะสำหรับตัวอย่างประเภทต่างๆ มากมาย รวมถึงจีโนมของพืชและสัตว์ จีโนมของจุลินทรีย์ และอื่นๆ โดยมีช่วงอินพุตตัวอย่างตั้งแต่ 1 นาโนกรัมถึง 1 ไมโครกรัม การแยกส่วนด้วยเอนไซม์ช่วยให้ขนาดของชิ้นส่วนสม่ำเสมอกันในสายพันธุ์ต่างๆ โดยมีความแตกต่างระหว่างสายพันธุ์น้อยที่สุด นอกจากนี้ ชุดนี้ยังจับคู่กับ Illumina ได้อีกด้วย หรือ เอ็มจีไอ อะแดปเตอร์และไพรเมอร์สำหรับการเรียงลำดับบนแพลตฟอร์มปริมาณงานสูง Illumina หรือ MGI
ข้อมูลจำเพาะ
| เลขที่แมว | 12972ES08 / 12972ES24 / 12972ES96 |
| ขนาด | 8 ที/24 ต/96 ที |
ส่วนประกอบ
| ส่วนประกอบ หมายเลข | ชื่อ | 12972ES08 | 12972อีเอส24 | 12972ES96 |
| 12972-ก | 80 ไมโครลิตร | 240 ไมโครลิตร | 960 ไมโครลิตร | |
| 12972-บี | สเมียร์เรส™ เอนไซม์ 4.0 | 80 ไมโครลิตร | 240 ไมโครลิตร | 960 ไมโครลิตร |
| 12972-ซี | สารเพิ่มประสิทธิภาพการรัด 4.0 | 240 ไมโครลิตร | 720 ไมโครลิตร | 3×960 ไมโครลิตร |
| 12972-ดี | รวดเร็ว ดีเอ็นเอ ไลเกส 4.0 | 80 ไมโครลิตร | 240 ไมโครลิตร | 2×480 ไมโครลิตร |
| 12972-อี | มิกซ์ขยายเสียง HF Ultima 2× | 200 ไมโครลิตร | 600 ไมโครลิตร | 3×800 ไมโครลิตร |
พื้นที่จัดเก็บ
ควรเก็บผลิตภัณฑ์นี้ไว้ที่ -25~-15℃ เป็นเวลา 1 ปี.
หมายเหตุ
1. เกี่ยวกับการดำเนินงาน
1. โปรดปฏิบัติงานโดยสวมเสื้อคลุมแล็บและถุงมือแบบใช้แล้วทิ้ง-เพื่อความปลอดภัยของคุณ
2. ละลายส่วนประกอบที่อุณหภูมิห้อง หลังการละลายน้ำแข็งผสมให้เข้ากันด้วยการปั่น หมุนหลอดสั้นๆ แล้ววางลงบนน้ำแข็งเพื่อใช้ในภายหลัง
3. เมื่อเตรียมสารละลายปฏิกิริยาในแต่ละขั้นตอน แนะนำให้ใช้ปิเปตเพื่อผสมให้เข้ากันหรือเขย่าเบาๆ การเขย่าแรงๆ อาจทำให้ปริมาณสารในห้องสมุดลดลง
4. ขอแนะนำให้ใช้ปลายปิเปตแบบกรองเพื่อหลีกเลี่ยงการปนเปื้อนข้าม ควรเปลี่ยนปลายปิเปตเมื่อประมวลผลตัวอย่างที่แตกต่างกัน
5. การดำเนินการที่ไม่เหมาะสมอาจทำให้เกิดการปนเปื้อนของละอองลอย ซึ่งส่งผลต่อความแม่นยำของผลลัพธ์ แนะนำให้แยกบริเวณการผสมปฏิกิริยา PCR และบริเวณการทดสอบการทำให้บริสุทธิ์ของผลิตภัณฑ์ PCR ออกจากกันทางกายภาพ ติดตั้งอุปกรณ์ เช่น ปิเปตเฉพาะทางสำหรับการสร้างห้องสมุดทำความสะอาดตามปกติสำหรับแต่ละพื้นที่โดยเช็ดพื้นผิวด้วยโซเดียมไฮโปคลอไรต์ 0.5% หรือน้ำยาฟอกขาว 10%
6. ผลิตภัณฑ์นี้ใช้เพื่อการวิจัยเท่านั้น
2. การแตกตัวของดีเอ็นเอ1. ชุดนี้ใช้ได้กับ 100 หน้า - 1อินพุต DNA 000 นาโนกรัม ขอแนะนำให้ใช้อินพุต DNA คุณภาพสูงด้วย A260/A280 = 1.8-2.0
2. การทดลองต่อไปนี้อาจได้รับผลกระทบหากนำเกลือที่มีความเข้มข้นสูง เช่น สารคีเลตโลหะ เข้ามาผสมกับดีเอ็นเอที่ป้อนเข้า เราขอแนะนำให้นำตัวอย่างดีเอ็นเอออกมา ดีดีเอช2โอ้ เพื่อการแตกกระจาย
3. กรุณาดูตาราง 6 สำหรับเวลาการแบ่งส่วนของตัวอย่าง DNA มาตรฐานชุดทดสอบนี้มีค่าการแตกตัวต่ำและครอบคลุม GC อย่างสม่ำเสมอสำหรับตัวอย่าง DNA ที่มีองค์ประกอบ GC หลากหลาย กรุณาปรับเวลาการแยกส่วนให้เหมาะสมกับความต้องการในการทดลองของคุณ
4. เพื่อให้การแตกตัวแม่นยำ โปรดเตรียมปฏิกิริยาบนน้ำแข็ง
3. การเชื่อมต่ออะแดปเตอร์
1. ชุด Illumina หรือ MGI Long Adapter (Barcoded Adapter) และชุด Short Adapter มีให้เลือกใช้ตามความต้องการในการทดลองของพวกเขา
2. ขอแนะนำให้เลือกใช้อะแดปเตอร์เชิงพาณิชย์ที่มีคุณภาพสูง หากเลือกอะแดปเตอร์ที่ผลิตเอง โปรดฝากบริษัทที่มีประสบการณ์ในการสังเคราะห์ไพรเมอร์ NGS และระบุถึงความจำเป็นในการควบคุมการปนเปื้อนอย่างเข้มงวด นอกจากนี้ ขอแนะนำให้เตรียมสารละลายแอนนีลลิ่ง DNA ในม้านั่งที่สะอาด และใช้อะแดปเตอร์เพียงชนิดเดียวในแต่ละครั้งเพื่อป้องกันการปนเปื้อนข้าม
3. โปรดละลายอะแดปเตอร์บนน้ำแข็งหรือที่อุณหภูมิ 4°C เมื่อใช้งานที่อุณหภูมิห้อง อุณหภูมิในห้องปฏิบัติการไม่ควรเกิน 25°C เพื่อป้องกันไม่ให้อะแดปเตอร์เสียสภาพ
4. คุณภาพและความเข้มข้นของอะแดปเตอร์จะส่งผลโดยตรงต่อประสิทธิภาพการเชื่อมต่อและผลผลิตของไลบรารี ความเข้มข้นของอะแดปเตอร์ที่สูงเกินไปจะเอื้อต่อการก่อตัวของไดเมอร์ของอะแดปเตอร์ ในขณะที่อะแดปเตอร์ที่น้อยเกินไปจะลดอัตราการเชื่อมต่อและผลผลิตของไลบรารี การเจือจางที่สอดคล้องกับบัฟเฟอร์ TE ตามปริมาณ DNA อินพุตเมื่อใช้อะแดปเตอร์ ตารางที่ 2 -3 แสดงรายการวิธีการเจือจางอะแดปเตอร์ที่แนะนำสำหรับปริมาณ DNA อินพุตที่แตกต่างกันโดยใช้ชุดนี้
ตารางที่ 1 ปริมาณอะแดปเตอร์ Illumina ที่แนะนำสำหรับ DNA อินพุตที่แตกต่างกัน
| อินพุตดีเอ็นเอ | การเจือจางอะแดปเตอร์ (ปริมาตรของอะแดปเตอร์ : ปริมาตรรวม) | ความเข้มข้น |
| 1 ง | 30-พับ (1 : 30- | 0.5 ไมโครโมลาร์ |
| 10 ง | 7.5-พับ (1 : 7.5- | 2 ไมโครโมลาร์ |
| 100 นาโนกรัม | 3-พับ (1 : 3- | 5 ไมโครโมลาร์ |
| 1,000 นาโนกรัม | 1.5-พับ (1 : 1.5- | 10 ไมโครโมลาร์ |
ตารางที่ 2 จำนวนอะแดปเตอร์ MGI ที่แนะนำสำหรับ DNA อินพุตที่แตกต่างกัน
| อินพุตดีเอ็นเอ | การเจือจางอะแดปเตอร์ (ปริมาตรของอะแดปเตอร์ : ปริมาตรรวม) | ความเข้มข้น |
| 1 ง | 20-พับ (1 : 20- | 0.5 ไมโครโมลาร์ |
| 10 ง | 5-พับ (1 : 5- | 2 ไมโครโมลาร์ |
| 100 นาโนกรัม | 2-พับ (1 : 2- | 5 ไมโครโมลาร์ |
| 1,000 นาโนกรัม | ไม่เจือจาง | 10 ไมโครโมลาร์ |
4. การทำความสะอาด DNA และการเลือกขนาดตามลูกปัด
1. ขั้นตอนการเลือกขนาดของชิ้นส่วน DNA สามารถดำเนินการได้หลังการเชื่อมต่ออะแดปเตอร์หรือหลังการขยายไลบรารี
2. ขอแนะนำให้ดำเนินการเลือกขนาดทันทีหลังจากการผูกอะแดปเตอร์หากปริมาณ DNA อินพุตมากกว่า 50 นาโนกรัม- มิฉะนั้นกรุณาเลือกขนาดหลังจากขยาย
3. สารเพิ่มประสิทธิภาพการรัดท่อประกอบด้วย PEG ที่มีความเข้มข้นสูง ซึ่งอาจส่งผลกระทบอย่างมีนัยสำคัญต่อการเลือกขนาดที่แม่นยำ ดังนั้น หากต้องเลือกขนาดทันทีหลังการรัดท่อด้วยอะแดปเตอร์ ขอแนะนำอย่างยิ่งให้เพิ่มขั้นตอนการทำความสะอาดเม็ดบีดก่อนการเลือกขนาด ขั้นตอนการเลือกขนาดสามารถดำเนินการได้โดยตรงหากดำเนินการก่อนการซ่อมแซมปลายท่อ/การต่อ dA หรือหลังจาก การขยายห้องสมุด
4. ควรปรับสมดุลลูกปัดแม่เหล็กที่อุณหภูมิห้องก่อนใช้งาน มิฉะนั้น ผลผลิตจะลดลง และผลการเลือกขนาดจะได้รับผลกระทบ
5. ควรผสมลูกปัดแม่เหล็กให้เข้ากันโดยใช้วิธีวอร์เท็กซ์หรือปิเปตก่อนใช้งาน
6. ห้ามดูดลูกปัดออกเมื่อถ่ายโอนของเหลวส่วนบน แม้ว่าจะมีลูกปัดเพียงเล็กน้อยก็อาจส่งผลต่อปฏิกิริยาต่อไปนี้ได้
7. ควรเตรียมเอธานอล 80% ใหม่ๆ มิฉะนั้น จะส่งผลต่อประสิทธิภาพการกู้คืน
8. เพื่อให้เลือกขนาดได้แม่นยำ ขอแนะนำให้เริ่มด้วยปริมาตรมากกว่า 100 μL หากน้อยกว่านี้ ขอแนะนำให้เพิ่มปริมาตรเป็น 100 μL ด้วยน้ำบริสุทธิ์พิเศษ
9. ควรทำให้ลูกปัดแม่เหล็กแห้งที่อุณหภูมิห้องก่อนทำการชะผลิตภัณฑ์ออก หากผลิตภัณฑ์แห้งไม่เพียงพอ อาจทำให้เอธานอลที่เหลือส่งผลต่อปฏิกิริยาที่เกิดขึ้นในภายหลังได้ง่าย หากแห้งมากเกินไป ลูกปัดแม่เหล็กจะแตกร้าวและลดปริมาณการฟอกให้บริสุทธิ์ โดยปกติแล้ว ควรทำให้ลูกปัดแห้งที่อุณหภูมิห้องเป็นเวลา 3-5 นาทีก็เพียงพอที่จะทำให้ลูกปัดแห้งสนิท
10. หากจำเป็น ตัวอย่าง DNA ที่ผ่านการทำให้บริสุทธิ์หรือเลือกขนาดจะถูกชะออกมา 0.1× สามารถเก็บบัฟเฟอร์ TE ที่อุณหภูมิ 4°C ได้นาน 1-2 สัปดาห์ หรือที่อุณหภูมิ -20°C ได้นาน 1 เดือน
5. การขยายเสียงของห้องสมุด
1. การขยายไลบรารีหรือไม่นั้นขึ้นอยู่กับปริมาณอินพุตของ DNA ประเภทของอะแดปเตอร์ แอปพลิเคชันข้อมูลการจัดลำดับ ฯลฯ จำเป็นต้องทำขั้นตอนการขยายหากใช้อะแดปเตอร์บางส่วน เมื่อใช้อะแดปเตอร์แบบเต็มความยาว หากอินพุต DNA < 200 นาโนกรัม ขอแนะนำให้ทำการขยาย มิฉะนั้น ไม่จำเป็นต้องทำการขยาย
2. จำนวนรอบการขยายสัญญาณควรได้รับการควบคุมอย่างเคร่งครัด การขยายสัญญาณที่ไม่เพียงพออาจส่งผลให้ผลผลิตของไลบรารีต่ำ การขยายสัญญาณมากเกินไปอาจทำให้เกิดอคติ ข้อผิดพลาด การอ่านซ้ำ และผลิตภัณฑ์ไคเมอริกที่เพิ่มขึ้น ตาราง 3 แสดงรายการหมายเลขรอบที่แนะนำโดยตั้งเป้าผลผลิตของห้องสมุดที่ 1 μg
ตารางที่ 3 จำนวนรอบที่แนะนำในการสร้าง ผลผลิตห้องสมุด 1,000 นาโนกรัม
| อินพุตดีเอ็นเอ | จำนวนรอบที่จำเป็นในการสร้างผลผลิตห้องสมุด 1 μg |
| 1,000 นาโนกรัม | 2 - 4 |
| 500 นาโนกรัม | 2 - 4 |
| 250 นาโนกรัม | 4 - 6 |
| 100 นาโนกรัม | 5 - 7 |
| 50 นาโนกรัม | 7 - 9 |
| 1 นาโนกรัม | 12 - 14 |
บันทึก-
1.ตาราง 3 แสดงจำนวนพารามิเตอร์ลูปโดยใช้การทดสอบ DNA อินพุตคุณภาพสูงประมาณ 200 bp คุณภาพของ DNA FFPE แตกต่างกันอย่างมาก และเมื่อคุณภาพของ DNA ไม่ดีหรือความยาวของไลบรารียาว จำนวนรอบจะต้องเพิ่มขึ้นอย่างเหมาะสมเพื่อให้ได้ไลบรารีที่เพียงพอ
2.ฉันจำเป็นต้องเลือกขนาด f ในระหว่างกระบวนการสร้างห้องสมุด ขอแนะนำให้เลือกจำนวนรอบที่สูงกว่าสำหรับการขยายห้องสมุด มิฉะนั้น ขอแนะนำให้เลือกจำนวนรอบที่น้อยกว่า
3.ฉันหากใช้อะแดปเตอร์ที่ไม่สมบูรณ์ จะต้องขยายอย่างน้อย 2 วงจรเพื่อสร้างอะแดปเตอร์ที่สมบูรณ์
6. การวิเคราะห์คุณภาพห้องสมุด
1. คุณภาพของห้องสมุดที่สร้างขึ้นโดยทั่วไปจะได้รับการวิเคราะห์โดยการวัดความเข้มข้นและการกระจายขนาด
2. สามารถวัดความเข้มข้นของห้องสมุดได้ด้วยวิธีที่ใช้ฟลูออเรสเซนต์ เช่น Qubit และ PicoGreen หรือ qPCR
3. ไม่แนะนำให้ใช้การวัดปริมาณตามการดูดกลืนแสง เช่น NanoDrop
4. แนะนำให้ใช้วิธี qPCR สำหรับการวัดปริมาณไลบรารี: วิธีที่ใช้ฟลูออเรสเซนต์ เช่น Qubit และ PicoGreen ไม่สามารถแยกความแตกต่างระหว่างโครงสร้าง dsDNA ที่ไม่สมบูรณ์ (ส่วนที่แทรกโดยไม่มีอะแดปเตอร์หรือส่วนที่ปลายด้านใดด้านหนึ่งถูกผูกด้วยอะแดปเตอร์) จากไลบรารีที่สมบูรณ์ได้ วิธี qPCR จะขยายและวัดไลบรารีที่สมบูรณ์โดยที่ปลายทั้งสองข้างถูกผูกด้วยอะแดปเตอร์เท่านั้น (ไลบรารีที่เรียงลำดับได้) ซึ่งจะทำให้การวัดปริมาณโหลดมีความแม่นยำมากขึ้น
5. สามารถวิเคราะห์การกระจายขนาดของห้องสมุดได้โดยใช้ Agilent Bioanalyzer หรืออุปกรณ์อื่นๆ ที่ใช้หลักการของอิเล็กโทรโฟรีซิสแบบเส้นเลือดฝอยหรือไมโครฟลูอิดิกส์
7. วัสดุอื่น ๆ
1. ลูกปัดแม่เหล็กทำความสะอาด DNA: Hieff NGSทีเอ็ม ลูกปัดคัดแยกดีเอ็นเอ (
2. อะแดปเตอร์: อะแดปเตอร์ครบชุดสำหรับ Illumina-
3. การวิเคราะห์คุณภาพห้องสมุด: Agilent 2100 Bioanalyzer DNA 1000 Chip/High Sensitivity Chip หรือผลิตภัณฑ์เทียบเท่าอื่น ๆ; สารเคมีเชิงปริมาณในห้องสมุด
4. วัสดุอื่นๆ: เอธานอลบริสุทธิ์ น้ำบริสุทธิ์ที่ผ่านการฆ่าเชื้อ ปลายปิเปตที่มีการกักเก็บต่ำ หลอด PCR ขาตั้งแม่เหล็ก เครื่องปั่นความร้อน ฯลฯ
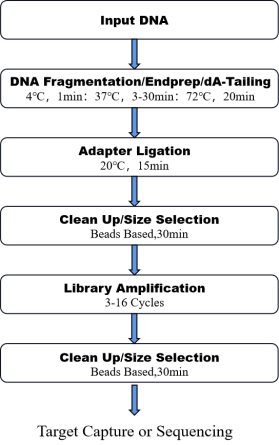
8. เวิร์กโฟลว์
รูปที่ 1.กระบวนการทำงานของ วันพ็อต โปร ดีเอ็นเอ ชุดเตรียมเข้าห้องสมุด
การชำระเงินและความปลอดภัย
ข้อมูลการชำระเงินของคุณได้รับการดำเนินการอย่างปลอดภัย เราไม่เก็บรายละเอียดบัตรเครดิตและไม่สามารถเข้าถึงข้อมูลบัตรเครดิตของคุณได้
การสอบถาม
คุณอาจชอบ
คำถามที่พบบ่อย
ผลิตภัณฑ์นี้มีวัตถุประสงค์เพื่อการวิจัยเท่านั้น และไม่ได้มีวัตถุประสงค์เพื่อใช้ในการรักษาหรือวินิจฉัยโรคในมนุษย์หรือสัตว์ ผลิตภัณฑ์และเนื้อหาได้รับการคุ้มครองโดยสิทธิบัตร เครื่องหมายการค้า และลิขสิทธิ์ที่เป็นของ
แอปพลิเคชั่นบางตัวอาจต้องใช้สิทธิ์ในทรัพย์สินทางปัญญาของบุคคลที่สามเพิ่มเติม