คำอธิบาย
เอชอีเค293 ชุดตรวจจับสารตกค้าง DNA ของเซลล์โฮสต์ใช้สำหรับการวิเคราะห์เชิงปริมาณของ HEK293 การแยกดีเอ็นเอของเซลล์โฮสต์ในตัวอย่างกลาง ผลิตภัณฑ์กึ่งสำเร็จรูปและผลิตภัณฑ์สำเร็จรูปของผลิตภัณฑ์ทางชีวภาพต่างๆ
ชุดนี้ใช้โพรบเรืองแสง Taqman และวิธีปฏิกิริยาลูกโซ่โพลีเมอเรส (PCR) ซึ่งมีขีดจำกัดการตรวจจับขั้นต่ำที่ระดับ fg และสามารถตรวจจับ HEK293 ที่เหลือได้เฉพาะและรวดเร็ว DNA ของเซลล์ การใช้เอนไซม์ UDG ร่วมกับ dUTP สามารถกำจัดการปนเปื้อนที่เกิดจากผลิตภัณฑ์จากการขยายพันธุ์ได้ ชุดอุปกรณ์นี้จะต้องใช้ร่วมกับชุดเตรียมตัวอย่าง DNA ที่เหลือ (Cat# 18461ES)
ข้อมูลจำเพาะ
| เลขที่แมว | 41331ES50/ 41331ES60 |
| ขนาด | 50 ตัน / 100 ตัน |
ส่วนประกอบ
| ส่วนประกอบ หมายเลข | ชื่อ | 41331ES50 | 41331ES60 |
| 41331-ก | เอชอีเค293 qPCR มิกซ์ (UDG พลัส) | 0.75 มล. | 1.5 มล. |
| 41331-ข | เอชอีเค293 ไพรเมอร์&โพรบ มิกซ์ | 250 ไมโครลิตร | 500 ไมโครลิตร |
| 41331-ค | บัฟเฟอร์เจือจางดีเอ็นเอ | 2×1.8 มล. | 4×1.8 มล. |
| 41331-ง | เอชอีเค293 การควบคุมดีเอ็นเอ (30 นาโนกรัม/ไมโครลิตร) | 25 ไมโครลิตร | 50 ไมโครลิตร |
พื้นที่จัดเก็บ
ควรเก็บผลิตภัณฑ์นี้ไว้ที่ -25~-15℃ เป็นเวลา 2 ปี
ทั้ง 41331-A และ 41331-B ควรเก็บรักษาให้พ้นจากแสง
รุ่นเครื่องมือที่ใช้ได้
รวมถึงแต่ไม่จำกัดเพียง:
Bio-Rad: โมดูลออปติก CFX96
เทอร์โมไซแอนทิฟิค: ABI 7500; ABI Quant Studio 5
คำแนะนำ
- เอชอีเค293การเจือจางมาตรฐาน DNA และการเตรียมเส้นโค้งมาตรฐาน
HEK293 รุ่น การควบคุม DNA ได้รับการเจือจางแบบไล่ระดับโดยใช้บัฟเฟอร์เจือจาง DNA ที่ให้มาในชุด-และการเจือจาง
ความเข้มข้นคือ 300 pg/μL, 30 pg/μL, 3 pg/μL, 300 fg/μL, 30 fg/μL
ดูคำแนะนำโดยละเอียดด้านล่าง:
- ละลายบัฟเฟอร์ควบคุมและเจือจาง DNA ของ HEK293DNA บนน้ำแข็ง หลังจากละลายหมดแล้ว ให้ปั่นเบาๆ เพื่อผสมให้เข้ากัน แล้วปั่นด้วยความเร็วต่ำเป็นเวลา 10 วินาที
- นำหลอดทดลองขนาด 1.5 มล. ที่สะอาด จำนวน 6 หลอด ที่มีเครื่องหมาย Std0, Std1, Std2, Std3, Std4 และ Std5 ออก
- เติมบัฟเฟอร์เจือจาง DNA 90 μL และตัวควบคุม HEK293DNA 10 μL ลงในหลอดไมโครฟิวจ์ 1.5 มล. ที่มีฉลาก Std0 ดังนี้ เจือจางเป็น 3 ng/μL ผสมแล้วปั่นเป็นเวลา 10 วินาที บรรจุมาตรฐาน DNA ที่เจือจางแล้ว และสามารถเก็บไว้ได้ในระยะสั้น (ไม่เกิน 3 เดือน) ที่อุณหภูมิ -25~-15℃-.โปรดหลีกเลี่ยงการแช่แข็งและละลายซ้ำๆ
- เติมบัฟเฟอร์เจือจาง DNA 90 μL ลงในหลอดอื่น-จากนั้นทำตามขั้นตอนด้านล่างสำหรับการเจือจางแบบต่อเนื่อง--
| หลอด | อัตราส่วนการเจือจาง | ความเข้มข้นมาตรฐาน |
| ส.ด.1 | บัฟเฟอร์เจือจาง DNA ขนาด 10 μL Std0 + 90 μL | 300 พิกกรัมต่อไมโครลิตร |
| ส.ด.2 | บัฟเฟอร์เจือจาง DNA ขนาด 10 μL Std1 + 90 μL | 30 พิกกรัมต่อไมโครลิตร |
| ส.3 | บัฟเฟอร์เจือจาง DNA ขนาด 10 μL Std2 + 90 μL | 3 พิกกรัมต่อไมโครลิตร |
| ส.4 | บัฟเฟอร์เจือจาง DNA ขนาด 10 μL Std3 + 90 μL | 300 ฟก./ไมโครลิตร |
| ส.5 | บัฟเฟอร์เจือจาง DNA ขนาด 10 μL Std4 + 90 μL | 30 ฟก./ไมโครลิตร |
ตารางที่ 1 การเจือจางแบบไล่ระดับมาตรฐาน
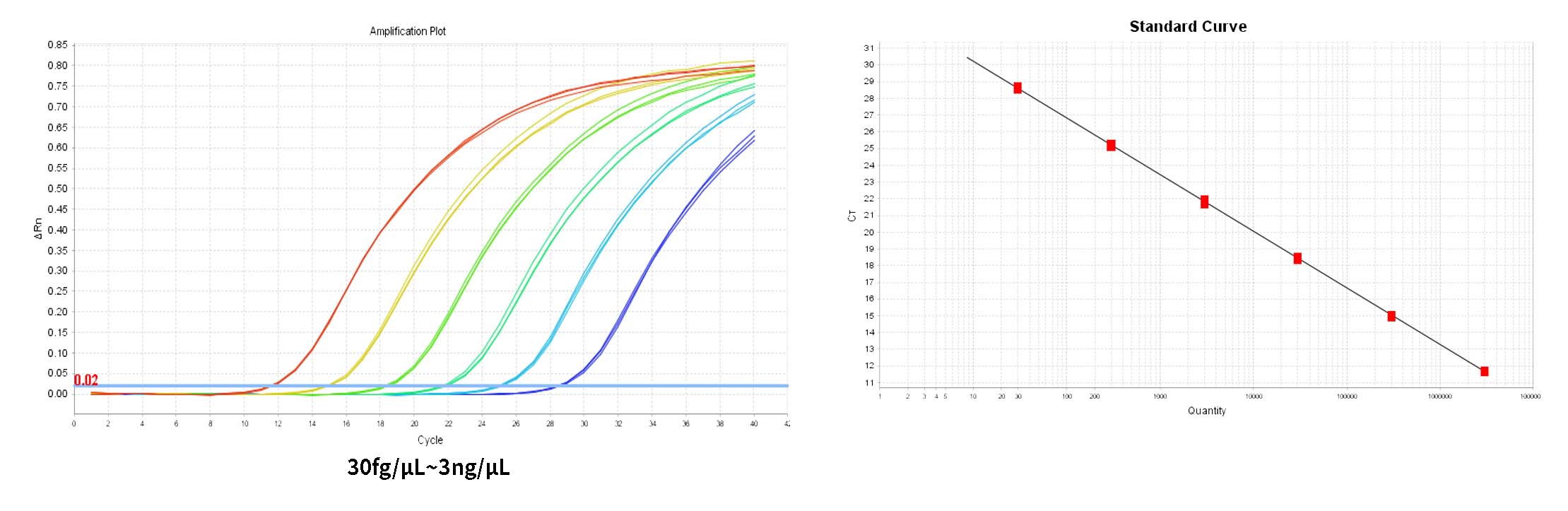
-จำเป็นต้องมีหลุมจำลองสามหลุมสำหรับความเข้มข้นแต่ละแบบ ช่วงการตรวจจับคือ 30 fg/μL~300pg/μL และสามารถขยายช่วงดังกล่าวได้
-เพื่อลดจำนวนครั้งของการแช่แข็ง-ละลายซ้ำและหลีกเลี่ยงการปนเปื้อน ขอแนะนำให้จัดเก็บตัวควบคุม DNA ในส่วนย่อยที่อุณหภูมิ -25~-15℃ เป็นครั้งแรก
-เมื่อละลายแล้ว สามารถเก็บบัฟเฟอร์เจือจาง DNA ไว้ที่อุณหภูมิ 2-8°C ได้นาน 7 วัน หากไม่ได้ใช้เป็นเวลานาน โปรดเก็บที่อุณหภูมิ -25~-15℃
-ตรวจสอบให้แน่ใจว่าเทมเพลตผสมกันหมด เขย่าส่วนผสมเบาๆ เป็นเวลา 15 วินาทีถึง 1 นาทีสำหรับการเจือจางในแต่ละระดับ
- การเตรียมการควบคุมการสกัดและการฟื้นฟู (ERC)
กำหนดความเข้มข้นของ HEK293 DNA ใน ERC ตามต้องการ (ตัวอย่าง ERC เตรียมโดยใช้ HEK293 DNA 30 pg เป็นตัวอย่าง) ดังต่อไปนี้
- เติมตัวอย่างทดสอบ 100 μL ลงในหลอด 1.5 มล. ที่สะอาด จากนั้นเติมมาตรฐาน DNA HEK293 3pg/μL (Std3) 10 μL และผสมให้เข้ากัน โดยทำเครื่องหมายเป็น ERC
- ดำเนินการสกัด DNA จากตัวอย่าง ERC ร่วมกับตัวอย่างทดสอบเพื่อเตรียมตัวอย่าง ERC ที่บริสุทธิ์
- การเตรียมสารละลายควบคุมเชิงลบ (NCS)
ตั้งค่าการควบคุมเชิงลบในการทดลอง ขั้นตอนการดำเนินการเฉพาะมีดังนี้:
1) เติมเมทริกซ์ตัวอย่าง 100 μL (หรือบัฟเฟอร์เจือจาง DNA) ลงในหลอดขนาด 1.5 มล. ที่สะอาด จากนั้นทำเครื่องหมายเป็น NCS
2) ดำเนินการสกัด DNA จากตัวอย่าง NCS ร่วมกับตัวอย่างทดสอบเพื่อเตรียมตัวอย่าง NCS ที่บริสุทธิ์
- การจัดเตรียมการควบคุมเทมเพลต (NTC)
ตั้งค่าการควบคุมแบบไม่มีเทมเพลตในการทดลอง ขั้นตอนการดำเนินการเฉพาะมีดังนี้:
1) NTC ไม่ต้องการการเตรียมตัวอย่างล่วงหน้า และสามารถกำหนดค่าได้ในขั้นตอนการตรวจจับ qPCR ของ DNA ที่เหลือ
2) ตัวอย่าง NTC ในแต่ละหลอดทดลองหรือหลุมคือ 20 μL Mix (เช่น 15 μL HEK293 qPCR Mix + 5 μL HEK293 Primer&probe Mix) + 10 μL DNA Dilution Buffer ขอแนะนำให้กำหนดค่าหลุมจำลองสามหลุม
- ระบบปฏิกิริยา PCR
| ส่วนประกอบ | ปริมาตร(μL) |
| เอชอีเค293 qPCR มิกซ์ (UDG พลัส)- | 15 |
| เอชอีเค293 ไพรเมอร์&โพรบ มิกซ์ | 5 |
| เทมเพลต DNA | 10 |
| ปริมาตรรวม- | 30 |
ตารางที่ 2 ระบบปฏิกิริยา
-คำนวณปริมาตรปฏิกิริยา PCR ทั้งหมดตามจำนวนปฏิกิริยา: qPCR Mix =(จำนวนปฏิกิริยา+2) × (15+5) μL (รวมการสูญเสียของหลุมปฏิกิริยาสองหลุม) แนะนำให้ทำซ้ำมากกว่าสามครั้งสำหรับแต่ละตัวอย่างในการทดลอง
-หลังจากปิดฝาหลอดหรือปิดผนึกแผ่นแล้ว ให้ปั่นหลอดปฏิกิริยาหรือแผ่นด้วยความเร็วต่ำเป็นเวลา 10 วินาที หลังจากเขย่าและผสมอย่างเพียงพอเป็นเวลา 5 วินาที ให้ปั่นซ้ำเพื่อรวบรวมของเหลวจากฝาหรือผนังจนถึงด้านล่าง หลีกเลี่ยงไม่ให้มีฟองอากาศระหว่างการทำงาน
ดูตารางด้านล่างสำหรับการตั้งค่าแผ่นที่แนะนำ:
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| เอ | กทช. |
| ทีเอส 1 | ทีเอส 1 | ทีเอส 1 |
| ชั้นประถมศึกษาปีที่ 1 | ชั้นประถมศึกษาปีที่ 1 | ชั้นประถมศึกษาปีที่ 1 |
|
|
|
| บี | กทช. |
| ทีเอส 2 | ทีเอส 2 | ทีเอส 2 |
| ชั้นประถมศึกษาปีที่ 2 | ชั้นประถมศึกษาปีที่ 2 | ชั้นประถมศึกษาปีที่ 2 |
|
|
|
| ซี | กทช. |
| ทีเอส 3 | ทีเอส 3 | ทีเอส 3 |
| ชั้นประถมศึกษาปีที่ 3 | ชั้นประถมศึกษาปีที่ 3 | ชั้นประถมศึกษาปีที่ 3 |
|
|
|
| ดี |
|
|
|
|
|
| ชั้นประถมศึกษาปีที่ 4 | ชั้นประถมศึกษาปีที่ 4 | ชั้นประถมศึกษาปีที่ 4 |
|
|
|
| อี | เอ็นซีเอส |
| อีอาร์ซี 1 | อีอาร์ซี 1 | อีอาร์ซี 1 |
| ชั้นประถมศึกษาปีที่ 5 | ชั้นประถมศึกษาปีที่ 5 | ชั้นประถมศึกษาปีที่ 5 |
|
|
|
| เอฟ | เอ็นซีเอส |
| อีอาร์ซี 2 | อีอาร์ซี 2 | อีอาร์ซี 2 |
|
|
|
|
|
|
|
| จี | เอ็นซีเอส |
| อีอาร์ซี 3 | อีอาร์ซี 3 | อีอาร์ซี 3 |
|
|
|
|
|
|
|
| ชม |
|
|
|
|
|
|
|
|
|
|
|
|
ตารางที่ 3 คอมพิวเตอร์บนกระดานอ้างอิง
เค้าโครงแผ่นประกอบด้วย: 5 Std (กราฟมาตรฐานของความเข้มข้นมาตรฐาน 5 รายการ), 1 NTC (ไม่มีการควบคุมเทมเพลต), 1 NCS (สารละลายควบคุมเชิงลบ), 3 TS (ตัวอย่างทดสอบ), 3 ERC (การควบคุมการกู้คืนการสกัด)สามหลุมจำลองสำหรับแต่ละตัวอย่าง
- การตั้งค่า แนวทางการใช้เครื่องมือ PCR(วิธีการ 2 ขั้นตอน) (เช่น เครื่องมือ Thermo ABI 7500 qPCR ซอฟต์แวร์เวอร์ชัน 2.0)
คำแนะนำต่อไปนี้ใช้ได้กับเครื่องมือ Thermo ABI 7500 qPCR (ซอฟต์แวร์เวอร์ชัน 2.0) เท่านั้น หากคุณใช้เครื่องมืออื่น โปรดดูคำแนะนำในการตั้งค่าในคู่มือเครื่องมือที่เกี่ยวข้อง
1) สร้างการทดลองใหม่ เลือกเทมเพลตการวัดเชิงปริมาณแบบสัมบูรณ์หรือแบบที่ผู้ใช้กำหนด
2) สร้างโพรบตรวจจับ 1 ตัว ชื่อ "HEK293-DNA" เลือกฟลูออโรโฟร์รีพอร์เตอร์เป็น "FAM" และดับฟลูออโรโฟร์เป็น "None" ฟลูออโรโฟร์อ้างอิงคือ "ROX" (ฟลูออโรโฟร์อ้างอิงอาจขึ้นอยู่กับรุ่นเครื่องมือ เป็นต้น เลือกว่าคุณต้องการเพิ่มหรือไม่)
3) ในบานหน้าต่าง 'ตัวอย่าง' ให้เพิ่มข้อมูลตัวอย่างทั้งหมดตามลำดับ จากนั้นเลือกหลุม เลือกเป้าหมายและตัวอย่างตามลำดับ ตั้งค่างานของ HEK293 มาตรฐาน DNA เป็นมาตรฐาน และกำหนดค่า 300000, 30000, 3000, 300, 30, 30 (หน่วยความเข้มข้นของ DNA ในแต่ละหลุมคือ fg/μL) ในคอลัมน์ Quantity และตั้งชื่อหลุมเป็น Std 1, มาตรฐาน 2, สตด 3, สตด 4, สตด. 5. ตั้งค่างานของ NTC เป็น NTC ตั้งค่า NCS, TS และ ERC โดยไม่ทราบชื่อ และตั้งชื่อตามเค้าโครงแผ่นด้านบน จากนั้นคลิกถัดไป
4) ตั้งค่าโปรแกรมการขยาย: ตั้งค่าปริมาตรปฏิกิริยาเป็น 30 μL
| ขั้นตอนการปั่นจักรยาน | อุณหภูมิ(℃) | เวลา | วงจร |
| การย่อยผลิตภัณฑ์ขยาย | 37℃ | 5 นาที | 1 |
| การเปลี่ยนสภาพเบื้องต้น | 95℃ | 10 นาที | 1 |
| การเปลี่ยนแปลงสภาพธรรมชาติ | 95℃ | 15 วินาที | 40 |
| การอบ/ขยาย (การรวบรวมฟลูออเรสเซนต์) | 60℃ | 30 วินาที |
ตารางที่ 4 ขั้นตอนการขยาย
- การวิเคราะห์ผล qPCR
1) ระบบจะแสดงค่า Threshold ในแผง Amplification Plot ของ Analysis โดยอัตโนมัติ ค่า Threshold ที่ระบบกำหนดไว้บางครั้งอาจใกล้เคียงกับค่าพื้นฐานเกินไป ส่งผลให้ค่า Ct ระหว่างหลุมจำลองแตกต่างกันมาก คุณสามารถปรับค่า Threshold ด้วยตนเองไปยังตำแหน่งที่เหมาะสมและคลิก Analyze จากนั้นคุณสามารถตรวจดูเบื้องต้นว่าเส้นโค้งการขยายสัญญาณเป็นปกติหรือไม่ใน Multicomponent Plot
2) ในแท็บการวิเคราะห์ผลลัพธ์ ให้ตรวจสอบกราฟเส้นโค้งมาตรฐาน ตรวจสอบค่าสำหรับ R2, ประสิทธิภาพ, ความลาดชัน และจุดตัดแกน Y สำหรับเส้นโค้งมาตรฐานปกติ R²>0.99 90% ≤Eff% ≤110%, -3.6≤ความชัน≤-3.1
3) ในบานหน้าต่าง 'ดูตารางหลุม' ในการวิเคราะห์ ความเข้มข้นของแต่ละตัวอย่างจะแสดงเป็นปริมาณ ซึ่งมีหน่วยเป็น fg/μL โดยสามารถแปลงหน่วยได้ในรายงานการวิเคราะห์
4) การตั้งค่าพารามิเตอร์ของการวิเคราะห์ผลจำเป็นต้องขึ้นอยู่กับรุ่นเฉพาะและเวอร์ชันของซอฟต์แวร์ที่ใช้ และโดยทั่วไปเครื่องมือสามารถตีความได้โดยอัตโนมัติ
5) คำนวณอัตราการฟื้นตัวของสไปค์โดยอิงจากผลการทดสอบของ TS ตัวอย่างที่ต้องการวัดและอัตราการฟื้นตัวของสไปค์ตัวอย่าง ERC โดยอัตราการฟื้นตัวของสไปค์จำเป็นต้องอยู่ระหว่าง 50%~150% สูตรมิเตอร์อัตราการฟื้นตัวของสไปค์: การกู้คืน (%) = {Sample spiked assay (eg.pg/μL) - Sample assay (eg.pg/μL)} x ปริมาตรการชะออก (μL) / ค่าเชิงทฤษฎีของปริมาณการเติม DNA (เช่น pg) x 100%
6) ค่า Ct ของ NCS ควบคุมเชิงลบควรมากกว่าค่าเฉลี่ยของ Ct ความเข้มข้นต่ำสุดของมาตรฐาน
- เทมเพลตควบคุม NTC แบบฟรีควรไม่ถูกกำหนดหรือค่า Ct ≥3
หมายเหตุ
- ผลิตภัณฑ์นี้ใช้เพื่อการวิจัยเท่านั้น
- เพื่อความปลอดภัยของคุณ โปรดปฏิบัติงานโดยสวมเสื้อคลุมแล็บและถุงมือแบบใช้แล้วทิ้ง
3. โปรดอ่านคู่มือนี้อย่างละเอียดก่อนใช้สารเคมีนี้ และการทดลองควรได้มาตรฐาน รวมถึงการจัดการตัวอย่าง การเตรียมระบบปฏิกิริยา และการเติมตัวอย่าง
4. ตรวจสอบให้แน่ใจว่าส่วนประกอบแต่ละชิ้นถูกปั่นด้วยความเร็วต่ำและปั่นด้วยความเร็วต่ำก่อนใช้งาน
การชำระเงินและความปลอดภัย
ข้อมูลการชำระเงินของคุณได้รับการดำเนินการอย่างปลอดภัย เราไม่เก็บรายละเอียดบัตรเครดิตและไม่สามารถเข้าถึงข้อมูลบัตรเครดิตของคุณได้
การสอบถาม
คุณอาจชอบ
คำถามที่พบบ่อย
ผลิตภัณฑ์นี้มีวัตถุประสงค์เพื่อการวิจัยเท่านั้น และไม่ได้มีวัตถุประสงค์เพื่อใช้ในการรักษาหรือวินิจฉัยโรคในมนุษย์หรือสัตว์ ผลิตภัณฑ์และเนื้อหาได้รับการคุ้มครองโดยสิทธิบัตร เครื่องหมายการค้า และลิขสิทธิ์ที่เป็นของ
แอปพลิเคชั่นบางตัวอาจต้องใช้สิทธิ์ในทรัพย์สินทางปัญญาของบุคคลที่สามเพิ่มเติม