คำอธิบาย
โช ชุดตรวจจับสารตกค้าง DNA ของเซลล์โฮสต์ใช้สำหรับการวิเคราะห์เชิงปริมาณของ CHO การแยกดีเอ็นเอของเซลล์โฮสต์ในตัวอย่างกลาง ผลิตภัณฑ์กึ่งสำเร็จรูปและผลิตภัณฑ์สำเร็จรูปของผลิตภัณฑ์ทางชีวภาพต่างๆ
ชุดนี้ใช้โพรบเรืองแสง Taqman และวิธีปฏิกิริยาลูกโซ่โพลีเมอเรส (PCR) ซึ่งมีขีดจำกัดการตรวจจับขั้นต่ำที่ระดับ fg และสามารถตรวจจับ CHO ที่เหลือได้เฉพาะและรวดเร็ว ดีเอ็นเอของเซลล์ ชุดอุปกรณ์นี้ต้องใช้ร่วมกับชุดเตรียมตัวอย่างดีเอ็นเอที่เหลือ (Cat# 18461ES)
ผลิตภัณฑ์ ส่วนประกอบ
| เลขที่ | ชื่อ | 41332อีเอส50 -50T- | 41332อีเอส60 -100ตัน- |
| 41332-ก | โช คิวพีซีอาร์ ผสม | 0.75 มล. | 1.5 มล. |
| 41332-ข | โช ไพรเมอร์&โพรบ ผสม | 250 ไมโครลิตร | 500 ไมโครลิตร |
| 41332-ค | การเจือจางดีเอ็นเอ บัฟเฟอร์ | 2×1.8 มล. | 4×1.8 มล. |
| 41332-ง | โช การควบคุมดีเอ็นเอ (30 นาโนกรัม/ไมโครลิตร) | 25 ไมโครลิตร | 50 ไมโครลิตร |
การขนส่งและการเก็บรักษา
1. จัดส่งด้วยน้ำแข็งแห้ง และเก็บรักษาที่อุณหภูมิ -20°C สำหรับ 2 ปี
ควรเก็บผลิตภัณฑ์นี้ไว้ที่ -25~-15℃ เป็นเวลา 2 ปี
ทั้ง 41332-A และ 41332-B ควรเก็บรักษาให้พ้นจากแสง
รุ่นเครื่องมือที่ใช้ได้
รวมถึงแต่ไม่จำกัดเพียง:
Bio-Rad: โมดูลออปติก CFX96
เทอร์โมไซแอนทิฟิค: ABI 7500; ABI Quant Studio 5
คำแนะนำ
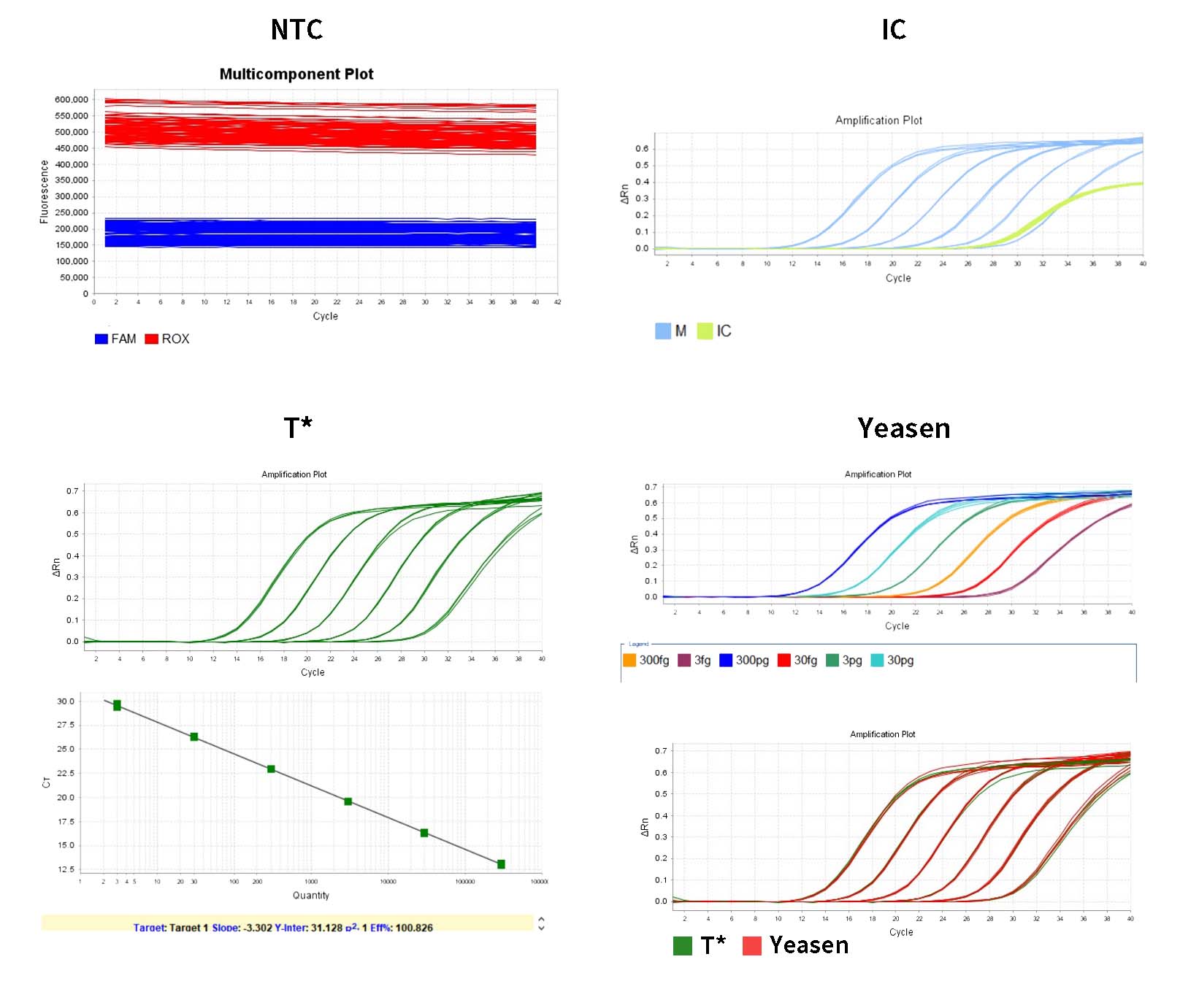
- โช การเจือจางมาตรฐาน DNA และการเตรียมเส้นโค้งมาตรฐาน
สำนักงานเทศบาล การควบคุม DNA ได้รับการเจือจางแบบไล่ระดับโดยใช้บัฟเฟอร์เจือจาง DNA ที่ให้มาในชุด-และการเจือจาง
ความเข้มข้นคือ 3 ng/μL, 300 pg/μL, 30 pg/μL, 3 pg/μL, 300 fg/μL, 30 fg/μL, 3 fg/μL
ดูคำแนะนำโดยละเอียดด้านล่าง:
- ละลายบัฟเฟอร์ควบคุม DNA และเจือจาง DNA ของ CHO บนน้ำแข็ง หลังจากละลายหมดแล้ว ให้ปั่นเบาๆ เพื่อผสมให้เข้ากัน แล้วปั่นด้วยความเร็วต่ำเป็นเวลา 10 วินาที
- นำหลอดทดลองขนาด 1.5 มล. ที่สะอาด 7 หลอด ซึ่งมีเครื่องหมาย Std0, Std1, Std2, Std3, Std4, Std5, Std6 ออก
- เติมบัฟเฟอร์เจือจาง DNA 90 μL และ CHODNA Control 10 μL ลงในหลอดไมโครฟิวจ์ 1.5 มล. ที่มีฉลาก Std0 ดังนี้ เจือจางเป็น 3 ng/μL ผสมแล้วปั่นเป็นเวลา 10 วินาที บรรจุมาตรฐาน DNA ที่เจือจางแล้ว และสามารถเก็บไว้ได้ในระยะสั้น (ไม่เกิน 3 เดือน) ที่อุณหภูมิ -25~-15℃-.โปรดหลีกเลี่ยงการแช่แข็งและละลายซ้ำๆ
- เติมบัฟเฟอร์เจือจาง DNA 90 μL ลงในหลอดอื่น-จากนั้นทำตามขั้นตอนด้านล่างสำหรับการเจือจางแบบต่อเนื่อง--
| หลอด | อัตราส่วนการเจือจาง | ความเข้มข้นมาตรฐาน |
| ส.ด.1 | บัฟเฟอร์เจือจาง DNA ขนาด 10 μL Std0 + 90 μL | 300 พิกกรัมต่อไมโครลิตร |
| ส.ด.2 | บัฟเฟอร์เจือจาง DNA ขนาด 10 μL Std1 + 90 μL | 30 พิกกรัมต่อไมโครลิตร |
| ส.3 | บัฟเฟอร์เจือจาง DNA ขนาด 10 μL Std2 + 90 μL | 3 พิกกรัมต่อไมโครลิตร |
| ส.4 | บัฟเฟอร์เจือจาง DNA ขนาด 10 μL Std3 + 90 μL | 300 ฟก./ไมโครลิตร |
| ส.5 | บัฟเฟอร์เจือจาง DNA ขนาด 10 μL Std4 + 90 μL | 30 ฟก./ไมโครลิตร |
| ชั้น ม.6 | 10 μL Std5 บัฟเฟอร์เจือจาง DNA ขนาด 90 μL | 3 ฟก./ไมโครลิตร |
ตารางที่ 1 การเจือจางแบบไล่ระดับมาตรฐาน
-จำเป็นต้องมีหลุมจำลองสามหลุมสำหรับความเข้มข้นแต่ละแบบ ช่วงการตรวจจับคือ 3 fg/μL~300pg/μL และสามารถขยายช่วงนี้ได้
-เพื่อลดจำนวนครั้งของการแช่แข็ง-ละลายซ้ำและหลีกเลี่ยงการปนเปื้อน ขอแนะนำให้จัดเก็บตัวควบคุม DNA ในส่วนย่อยที่อุณหภูมิ -25~-15℃ เป็นครั้งแรก
-เมื่อละลายแล้ว สามารถเก็บบัฟเฟอร์เจือจาง DNA ไว้ที่อุณหภูมิ 2-8°C ได้นาน 7 วัน หากไม่ได้ใช้เป็นเวลานาน โปรดเก็บที่อุณหภูมิ -25~-15℃
-ตรวจสอบให้แน่ใจว่าเทมเพลตผสมกันหมด เขย่าส่วนผสมเบาๆ เป็นเวลา 15 วินาทีถึง 1 นาทีสำหรับการเจือจางในแต่ละระดับ
- การเตรียมการควบคุมการสกัดและการฟื้นฟู (ERC)
กำหนดความเข้มข้นของ CHO DNA ใน ERC ตามต้องการ (ตัวอย่าง ERC เตรียมโดยใช้ CHO DNA 30 pg เป็นตัวอย่าง) ดังต่อไปนี้
- เติมตัวอย่างทดสอบ 100 μL ลงในหลอด 1.5 มล. ที่สะอาด จากนั้นเติม CHO DNA Standard (Std3) 3pg/μL 10 μL และผสมให้เข้ากัน โดยทำเครื่องหมายเป็น ERC
- ดำเนินการสกัด DNA จากตัวอย่าง ERC ร่วมกับตัวอย่างทดสอบเพื่อเตรียมตัวอย่าง ERC ที่บริสุทธิ์
- การเตรียมสารละลายควบคุมเชิงลบ (NCS)
ตั้งค่าการควบคุมเชิงลบในการทดลอง ขั้นตอนการดำเนินการเฉพาะมีดังนี้:
1) เติมเมทริกซ์ตัวอย่าง 100 μL (หรือบัฟเฟอร์เจือจาง DNA) ลงในหลอดขนาด 1.5 มล. ที่สะอาด จากนั้นทำเครื่องหมายเป็น NCS
2) ดำเนินการสกัด DNA จากตัวอย่าง NCS ร่วมกับตัวอย่างทดสอบเพื่อเตรียมตัวอย่าง NCS ที่บริสุทธิ์
- การจัดเตรียมการควบคุมเทมเพลต (NTC)
ตั้งค่าการควบคุมแบบไม่มีเทมเพลตในการทดลอง ขั้นตอนการดำเนินการเฉพาะมีดังนี้:
1) NTC ไม่ต้องการการเตรียมตัวอย่างล่วงหน้า และสามารถกำหนดค่าได้ในขั้นตอนการตรวจจับ qPCR ของ DNA ที่เหลือ
2) ตัวอย่าง NTC ในแต่ละหลอดทดลองหรือหลุมคือ 20 μL Mix (เช่น 15 μL CHO qPCR Mix + 4 μL CHO Primer&probe Mix + 1 μL IC) + 1 μL DNA Dilution Buffer ขอแนะนำให้กำหนดค่าหลุมจำลองสามหลุม
- ระบบปฏิกิริยา PCR
| ส่วนประกอบ | ปริมาตร(μL) |
| โช qPCR มิกซ์- | 15 |
| โช ไพรเมอร์&โพรบ มิกซ์ | 5 |
| เทมเพลต DNA | 10 |
| ปริมาตรรวม- | 30 |
ตารางที่ 2 ระบบปฏิกิริยา
-คำนวณปริมาตรปฏิกิริยา PCR ทั้งหมดตามจำนวนปฏิกิริยา: qPCR Mix =(จำนวนปฏิกิริยา+2) × (15+4+1) μL (รวมการสูญเสียของหลุมปฏิกิริยาสองหลุม) แนะนำให้ทำซ้ำมากกว่าสามครั้งสำหรับแต่ละตัวอย่างในการทดลอง
-หลังจากปิดฝาหลอดหรือปิดผนึกแผ่นแล้ว ให้ปั่นหลอดปฏิกิริยาหรือแผ่นด้วยความเร็วต่ำเป็นเวลา 10 วินาที หลังจากเขย่าและผสมอย่างเพียงพอเป็นเวลา 5 วินาที ให้ปั่นซ้ำเพื่อรวบรวมของเหลวจากฝาหรือผนังจนถึงด้านล่าง หลีกเลี่ยงไม่ให้มีฟองอากาศระหว่างการทำงาน
ดูตารางด้านล่างสำหรับการตั้งค่าแผ่นที่แนะนำ:
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| เอ | กทช. |
| ทีเอส 1 | ทีเอส 1 | ทีเอส 1 |
| ชั้นประถมศึกษาปีที่ 1 | ชั้นประถมศึกษาปีที่ 1 | ชั้นประถมศึกษาปีที่ 1 |
|
|
|
| บี | กทช. |
| ทีเอส 2 | ทีเอส 2 | ทีเอส 2 |
| ชั้นประถมศึกษาปีที่ 2 | ชั้นประถมศึกษาปีที่ 2 | ชั้นประถมศึกษาปีที่ 2 |
|
|
|
| ซี | กทช. |
| ทีเอส 3 | ทีเอส 3 | ทีเอส 3 |
| ชั้นประถมศึกษาปีที่ 3 | ชั้นประถมศึกษาปีที่ 3 | ชั้นประถมศึกษาปีที่ 3 |
|
|
|
| ดี |
|
|
|
|
|
| ชั้นประถมศึกษาปีที่ 4 | ชั้นประถมศึกษาปีที่ 4 | ชั้นประถมศึกษาปีที่ 4 |
|
|
|
| อี | เอ็นซีเอส |
| อีอาร์ซี 1 | อีอาร์ซี 1 | อีอาร์ซี 1 |
| ชั้นประถมศึกษาปีที่ 5 | ชั้นประถมศึกษาปีที่ 5 | ชั้นประถมศึกษาปีที่ 5 |
|
|
|
| เอฟ | เอ็นซีเอส |
| อีอาร์ซี 2 | อีอาร์ซี 2 | อีอาร์ซี 2 |
| ชั้นประถมศึกษาปีที่ 6 | ชั้นประถมศึกษาปีที่ 6 | ชั้นประถมศึกษาปีที่ 6 |
|
|
|
| จี | เอ็นซีเอส |
| อีอาร์ซี 3 | อีอาร์ซี 3 | อีอาร์ซี 3 |
|
|
|
|
|
|
|
| ชม |
|
|
|
|
|
|
|
|
|
|
|
|
ตารางที่ 3 คอมพิวเตอร์บนกระดานอ้างอิง
เค้าโครงแผ่นประกอบด้วย: 6 Std (กราฟมาตรฐานของความเข้มข้นมาตรฐาน 6 ตัว), 1 NTC (ไม่มีการควบคุมเทมเพลต), 1 NCS (สารละลายควบคุมเชิงลบ), 3 TS (ตัวอย่างทดสอบ), 3 ERC (การควบคุมการกู้คืนการสกัด)สามหลุมจำลองสำหรับแต่ละตัวอย่าง
- การตั้งค่า แนวทางการใช้เครื่องมือ PCR(วิธีการ 2 ขั้นตอน) (เช่น เครื่องมือ Thermo ABI 7500 qPCR ซอฟต์แวร์เวอร์ชัน 2.0)
คำแนะนำต่อไปนี้ใช้ได้กับเครื่องมือ Thermo ABI 7500 qPCR (ซอฟต์แวร์เวอร์ชัน 2.0) เท่านั้น หากคุณใช้เครื่องมืออื่น โปรดดูคำแนะนำในการตั้งค่าในคู่มือเครื่องมือที่เกี่ยวข้อง
1) สร้างการทดลองใหม่ เลือกเทมเพลตการวัดเชิงปริมาณแบบสัมบูรณ์หรือแบบที่ผู้ใช้กำหนด
2) สร้างหัววัดการตรวจจับ 1 ตัว ชื่อ "CHO-DNA" เลือกฟลูออโรโฟร์รายงานเป็น "FAM" และดับฟลูออโรโฟร์เป็น "ไม่มี" สร้างหัววัดตรวจจับเพิ่มอีก 1 ตัว ตั้งชื่อว่า "IC" และเลือกสารเรืองแสงรายงานเป็น "CY5" และการดับฟลูออเรสเซนต์เป็น "ไม่มี" ค่าฟลูออเรสเซนต์อ้างอิงคือ "ROX" (ค่าฟลูออเรสเซนต์อ้างอิงอาจขึ้นอยู่กับรุ่นเครื่องมือ ฯลฯ เลือกว่าคุณต้องการเพิ่มหรือไม่)
3) ในบานหน้าต่าง 'ตัวอย่าง' ให้เพิ่มข้อมูลตัวอย่างทั้งหมดตามลำดับ จากนั้นเลือกหลุม เลือกเป้าหมายและตัวอย่างตามลำดับ ตั้งค่างานของ CHO มาตรฐาน DNA เป็นมาตรฐาน และกำหนดค่า 300000, 30000, 3000, 300, 30, 3 (หน่วยความเข้มข้นของ DNA ในแต่ละหลุมคือ fg/μL) ในคอลัมน์ Quantity และตั้งชื่อหลุมว่า Std 1, มาตรฐาน 2, สตด 3, สตด 4, สตด. 5, Std 6 ตามลำดับ ตั้งค่างานของ NTC เป็น NTC ตั้งค่า NCS, TS และ ERC โดยไม่ทราบชื่อ และตั้งชื่อตามเค้าโครงแผ่นด้านบน จากนั้นคลิกถัดไป
4) ตั้งค่าโปรแกรมการขยาย: ตั้งค่าปริมาตรปฏิกิริยาเป็น 30 μL
| ขั้นตอนการปั่นจักรยาน | อุณหภูมิ(℃) | เวลา | วงจร |
| การเปลี่ยนสภาพเบื้องต้น | 95℃ | 5 นาที | 1 |
| การเปลี่ยนแปลงสภาพธรรมชาติ | 95℃ | 15 วินาที | 40 |
| การอบ/ขยาย (การรวบรวมฟลูออเรสเซนต์) | 60℃ | 30 วินาที |
ตารางที่ 4 ขั้นตอนการขยาย
- การวิเคราะห์ผล qPCR
1) ระบบจะแสดงค่า Threshold ในแผง Amplification Plot ของ Analysis โดยอัตโนมัติ ค่า Threshold ที่ระบบกำหนดไว้บางครั้งอาจใกล้เคียงกับค่าพื้นฐานเกินไป ส่งผลให้ค่า Ct ระหว่างหลุมจำลองแตกต่างกันมาก คุณสามารถปรับค่า Threshold ด้วยตนเองไปยังตำแหน่งที่เหมาะสมและคลิก Analyze จากนั้นคุณสามารถตรวจดูเบื้องต้นว่าเส้นโค้งการขยายสัญญาณเป็นปกติหรือไม่ใน Multicomponent Plot
2) ในแท็บการวิเคราะห์ผลลัพธ์ ให้ตรวจสอบกราฟเส้นโค้งมาตรฐาน ตรวจสอบค่าสำหรับ R2, ประสิทธิภาพ, ความลาดชัน และจุดตัดแกน Y สำหรับเส้นโค้งมาตรฐานปกติ R²>0.99 90% ≤Eff% ≤110%, -3.6≤ความชัน≤-3.1
3) ในบานหน้าต่าง 'ดูตารางหลุม' ในการวิเคราะห์ ความเข้มข้นของแต่ละตัวอย่างจะแสดงเป็นปริมาณ ซึ่งมีหน่วยเป็น fg/μL โดยสามารถแปลงหน่วยได้ในรายงานการวิเคราะห์
4) การตั้งค่าพารามิเตอร์ของการวิเคราะห์ผลจำเป็นต้องขึ้นอยู่กับรุ่นเฉพาะและเวอร์ชันของซอฟต์แวร์ที่ใช้ และโดยทั่วไปเครื่องมือสามารถตีความได้โดยอัตโนมัติ
5) คำนวณอัตราการฟื้นตัวของสไปค์โดยอิงจากผลการทดสอบของ TS ตัวอย่างที่ต้องการวัดและอัตราการฟื้นตัวของสไปค์ตัวอย่าง ERC โดยอัตราการฟื้นตัวของสไปค์จำเป็นต้องอยู่ระหว่าง 50%~150% สูตรมิเตอร์อัตราการฟื้นตัวของสไปค์: การกู้คืน (%) = {Sample spiked assay (eg.pg/μL) - Sample assay (eg.pg/μL)} x ปริมาตรการชะออก (μL) / ค่าเชิงทฤษฎีของปริมาณการเติม DNA (เช่น pg) x 100%
6) ค่า Ct ของ NCS ควบคุมเชิงลบควรมากกว่าค่าเฉลี่ยของ Ct ความเข้มข้นต่ำสุดของมาตรฐาน
- เทมเพลตควบคุม NTC แบบฟรีควรไม่ถูกกำหนดหรือค่า Ct ≥3
หมายเหตุ
- ผลิตภัณฑ์นี้ใช้เพื่อการวิจัยเท่านั้น
- เพื่อความปลอดภัยของคุณ โปรดปฏิบัติงานโดยสวมเสื้อคลุมแล็บและถุงมือแบบใช้แล้วทิ้ง
3. โปรดอ่านคู่มือนี้อย่างละเอียดก่อนใช้สารเคมีนี้ และการทดลองควรได้มาตรฐาน รวมถึงการจัดการตัวอย่าง การเตรียมระบบปฏิกิริยา และการเติมตัวอย่าง
4. ตรวจสอบให้แน่ใจว่าส่วนประกอบแต่ละชิ้นถูกปั่นด้วยความเร็วต่ำและปั่นด้วยความเร็วต่ำก่อนใช้งาน
การชำระเงินและความปลอดภัย
ข้อมูลการชำระเงินของคุณได้รับการดำเนินการอย่างปลอดภัย เราไม่เก็บรายละเอียดบัตรเครดิตและไม่สามารถเข้าถึงข้อมูลบัตรเครดิตของคุณได้
การสอบถาม
คุณอาจชอบ
คำถามที่พบบ่อย
ผลิตภัณฑ์นี้มีวัตถุประสงค์เพื่อการวิจัยเท่านั้น และไม่ได้มีวัตถุประสงค์เพื่อใช้ในการรักษาหรือวินิจฉัยโรคในมนุษย์หรือสัตว์ ผลิตภัณฑ์และเนื้อหาได้รับการคุ้มครองโดยสิทธิบัตร เครื่องหมายการค้า และลิขสิทธิ์ที่เป็นของ
แอปพลิเคชั่นบางตัวอาจต้องใช้สิทธิ์ในทรัพย์สินทางปัญญาของบุคคลที่สามเพิ่มเติม