شرح
E.coli کیت تشخیص باقیمانده DNA سلول میزبان برای تجزیه و تحلیل کمی E.coli استفاده می شود باقیمانده DNA سلول میزبان در نمونه های میانی، محصولات نیمه تمام و نهایی محصولات بیولوژیکی مختلف.
این کیت از پروب فلورسنت Taqman و روش واکنش زنجیره ای پلیمراز (PCR) استفاده می کند که حداقل حد تشخیص سطح fg را دارد و می تواند به طور خاص و سریع باقیمانده E.coli را تشخیص دهد. DNA سلولی کیت باید همراه با کیت آماده سازی نمونه DNA باقیمانده (Cat# 18461ES) استفاده شود.
مشخصات
| گربه شماره | 41308ES50-EN / 41308ES60-EN |
| اندازه | 50 T-EN / 100 T-EN |
اجزاء
| اجزای شماره | نام | 41308ES50-EN | 41308ES60-EN |
| 41308-A | E.coli مخلوط qPCR | 0.75 میلی لیتر | 1.5 میلی لیتر |
| 41308-B | E.coli مخلوط پرایمر و پروب | 250 میکرولیتر | 500 میکرولیتر |
| 41308-C | بافر رقت DNA | 2×1.8 میلی لیتر | 4×1.8 میلی لیتر |
| 41308-د | E.coli کنترل DNA (30 نانوگرم در میکرولیتر) | 25 میکرولیتر | 50 میکرولیتر |
ذخیره سازی
این محصول باید به مدت 2 سال در دمای -25 ~ -15 درجه سانتیگراد نگهداری شود.
هر دو 41308-A و 41308-B باید در محافظت از نور نگهداری شوند.
مدل های ابزار قابل اجرا
شامل اما نه محدود به:
Bio-Rad: ماژول نوری CFX96.
Thermo Scientific: ABI 7500; ABI Quant Studio 5.
دستورالعمل ها
- E.coli. رقت استاندارد DNA و تهیه منحنی استاندارد
E.coli کنترل DNA با استفاده از بافر رقت DNA ارائه شده در کیت، گرادیان رقیق شد*و رقیق سازی
غلظت 300 pg/μL، 30 pg/μL، 3 pg/μL، 300 fg/μL، 30 fg/μL است.
دستورالعمل های دقیق را در زیر مشاهده کنید:
- کنترل coliDNA و بافر رقت DNA را روی یخ ذوب کنید. پس از ذوب کامل، به آرامی ورتکس کنید تا مخلوط شود و با سرعت کم به مدت 10 ثانیه سانتریفیوژ کنید.
- لوله های 1.5 میلی لیتری sixclean را که با Std0، Std1، Std2، Std3، Std4، Std5 مشخص شده اند، خارج کنید.
- 90 میکرولیتر بافر رقت DNA و 10 میکرولیتر coliDNA Control را به لوله میکروفیوژ 1.5 میلی لیتری با برچسب Std0 اضافه کنید. تا 3 نانوگرم در میکرولیتر رقیق شود. مخلوط کنید و سپس به مدت 10 ثانیه سانتریفیوژ کنید. استاندارد DNA رقیق شده را بسته بندی کنید و می توان آن را در کوتاه مدت (حداکثر 3 ماه) در دمای -25-~-15 درجه سانتیگراد ذخیره کرد.**.لطفاً از انجماد و ذوب مکرر خودداری کنید.
- 90 میکرولیتر بافر رقت DNA را به لوله های دیگر اضافه کنید***، سپس روش زیر را برای رقت های سریال دنبال کنید****.
| لوله | نسبت رقیق سازی | غلظت استاندارد |
| Std1 | 10 میکرولیتر Std0 + 90 میکرولیتر بافر رقت DNA | 300 pg/μL |
| Std2 | 10 میکرولیتر Std1 + 90 میکرولیتر بافر رقت DNA | 30 pg/μL |
| Std3 | 10 میکرولیتر Std2 + 90 میکرولیتر بافر رقت DNA | 3 pg/μL |
| Std4 | 10 میکرولیتر Std3 + 90 میکرولیتر DNA بافر رقیق سازی | 300 fg/μL |
| Std5 | 10 میکرولیتر Std4 + 90 میکرولیتر DNA بافر رقیق سازی | 30 fg/μL |
جدول 1 رقت شیب استاندارد
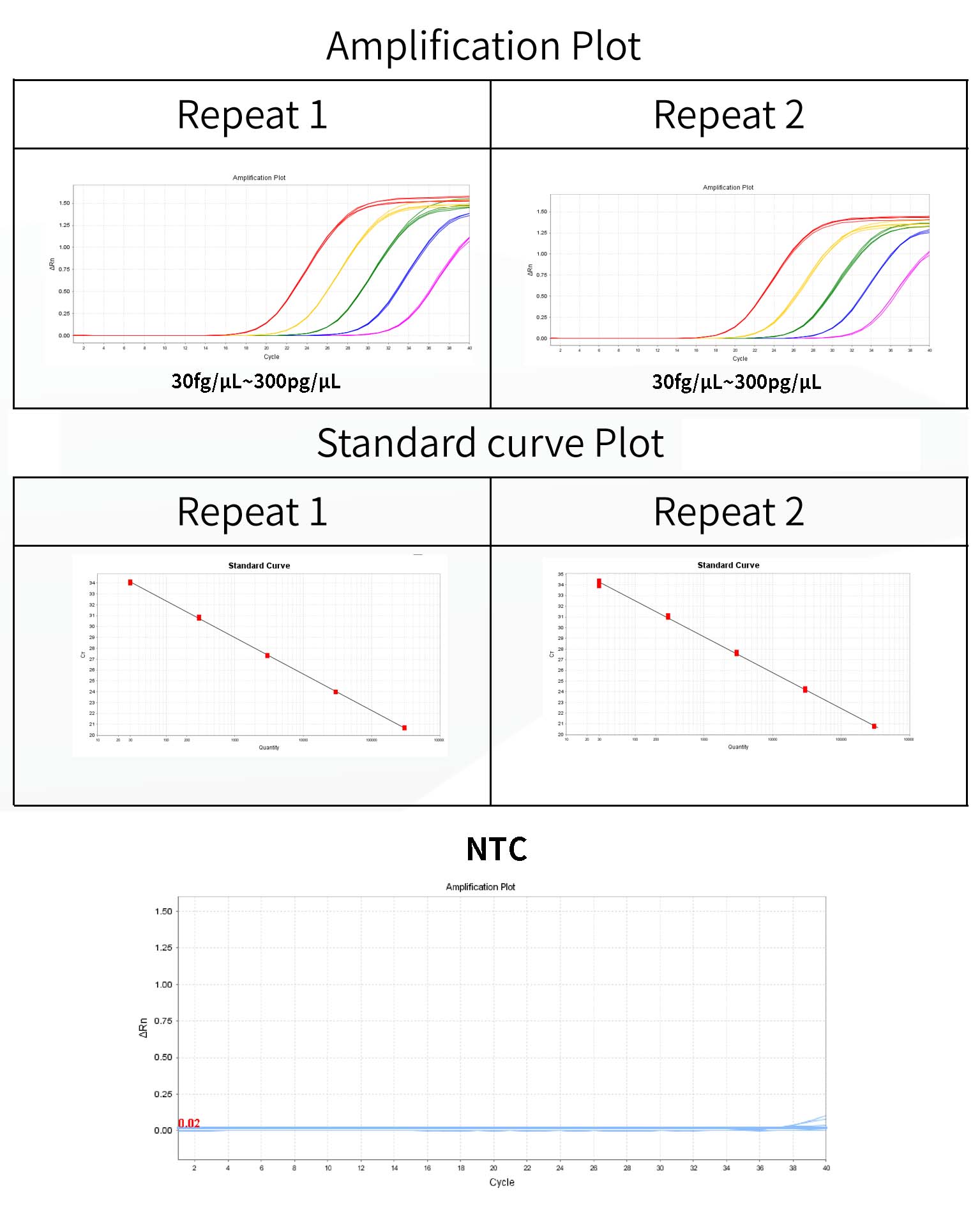
*سه چاه تکرار برای هر غلظت مورد نیاز است. محدوده تشخیص 30 است fg/μL~300pg/μL و این محدوده قابل افزایش است.
**برای کاهش تعداد دفعات انجماد-ذوب مجدد و جلوگیری از آلودگی، توصیه میشود برای اولین بار کنترل DNA را در مقادیر جزئی در دمای -25-~15 درجه سانتیگراد ذخیره کنید.
***پس از ذوب، بافر رقت DNA را می توان در دمای 2-8 درجه سانتیگراد به مدت 7 روز ذخیره کرد، اگر برای مدت طولانی استفاده نمی شود، لطفاً در دمای -25-~-15 درجه سانتیگراد نگهداری شود.
****اطمینان حاصل کنید که قالب کاملا مخلوط شده است، مخلوط را به آرامی به مدت 15 ثانیه تا 1 دقیقه برای هر رقت شیب تکان دهید.
- آماده سازی کنترل بازیابی استخراج (ERC).
غلظت DNA E.coli را در ERC در صورت نیاز تنظیم کنید (نمونه ERC با 30 pg DNA E.coli به عنوان مثال تهیه شد)، به صورت زیر:
- 100 میکرولیتر نمونه آزمایشی را در یک لوله تمیز 1.5 میلی لیتری اضافه کنید، سپس 10 میکرولیتر 3pg/μL E.coli DNA Standard (Std3) را اضافه کنید و با علامت ERC به خوبی مخلوط کنید.
- استخراج DNA از نمونه ERC را همراه با نمونه های آزمایشی انجام دهید تا نمونه ERC خالص شده آماده شود.
- آماده سازی محلول کنترل منفی (NCS).
کنترل منفی را در آزمایش تنظیم کنید، مراحل عملیات خاص به شرح زیر است:
1) 100 میکرولیتر ماتریس نمونه (یا بافر رقت DNA) را به یک لوله تمیز 1.5 میلی لیتری اضافه کنید، سپس به عنوان NCS علامت گذاری کنید.
2) استخراج DNA نمونه NCS را همراه با نمونه های آزمایشی برای تهیه نمونه خالص شده NCS انجام دهید.
- بدون آماده سازی کنترل الگو (NTC).
کنترل بدون الگو را در آزمایش تنظیم کنید، مراحل عملیات خاص به شرح زیر است:
1) NTC نیازی به پیش تیمار نمونه ندارد و می توان آن را در مرحله تشخیص qPCR DNA باقیمانده پیکربندی کرد.
2) نمونه NTC در هر لوله یا چاه 20 میکرولیتر مخلوط (یعنی 15 میکرولیتر E.coli qPCR Mix + 5 میکرولیتر E.coli Primer&probe Mix) + 10 میکرولیتر بافر رقت DNA است. توصیه می شود که سه چاه تکراری را پیکربندی کنید.
- سیستم واکنش PCR
| جزء | حجم (μL) |
| E.coli مخلوط qPCR* | 15 |
| E.coli مخلوط پرایمر و پروب | 5 |
| الگوی DNA | 10 |
| حجم کل** | 30 |
سیستم واکنش جدول 2
*حجم کل واکنش PCR را با تعداد واکنش ها محاسبه کنید: qPCR Mix =(تعداد واکنش ها+2) × (15+5) میکرولیتر (شامل تلفات دو چاه واکنش). بیش از سه تکرار برای هر نمونه در آزمایش توصیه می شود.
**پس از بستن لوله یا آب بندی صفحه، لوله یا صفحه واکنش را با سرعت کم به مدت 10 ثانیه سانتریفیوژ کنید. پس از تکان دادن و مخلوط کردن کافی به مدت 5 ثانیه، سانتریفیوژ را تکرار کنید تا مایع از درب یا دیواره به پایین جمع شود. از ایجاد حباب در حین کار اجتناب کنید.
برای تنظیم صفحه توصیه شده به جدول زیر مراجعه کنید:
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| الف | NTC |
| TS 1 | TS 1 | TS 1 |
| Std 1 | Std 1 | Std 1 |
|
|
|
| ب | NTC |
| TS 2 | TS 2 | TS 2 |
| Std 2 | Std 2 | Std 2 |
|
|
|
| سی | NTC |
| TS 3 | TS 3 | TS 3 |
| Std 3 | Std 3 | Std 3 |
|
|
|
| دی |
|
|
|
|
|
| Std 4 | Std 4 | Std 4 |
|
|
|
| E | NCS |
| ERC 1 | ERC 1 | ERC 1 |
| Std 5 | Std 5 | Std 5 |
|
|
|
| اف | NCS |
| ERC 2 | ERC 2 | ERC 2 |
|
|
|
|
|
|
|
| جی | NCS |
| ERC 3 | ERC 3 | ERC 3 |
|
|
|
|
|
|
|
| اچ |
|
|
|
|
|
|
|
|
|
|
|
|
جدول 3 برد مرجع رایانه روی
طرح صفحه شامل: 5 Std (منحنی استاندارد 5 غلظت استاندارد)، 1 NTC (بدون کنترل الگو)، 1 NCS (محلول کنترل منفی)، 3 TS (نمونه آزمایش)، 3 ERC (کنترل بازیابی استخراج).سه چاه تکرار برای هر نمونه.
- راه اندازی دستورالعمل برای ابزار PCR(روش 2 مرحله ای) (به عنوان مثال. ابزار Thermo ABI 7500 qPCR، نرم افزار نسخه 2.0)
دستورالعمل های زیر فقط برای ابزار Thermo ABI 7500 qPCR (نسخه نرم افزار 2.0) اعمال می شود. اگر از ابزار دیگری استفاده می کنید، برای دستورالعمل های راه اندازی به راهنمای ابزار قابل اجرا مراجعه کنید.
1) یک آزمایش جدید ایجاد کنید، الگوی کمیت مطلق یا تعریف شده توسط کاربر را انتخاب کنید.
2) 1 کاوشگر تشخیص به نام "E.coli-DNA" ایجاد کنید، فلوروفور گزارشگر را به عنوان "FAM" انتخاب کنید و فلوروفور را به عنوان "هیچکدام" خاموش کنید. فلورسانس مرجع ROX است" (فلورسانس مرجع می تواند بر اساس مدل ابزار و غیره باشد، انتخاب کنید که آیا نیاز به اضافه کردن آن دارید یا خیر).
3) در قسمت "نمونه ها"، تمام اطلاعات نمونه ها را به نوبه خود اضافه کنید. سپس چاه ها را انتخاب کنید، هدف و نمونه ها را به ترتیب انتخاب کنید. وظیفه E.coli را تنظیم کنید استاندارد DNA به عنوان استاندارد، و مقادیر 300000، 30000، 3000، 300، 30 (واحد غلظت DNA در هر چاهک fg/μL است) را در ستون Quantity اختصاص دهید و چاه ها را Std نام گذاری کنید. 1، Std 2، Std 3، Std 4، Std 5، به ترتیب. وظیفه NTC را به عنوان NTC تنظیم کنید. NCS، TS و ERC را تنظیم کنید به عنوان ناشناخته، و آنها را مطابق با طرح صفحه بالا نامگذاری کرد. سپس روی next کلیک کنید.
4) برنامه تقویت را تنظیم کنید: حجم واکنش را 30 میکرولیتر تنظیم کنید.
| مرحله چرخه | دما (℃) | زمان | چرخه ها |
| دناتوراسیون اولیه | 95 درجه سانتیگراد | 10 دقیقه | 1 |
| دناتوره سازی | 95 درجه سانتیگراد | 15 ثانیه | 40 |
| بازپخت / گسترش (مجموعه فلورسانس) | 60 درجه سانتیگراد | 30 ثانیه |
جدول 4 روش تقویت
- تجزیه و تحلیل نتایج qPCR
1) سیستم به طور خودکار آستانه را در پنل Amplification Plot تجزیه و تحلیل می دهد. آستانه ارائه شده توسط سیستم گاهی اوقات خیلی نزدیک به خط پایه است و در نتیجه تفاوت زیادی در Ct بین چاهک های تکراری ایجاد می شود. می توانید به صورت دستی آستانه را در یک موقعیت مناسب تنظیم کنید و روی آنالیز کلیک کنید. سپس می توانید در ابتدا بررسی کنید که آیا منحنی تقویت در Plot Multicomponent نرمال است یا خیر.
2) در تب Result Analysis، نمودار منحنی استاندارد را مرور کنید. مقادیر R را تأیید کنید2, کارایی, شیب و رهگیری Y. برای یک منحنی استاندارد نرمال، R²> 0.99، 90%≤Eff%≤110%، -3.6≤شیب≤-3.1.
3) در صفحه "نمایش جدول چاه" در تجزیه و تحلیل، غلظت هر نمونه به مقدار نشان داده شده است، واحد fg/μL است، واحدها را می توان در گزارش سنجش تبدیل کرد.
4) تنظیمات پارامتر تجزیه و تحلیل نتیجه باید بر اساس مدل خاص و نسخه نرم افزار مورد استفاده باشد و به طور کلی می تواند به طور خودکار توسط ابزار تفسیر شود.
5) نرخ بازیابی سنبله را بر اساس نتایج آزمایش TS نمونه اندازه گیری شده و ERC بازیابی سنبله نمونه محاسبه کنید، میزان بازیابی سنبله ها باید بین 50٪ تا 150٪ باشد.فرمول میزان بازیابی سنج: بازیابی (%) = {Sample spiked assay (eg.pg/μL) - Sample assay (eg.pg/μL)} x حجم شستشو (μL) / مقدار نظری مقدار افزودن DNA (به عنوان مثال pg) x 100٪.
6) مقدار Ct کنترل منفی NCS باید بیشتر از میانگین کمترین غلظت Ct استاندارد باشد.
- کنترل بدون الگو NTC باید نامشخص یا مقدار Ct ≥3 باشد
یادداشت ها
- این محصول فقط برای استفاده تحقیقاتی است.
- لطفا برای ایمنی خود با کت های آزمایشگاهی و دستکش های یکبار مصرف کار کنید.
3. لطفاً قبل از استفاده از این معرف، این راهنما را به دقت بخوانید و آزمایش باید استاندارد شود، از جمله جابجایی نمونه، آماده سازی سیستم واکنش و افزودن نمونه.
4. اطمینان حاصل کنید که هر جزء قبل از استفاده به طور کامل ورتکس شده و با سرعت کم سانتریفیوژ شده است.
پرداخت و امنیت
اطلاعات پرداخت شما با اطمینان پردازش می شود. ما جزئیات کارت اعتباری را ذخیره نمی کنیم و به اطلاعات کارت اعتباری شما دسترسی نداریم.
تحقیق
شما همچنین ممکن است دوست داشته باشید
پرسش
این محصول فقط برای اهداف تحقیقاتی است و برای استفاده درمانی یا تشخیصی در انسان یا حیوانات در نظر گرفته نشده است. محصولات و محتوا توسط پتنت ها، علائم تجاری، و حق چاپ متعلق به Yeasen Biotechnology محافظت می شوند. نمادهای علامت تجاری نشان دهنده کشور مبدا هستند، نه لزوما ثبت در همه مناطق.
برخی از برنامههای کاربردی ممکن است به حقوق مالکیت معنوی شخص ثالث دیگری نیاز داشته باشند.
Yeasen به علم اخلاق اختصاص داده شده است و معتقد است که تحقیقات ما باید به سؤالات مهم و در عین حال اطمینان از ایمنی و استانداردهای اخلاقی بپردازد.