PCR フリー ライブラリの構築方法を学習しましたか?
ハイスループットシーケンシングでは、従来のライブラリー構築にはPCR増幅が必要です。一方では、微量DNAサンプルを増幅し、ライブラリー収量を増やすことです。他方では、蛍光信号を増幅できるため、シーケンサーが蛍光信号を捕捉して識別しやすくなり、シーケンシング精度が向上します。しかし、PCRは「両刃の剣」のようなものです。初期サンプル量が少ないという問題を解決し、蛍光信号を増幅する一方で、増幅エラーやバイアスも発生し、ゲノム配列の「素顔」を完全に提示することはできません。さらに、PCRポリメラーゼには一定の増幅バイアスがあります。一部の領域、特に高GCまたは低GC、二次構造、その他の領域は増幅効率が低く、カバーが困難です。また、多数の誤ったInDelを導入し、多数の重複をもたらします。 、DNAデータ量の無駄を引き起こし、シーケンシングのコストを増加させます。したがって、PCRフリー技術の導入は、PCRの利点だけでなく、PCRの欠点も克服します。 PCR フリー ライブラリは、高感度で高品質のライブラリをもたらし、微量サンプルの検出を実現するだけでなく、高い精度を実現し、変異研究のフォローアップ検証の作業負荷を軽減します。では、PCR フリー ライブラリとは正確には何であり、その利点は何でしょうか。
1. PCRフリーライブラリとは何ですか?
2. PCRフリー技術の利点は何ですか?
3.PCRフリーのデータ表示
4. よくある質問
5. 製品情報
6. 読書について
1. PCRフリーライブラリとは何ですか?
次世代シーケンシング技術は、現代の分子生物学におけるハイスループットシーケンシング研究で最も一般的に使用されている技術です。従来の第1世代シーケンシングでは、研究者のニーズを完全に満たすことができなくなりました。モデル生物のゲノム再シーケンシングと非モデル生物のゲノムシーケンシングには、より多くのコストが必要です。低速、高スループット、高速シーケンシング技術により、第2世代シーケンシング技術が誕生しました。第2世代シーケンシング技術の中核原理は、合成によるシーケンシング、つまり、新しく合成された末端標識塩基情報をキャプチャしてDNA配列を決定することです。DNAシーケンシングのための第2世代シーケンシング技術の主な原理は、最初にDNAを断片化し、断片化されたDNAの末端を修復し、次に両側に特定のアダプターを追加し、異なる方法を使用して空間的に固定された数百万のPCRを生成することです。クローンアレイの場合、プライマーハイブリダイゼーションと酵素伸長反応を使用してシーケンシングデータを取得し、各伸長反応によって組み込まれた蛍光ラベルを画像化します。
次世代シーケンシングによる DNA シーケンシングには、主にライブラリの準備とマシン上でのシーケンシングという 2 つのプロセスが含まれます。ライブラリの準備中、ランダムに中断されたゲノム断片は通常、標準 PCR によって増幅されます。ただし、一部の特殊なテンプレートでは、複雑な二次構造や熱安定性の悪さなどの要因があり、テンプレート PCR の増幅優先度に影響するため、すべてのゲノム配列を PCR 増幅ライブラリに均等に反映できるわけではありません。特に、GC や AT 含有量が高い一部のテンプレートでは、PCR 法を使用してライブラリを構築することが困難な場合があります。マシン上でのシーケンシングでは、ライブラリ構築プロセス中に PCR が実行されないことを除いて、PCR フリー ライブラリと従来の PCR ライブラリに違いはありません。PCR フリー ライブラリは、理論的にはデータ読み取りの分布を改善し、より均一なゲノム カバレッジを生成できます。PCR フリー ライブラリは、ライブラリ構築方法を補完するものです。 PCR 増幅なしでオンボード ライブラリに直接準備されるため、一部の高 GC 領域または高 AT 領域のカバレッジが向上し、PCR、データ バイアス、およびシーケンスの繰り返しによって導入されるエラー ベースが削減されます。
従来のNGSライブラリ構築の核心ステップは、ゲノム断片化、末端修復、アダプター付加、PCR、シーケンシング前のシグナル増幅です。では、PCRフリーライブラリ構築はどうでしょうか?その名の通り、PCRを必要としないライブラリ構築プロセスです。ライブラリ構築の核心ステップは、ゲノム断片化、末端修飾、リンカー付加、ライブラリ品質管理です。しかし、実際には、これはライブラリ構築プロセスでのみPCRフリーと見なすことができます。本当のPCRフリーは、ライブラリ構築からシーケンシングまでのプロセスでライブラリ増幅なし(シングル分子シーケンシング技術など)またはPCR増幅エラーが蓄積されないシーケンシング技術(MGIのDNBseqなど)である必要があります。

図1. 従来のライブラリ構築とPCRフリーライブラリ構築のフローチャート
2. PCRフリー技術の利点は何ですか?
通常のライブラリ構築プロセスでは、増幅酵素がエラーを導入する可能性があります。 循環増幅により、これらのエラーが蓄積されて増幅され、DNA配列複製の忠実度が低下します。 さらに、DNAポリメラーゼにも一定の増幅バイアスがあり、特にGC含有量や二次構造に大きな差がある領域では、増幅効率が低下し、カバレッジの均一性が悪くなります。 誤ったInDelを導入すると同時に、大量の重複ももたらし、DNAデータ量の無駄を引き起こし、シーケンシングコストを増加させます。 PCRフリー技術は、従来のライブラリ構築におけるPCR増幅によって導入される上記の問題を十分に回避できます。 ライブラリ構築とシーケンシングのプロセスでPCR増幅プロセスがある場合、ゲノムカバレッジの均一性に大きな影響を与えます。 DNAテンプレートには、複雑な二次構造を持つものもあれば、熱安定性に大きな差があるものもあります。 これらの要因はPCR増幅の効率に影響を与えます。したがって、PCR増幅プロセスでは、すべてのゲノム断片が同じ増幅効率を得られるとは保証できませんが、ゲノムの高GC領域または低GC領域のカバレッジが低いなど、明らかな増幅バイアスがあります。ただし、PCRフリーシーケンシングの全プロセスにはPCRはありません。PCRライブラリ構築と比較して、ゲノムの高GC領域とAT / TAリピート領域のカバレッジが向上します。具体的な利点は次のとおりです。
2.1 ライブラリ準備プロセスの合理化、時間の節約
PCR フリーのライブラリ準備により、PCR 増幅やその他の手順が不要になり、ライブラリ準備プロセスが合理化され、時間が節約されます。
2.2 ライブラリ準備コストの削減
従来のライブラリ作成プロセスでは、配列の信頼性を確保するために、PCR 増幅に高価な高忠実度酵素が使用されます。同時に、PCR-Free では PCR 増幅ステップが不要になるため、ライブラリ作成コストが削減されます。
2.3 増幅バイアスを減らす
DNA 増幅酵素には、特に GC 含有量や二次構造に大きな違いがあるテンプレートの場合、一定の増幅バイアスがあります。そのため、PCR-Free はポリメラーゼによって引き起こされる増幅バイアスを効果的に回避できます。
2.4 PCR重複なし
PCR-Free は重複読み取りを減らし、ユニーク マッピング率と効果的なデータ利用率を高めます。
2.5 エラーの削減
PCR 増幅では、挿入と削除の複製エラーが発生する可能性が高く、インデル配列のエラー率が高くなりますが、PCR フリーでは、このようなエラーの発生と蓄積を効果的に回避できます。
3.PCRフリーのデータ表示
Hieff NGS™ Ultima Pro PCR Free DNA Library Prep Kit V2 (カタログ番号 12196ES) は、競合製品よりも優れたライブラリ変換率を誇ります。
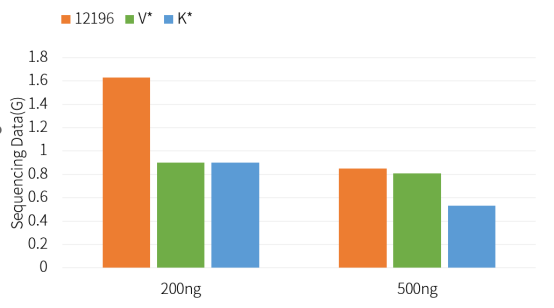
図2. PCRフリーライブラリ構築のためのオフマシンシーケンシングデータの量
4. よくある質問
Q: PCR フリー ライブラリに適したサンプルの種類は何ですか?
A: 二次構造が複雑で、GC が高く、PCR が優先されるサンプルでは、PCR フリーのライブラリを選択して構築できます。
Q: PCR フリー ライブラリの一般的なフラグメント サイズはどれくらいですか?
A: PCRフリーライブラリ自体には、フラグメントサイズに関する特別な要件はありません。PCR産物ライブラリは、PCR産物のサイズに応じてライブラリが構築されます。ゲノムライブラリの場合、ライブラリデータの品質が比較的良好になるように、約350bpが推奨されます。
5. 製品情報
表1. 製品情報
| 製品名 | カタログ番号 | サイズ |
| Hieff NGS™ Ultima Pro PCR フリー DNA ライブラリ準備キット V2 | 12196ES24/96 | 24/96T |
6. 読書について
DNAサンプルからのNGSライブラリ作成ソリューション
NGS関連の技術について、どれくらいご存知ですか?
