説明
Hieff NGS TM EvoMax RNA ライブラリ調製キット (dUTP) は、プレミックスされたアクチノマイシン D フリーで鎖特異的なトータル RNA シーケンシング ライブラリ調製キットで、Illumina および MGI プラットフォームと互換性があります。この製品には、チューブとシーリング キットの 2 種類があり、自動液体処理装置でも手動操作でも、このキットの方が便利です。キットには、RNA 断片化試薬、逆転写試薬、鎖特異的な ds-cDNA 合成試薬、ライブラリ増幅試薬が含まれています。mRNA 精製キットまたは rRNA 除去キットと連携して、mRNA または lncRNA 研究を行うことができます。この製品は、アクチノマイシン D を使用せずに鎖特異性の高いライブラリを取得するために逆転写モジュールを最適化し、実験者の安全性を大幅に確保します。すべての試薬は、ライブラリ調製の安定性と再現性を最大限に高めるために、厳格な品質管理と機能検証を受けています。
仕様
|
カタログ番号 |
12340ES24/12340ES96/12340ES97/12340ES98 |
|
サイズ |
24 T / 96 T / 96 T (オートメーション) / 96 T (プレート) |
コンポーネント
|
コンポーネント番号 |
名前 |
12340ES24 |
12340ES96 |
12340ES97 (自動化) |
12340ES98 (皿 )** |
|
12340-A |
フラグメント/プライムバッファー |
450μL |
2×900μL |
2×1064μL |
8×266μL |
|
12340-B |
1番目の反応モジュール2.0 |
192μL |
768μL |
960μL |
8×120μL |
|
12340-C |
2番目の反応モジュール(dUTP) |
840μL |
3×1120μL |
3×1280μL |
8×480μL |
|
12340-D |
ライゲーション反応モジュール |
840μL |
3×1120μL |
3×1280μL |
8×480μL |
|
12340-E |
2×Super Canace® II ハイファイミックス |
600μL |
2×1200μL |
2×1360μL |
8×340μL |
|
* |
プライマーミックス* |
/ |
/ |
/ |
/ |
注: * 注記は、コンポーネントがキットに含まれていないことを示します。完全なアダプターを使用する場合はプライマー ミックスが必要ですが、そうでない場合は不要です。このキットは Illumina および MGI プラットフォームと互換性がありますが、完全なアダプターを使用する場合は、Illumina® または MGI プラットフォーム用の追加のプライマー ミックス (カタログ番号 13334 Primer Mix for MGI TMおよびカタログ番号 13335 Primer Mix for Illumina®) が必要です。
形
プレート試薬グループのレイアウト。

図1: シーリングプレートライブラリキットの試薬レイアウト

図 2. EvoMax RNA ライブラリ準備キットの簡略化されたコンポーネント。
ストレージ
この製品は-25〜-15℃で1年間保存する必要があります。
説明書
1. 20 μL システムに追加する推奨量は 0.1 ~ 1 単位 (U) であり、実際の結果に基づいて入力量を調整できます。
2. 実験の要求に応じて、dUTP の最終濃度を 0.2~0.6 mM の間で調整し、0.2 mM dTTP を選択的に添加することができます。
3. 25〜37℃での反応時間は、実験の必要に応じて5〜10分以内に調整できます。
注記
1.操作
1) 安全のため、 白衣と使い捨て手袋を着用して作業してください。
2) 室温で成分を解凍します。上下に数回反転させてよく混ぜ、軽くスピンダウンして氷上に置いて使用します。
3) 各ステップの反応は、加熱蓋付きのサーマルサイクラーで実行することをお勧めします。サーマルサイクラーは、使用前に設定温度まで予熱しておく必要があります。
4) RNase 汚染のない備品と実験エリアの定期的な清掃が必要です。RNase 汚染を除去するには、ThermoFisher の RNAZap TM高効率核酸除去スプレーの使用が推奨されます。
5) 不適切な操作はエアロゾル汚染を引き起こし、結果の精度に影響を与える可能性が非常に高くなります。PCR 反応混合領域と PCR 産物精製アッセイ領域を物理的に分離することを必須とすることをお勧めします。ライブラリ構築用の専用ピペットなどの機器を備えています。
6) この試薬は1回限りの使用です。複数回の使用は固く禁じられています。
7) この製品は研究用途にのみ使用できます。
2. アダプターライゲーション
1) イルミナまたは MGI ロング アダプター (バーコード アダプター) キットとショート アダプター キットは、お客様の実験要件に応じて選択できます。
2) 高品質の市販のアダプターを選択することをお勧めします。自作のアダプターを選択する場合は、NGSプライマー合成の経験がある会社に委託し、厳格な汚染管理の必要性に留意してください。また、クロスコンタミネーションを防ぐために、クリーンベンチでDNAアニーリング溶液を調製し、毎回1種類のアダプターのみを操作することをお勧めします。
3) アダプターは氷の上または 4°C で解凍してください。室温で操作する場合は、アダプターの変性を防ぐために、実験室の温度が 25°C を超えないようにしてください。
4) アダプターの濃度は、ライゲーション効率とライブラリ収量に直接影響します。キットに添加されるアダプターの量は 5 μl に固定されています。アダプターは 0.1×TE バッファーで希釈することが推奨されており、希釈したアダプターは 4°C で 48 時間保存できます。表 1 に、入力 RNA のさまざまな量に対する推奨アダプター量を示します。
表1-1 異なる入力RNAに対するイルミナアダプターの推奨量
|
入力総RNA |
Illumina® アダプターストック濃度 |
|
10 ng |
1μM |
|
100 ng |
1.5μM |
|
500 ng |
3μM |
| ≥1μg |
5μM |
表1-2 異なる入力RNAに対する推奨MGIアダプター量
|
入力総RNA |
MGI® アダプターストック濃度 |
|
10〜99 ng |
1μM |
|
100〜499 ng |
2μM |
|
500〜4000ng |
5μM |
* アダプターの使用は、トータルRNAサンプルの種類や入力量に応じて調整できます。
3. ライブラリの拡張
1) キットに含まれる高忠実度 DNA ポリメラーゼは、第 1 世代 DNA ポリメラーゼをベースに、増幅の均一性が大幅に向上し、増幅の偏りが見られません。
2) 増幅サイクル数は厳密に制御する必要があります。増幅が不十分だとライブラリ収量が低下する可能性があり、増幅が多すぎるとバイアス、エラー、重複読み取り、キメラ産物、拡張変異の蓄積が増加する可能性があります。表 2 に PCR 増幅の推奨サイクル数を示します。
3) 表 2 で推奨されているサイクル数は、ライブラリの準備要件の大部分を満たすことができます。サンプルの品質が悪い場合 (劣化が著しい FFPE サンプルなど)、実際の状況に応じてサイクル数を適切に増やすことができます。mRNA は種や組織によって異なるため、増幅サイクル数を調整する必要があることに注意してください。
表2 入力RNA総量と推奨増幅サイクル表*
|
入力総RNA |
サイクル数 |
|
10 ng |
16 |
|
100 ng |
14 |
|
500 ng |
12 |
|
1μg |
11 |
[注意]:*ライブラリの収量は、入力量と増幅サイクル数に関係するだけでなく、サンプルの品質、断片化条件、ソート条件によっても影響を受けます。ライブラリ構築の過程では、実際の状況に応じて最も適切な条件を選択してください。
4. ビーズベースのDNAクリーンアップとサイズ選択
1) ライブラリ構築プロセスには、DNA 精製磁気ビーズを必要とするステップが複数あります。DNA 精製とサイズ選択には、Hieff NGS™ DNA 選択ビーズ (Yeasen Cat#12601) または AMPure® XP 磁気ビーズ (Beckman Cat#A63880) をお勧めします。
2) 磁気ビーズは使用前に室温に調整する必要があります。そうしないと、収量が減少し、サイズ選択効果に影響が出ます。
3) 磁気ビーズは使用前にボルテックスまたはピペッティングでよく混合する必要があります。
4) 上清を移す際にビーズを吸引しないでください。微量のビーズでもその後の反応に影響を与える可能性があります。
5) 80% エタノールは新しく調製する必要があります。そうでないと回収効率に影響します。
6) 磁気ビーズは、生成物を溶出する前に室温で乾燥させる必要があります。乾燥が不十分だと、残留エタノールが後続の反応に影響を与えやすくなります。乾燥が過剰だと、磁気ビーズが割れて精製収率が低下します。通常、室温で 3 ~ 5 分間乾燥させると、ビーズが完全に乾燥します。
7) 必要に応じて、TE バッファーで溶出された精製またはサイズ選択された DNA サンプルは、4°C で 1 ~ 2 週間、または -20°C で 1 か月間保存できます。
5. ライブラリ品質分析
一般的に、構築されたライブラリの品質は、濃度検出と長さ分布検出によって評価できます。
6. その他の資料
1) mRNA濃縮キット: Hieff NGS® mRNA分離マスターキットV2 (Yeasen カタログ番号 12629)。
2) rRNA 除去キット: Hieff NGS® MaxUp Human rRNA Depletion Kit (rRNA & ITS / ETS) (Yeasen Cat # 12257) またはその他の rRNA 除去キット。
3) 磁気ビーズからのRNA精製: Hieff NGS® RNAクリーナー (Yeasen カタログ番号 12602) または同等の製品。
4) DNA精製磁気ビーズ:Hieff NGS® DNA選択ビーズ(Yeasenカタログ番号12601)またはAMPure® XPビーズ(A63880)またはその他の同等製品。
5) RNA品質管理: Agilent 2100 Bioanalyzer RNA 6000 Nano / Pico Chip または同等の製品。
6) アダプタ: Illumina® 用完全アダプタ (カタログ番号 13519-13520 または同等品)、または MGI® 用完全アダプタ (カタログ番号 13360-13362 または同等品)。
7. 図書館の品質検査
Agilent 2100 Bioanalyzer DNA 1000 Chip / High Sensitivity Chip またはその他の同等製品、ライブラリ定量化試薬。
8. その他の資料
無水エタノール、滅菌超純水、低吸着ガンヘッド、PCRチューブ、磁気フレーム、PCR装置など。
フローチャート

図1: RNAライブラリ構築プロセス
さまざまな品質のFFPEサンプル用のRNAライブラリの準備
著しく劣化した FFPE RNA (DV 200 <50%) および低入力サンプルの場合、ライブラリの損失を減らすために、アダプター連結後に二重精製プロトコルを使用することをお勧めします。
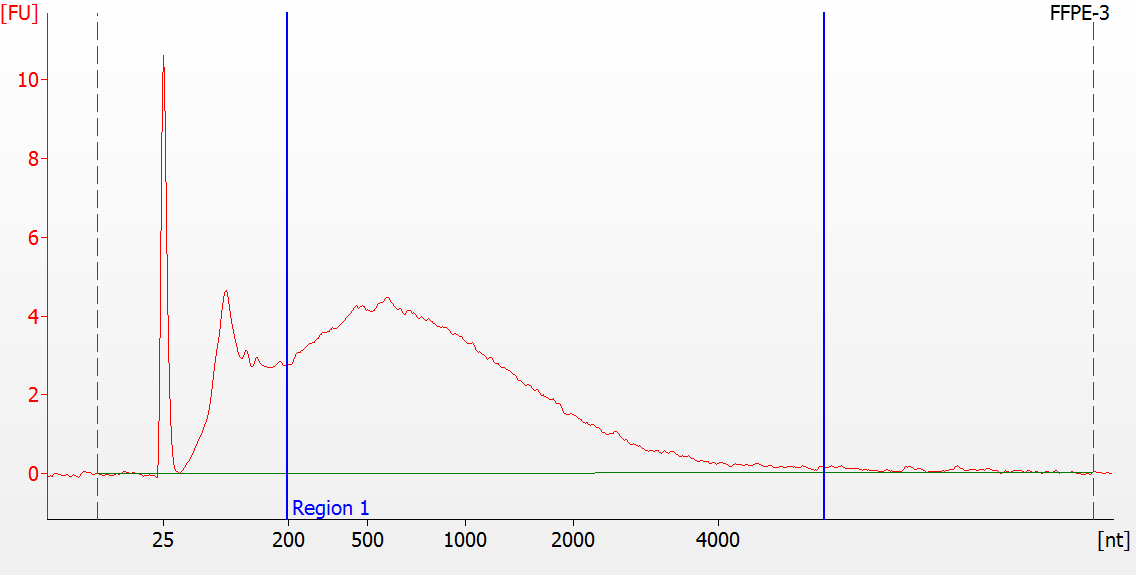
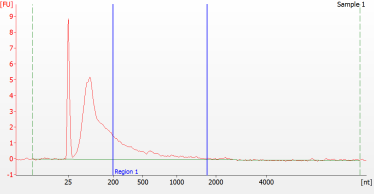
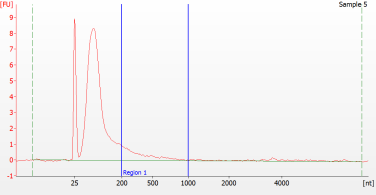
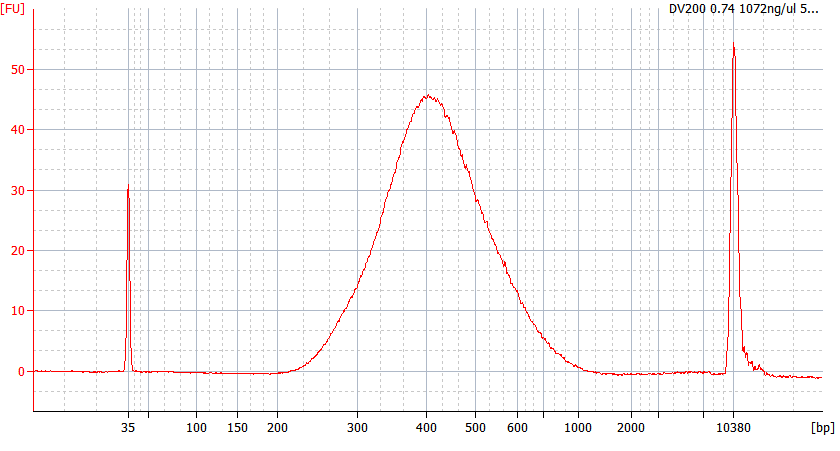
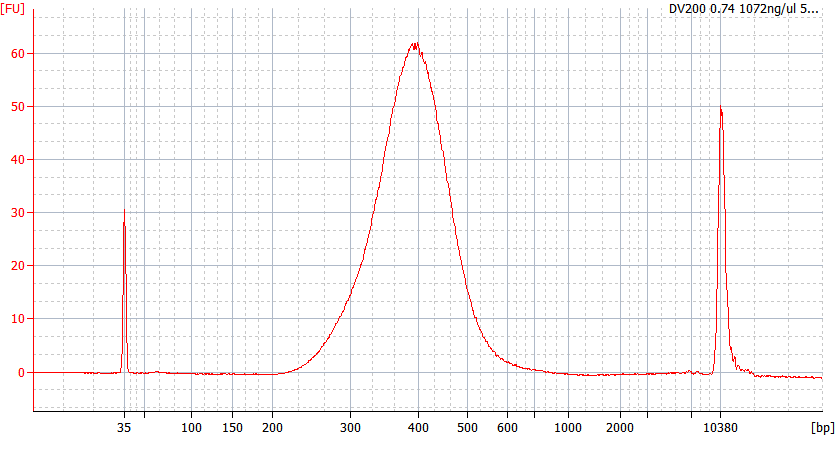
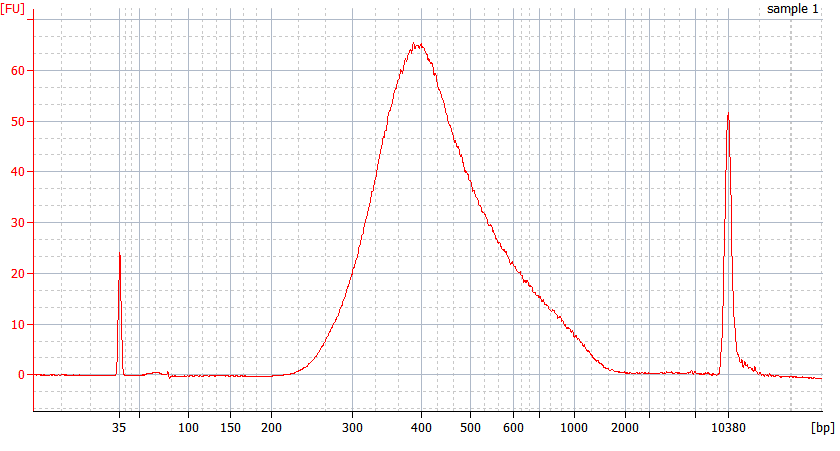
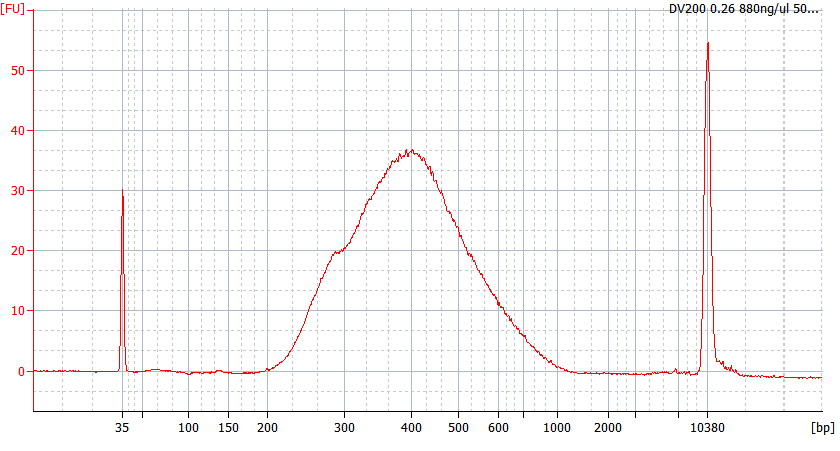
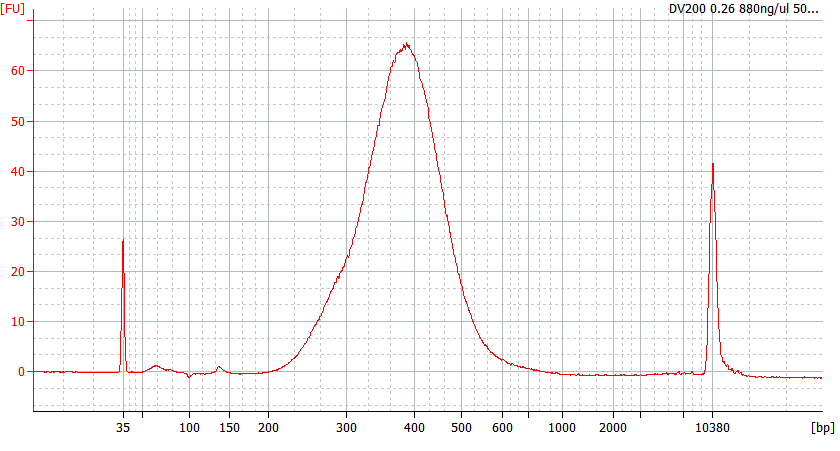
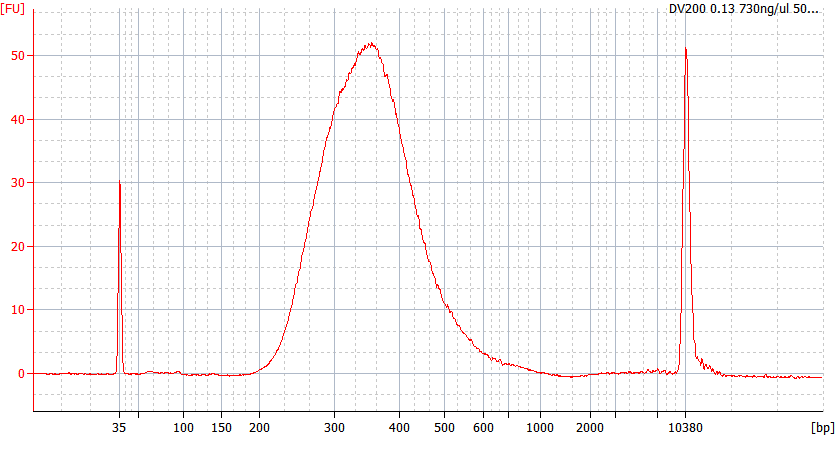
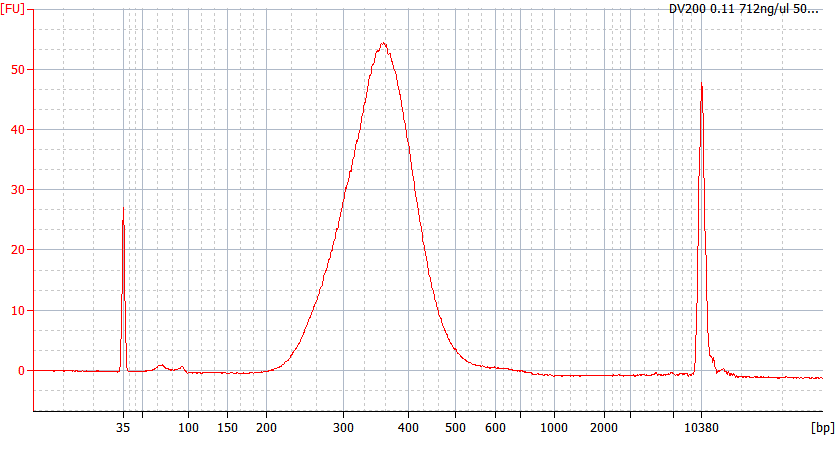
異なる品質のFFPEサンプルのピークパターン
|
FFPE RNAサンプル |
|
|
|
サンプル1 RIN=2.2; DV200=74%
|
|
|
サンプル2 RIN=2.5; DV200=26% |
|
|
サンプル3 RIN=2.5; DV200=11% |
|
|
入力RNA: 500 ng 、 ライブラリ増幅:12サイクル、 ライブラリ収量: 717.2ng |
|
|
入力RNA: 500 ng 断片化: 94℃、7分 ライブラリ増幅: 13 サイクル ライブラリ収量: 437.8 ng |
|
|
入力RNA: 100 ng 断片化: 94℃、7分 二重精製:0.6×、0.8× ライブラリ増幅: 15サイクル ライブラリ収量: 206.8 ng |
|
サンプル2. RNAライブラリ調製結果 |
|
|
|
入力RNA: 500 ng 断片化: 85℃、8分 二重精製:0.6×、0.8× ライブラリ増幅: 12サイクル ライブラリ収量: 207 ng |
|
|
入力RNA: 500 ng 断片化: 85℃、8分 浄化と 選択: 0.6× & 0.7× / 0.15× ライブラリ増幅: 13 サイクル ライブラリ収量: 98.56 ng |
|
サンプル3. RNAライブラリ調製結果 |
|
|
|
入力RNA: 500 ng 断片化: 65℃、8分 二重精製:0.6×、0.8× ライブラリ増幅: 12サイクル ライブラリ収量: 354.2 ng |
|
|
投入量: 500 ng 断片化: 65℃、8分 ライブラリ増幅: 13 サイクル ライブラリ収量: 172.48 ng |
支払いとセキュリティ
お支払い情報は安全に処理されます。 クレジットカードの詳細を保存したり、クレジットカード情報にアクセスすることはありません
問い合わせ
よくある質問
この製品は研究目的のみに使用され、人間や動物の治療や診断に使用することを意図したものではありません。製品とコンテンツは、
特定のアプリケーションでは、追加のサードパーティの知的財産権が必要になる場合があります。