説明
複製能レンチウイルス (RCL) 検出キットは、レンチウイルスベクターの潜在的なリスクに関連するさまざまな細胞製品で発生する可能性のある複製レンチウイルスを定量的に検出するために使用されます。
このキットは、レンチウイルスエンベロープタンパク質の VSV-G 遺伝子配列に特異的なプライマーを設計します。また、Taqman 蛍光プローブとポリメラーゼ連鎖反応 (PCR) 法を採用しており、1 コピー/μL レベルの検出限界を持ち、複製可能なレンチウイルスのリスクを特異的かつ迅速に検出できます。このキットは、残留 DNA サンプル調製キット (カタログ番号 18461ES) と一緒に使用する必要があります。
コンポーネント
|
コンポーネント番号 |
名前 |
41311ES50 |
41311ES60 |
|
41311-A |
RCL qPCR ミックス |
0.75mL |
1.5mL |
|
41311-B |
RCLプライマー&プローブミックス |
200μL |
400μL |
|
41311-C |
DNA希釈バッファー |
2×1.8mL |
4×1.8mL |
|
41311-D |
RCL DNAコントロール(5×10E8コピー/μL)) |
25μL |
50μL |
|
41311-E |
IC* |
50μL |
100μL |
* IC:内部統制。
ストレージ
この製品は-25〜-15℃で2年間保存する必要があります。
41311-A と 41311-B はどちらも光を避けて保管する必要があります。
適用可能な機器モデル
これには、Bio-Rad: CFX96 光学モジュール、Thermo Scientific: ABI 7500、ABI Quant Studio 5、ABI Step OnePlus などが含まれますが、これらに限定されません。
説明書
- RCL DNA標準希釈と標準曲線の作成
RCL DNA コントロールは、キット* に付属の DNA 希釈バッファーを使用して勾配希釈され、希釈濃度は 5×10E7 コピー/μL、5×10E6 コピー/μL、5×10E5 コピー/μL、5×10E4 コピー/μL、5×10E3 コピー/μL、5×10E2 コピー/μL、5×10E1 コピー/μL です。
詳細な手順は以下を参照してください。
1) RCL DNA コントロールと DNA 希釈バッファーを氷上で解凍します。完全に解凍したら、軽くボルテックスして混ぜ、低速で 10 秒間遠心分離します。
2) Std0、Std1、Std2、Std3、Std4、Std5、Std6 とマークされた 7 本の清潔な 1.5 mL チューブを取り出します。
3) DNA希釈バッファー90μLとRCL DNAコントロール10μLを、Std0とラベルされた1.5mLマイクロチューブに加え、5×10E7コピー/μLに希釈します。混合してから10秒間遠心分離します。希釈したDNA標準液を小分けし、-25~-15℃で短期間(3か月以内)保存できます**。凍結融解を繰り返さないでください。
4) 他のチューブ***に90μLのDNA希釈バッファーを加え、以下の手順に従って連続希釈****を行います。
|
チューブ |
希釈率 |
標準濃度 |
|
標準1 |
10 μL Std0 + 90 μL DNA希釈バッファー |
5×10E6コピー/μL |
|
スタンダード2 |
10 μL Std1 + 90 μL DNA希釈バッファー |
5×10E5コピー/μL |
|
スタンダード3 |
10 μL Std2 + 90 μL DNA希釈バッファー |
5×10E4コピー/μL |
|
スタンダード4 |
10 μL Std3 + 90 μL DNA希釈バッファー |
5×10E3コピー/μL |
|
スタンダード5 |
10 μL Std4 + 90 μL DNA希釈バッファー |
5×10E2コピー/μL |
|
6年生 |
10 μL Std5 + 90 μL DNA希釈バッファー |
5×10E1コピー/μL |
表1 標準勾配希釈
*各濃度ごとに3つの複製ウェルが必要です。検出範囲は5×10E1コピー/μL~5×10E6コピー/μLで、必要に応じてこの範囲を拡大できます。
** 凍結融解の繰り返し回数を減らし、汚染を防ぐために、DNAコントロールを初めて保存するときは、小分けにして-25〜-15℃で保存することをお勧めします。
*** 解凍後、DNA希釈バッファーは2〜8℃で7日間保存できます。長期間使用しない場合は、-25〜-15℃で保存してください。
**** テンプレートが完全に混合されていることを確認し、各勾配希釈ごとに混合物を 15 秒から 1 分間軽く振ってください。
- 抽出回収制御(ERC)の準備
必要に応じて、ERC 内の RCL DNA の濃度を次のように設定します (ERC サンプルは、例として 5×10E4 コピーの RCL DNA で準備されました)。
1) 100 μL の試験サンプルを清潔な 1.5 mL チューブに加え、次に 10 μL の 5×10E3 コピー/μL RCL DNA 標準 (Std4) を加えてよく混ぜ、ERC とマークします。
2) ERCサンプルのDNA抽出を試験サンプルとともに行い、精製されたERCサンプルを調製します。
- ネガティブコントロール溶液(NCS)の調製
実験でネガティブコントロールを設定します。具体的な操作手順は次のとおりです。
1) 100 μL のサンプル マトリックス (または DNA 希釈バッファー) を清潔な 1.5 mL チューブに追加し、NCS とマークします。
2) NCS サンプルの DNA 抽出をテストサンプルと一緒に実行して、精製 NCS サンプルを調製します。
- テンプレートコントロール(NTC)の準備なし
実験でテンプレートなしコントロールを設定する場合、具体的な操作手順は次のとおりです。
1) NTC はサンプルの前処理を必要とせず、残留 DNA 含有量の qPCR 検出の段階で設定できます。
2) 各チューブまたはウェルの NTC サンプルは、20 μL ミックス (つまり、15 μL RCL qPCR ミックス + 4 μL RCL プライマー&プローブ ミックス + 1 μL IC) + 10 μL DNA 希釈バッファーです。3 つの複製ウェルを構成することをお勧めします。
- PCR反応システム
|
成分 |
容量(μL) |
|
RCL qPCR ミックス* |
15 |
|
RCLプライマー&プローブミックス |
4 |
|
IC |
1 |
|
DNAテンプレート |
10 |
|
総量** |
30 |
表2 反応システム
* PCR 反応の総量を反応数で計算します: qPCR Mix = (反応数 + 2) × (15 + 4 + 1) μL (2 つの反応ウェルの損失を含む)。実験では、各サンプルに対して 3 回以上の反復が推奨されます。
** チューブにキャップをするかプレートを密封した後、反応チューブまたはプレートを低速で 10 秒間遠心分離します。 5 秒間十分に振とうおよび混合した後、遠心分離を繰り返して、蓋または壁から底に液体を集めます。 操作中に気泡が発生しないようにしてください。
推奨されるプレート設定については、下の表を参照してください。
|
1 |
2 |
3 |
4 |
5 |
6 |
7 |
8 |
9 |
10 |
11 |
12 |
|
|
あ |
NTSC について |
TS1 |
TS1 |
TS1 1 1 1 |
標準1 |
標準1 |
標準1 |
|||||
|
B |
NTSC について |
TS2 2010年11月 |
TS2 2010年11月 |
TS2 2010年11月 |
標準2 |
標準2 |
標準2 |
|||||
|
C |
NTSC について |
TS3 3 3 4 5 6 7 8 9 10 11 12 13 |
TS3 3 3 4 5 6 7 8 9 10 11 12 13 |
TS3 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 30 31 32 33 34 36 37 38 39 49 40 51 42 43 52 53 64 75 86 87 98 99 99 100 100 |
3年生 |
3年生 |
3年生 |
|||||
|
だ |
4年生 |
4年生 |
4年生 |
|||||||||
|
え |
NCSC の |
1 号 |
1 号 |
1 号 |
5年生 |
5年生 |
5年生 |
|||||
|
ふ |
NCSC の |
ER2 ... |
ER2 ... |
ER2 ... |
6年生 |
6年生 |
6年生 |
|||||
|
グ |
NCSC の |
ER3 について |
ER3 について |
ER3 について |
||||||||
|
H |
表3 コンピュータオンリファレンスボード
プレートレイアウトには、6 Std (6 つの標準濃度の標準曲線)、1 NTC (テンプレートなしのコントロール)、1 NCS (ネガティブコントロール溶液)、3 TS (テストサンプル)、3 ERC (抽出回収コントロール) が含まれます。各サンプルに対して 3 つの複製ウェルがあります。
- PCR 機器のセットアップガイドライン
以下の手順は、Thermo ABI 7500 qPCR 機器 (ソフトウェア バージョン 2.0) にのみ適用されます。別の機器を使用する場合は、該当する機器ガイドのセットアップ ガイドラインを参照してください。
1) 新しい実験を生成し、絶対定量化またはユーザー定義のテンプレートを選択します。
2) 「RCL-DNA」という名前の検出プローブを 1 つ作成し、レポーター蛍光体として「FAM」、消光蛍光体として「なし」を選択します。さらに「IC」という名前の検出プローブを 1 つ作成し、レポーター蛍光体として「CY5」、消光蛍光体として「なし」を選択します。参照蛍光は「ROX」です (参照蛍光は機器モデルなどに基づいて決定できます。追加する必要があるかどうかを選択します)。
3) 「サンプル」ペインで、すべてのサンプル情報を順番に追加します。次に、ウェルを選択し、それに応じてターゲットとサンプルを選択します。RCL DNA 標準のタスクを標準に設定し、数量列に 5000000、500000、50000、5000、500、50 (各ウェルの DNA 濃度の単位はコピー/μL) の値を割り当て、ウェルにそれぞれ Std 1、Std 2、Std 3、Std 4、Std 5、Std 6 という名前を付けます。NTC のタスクを NTC に設定します。NCS、TS、および ERC を不明に設定し、上記のプレートレイアウトに従ってそれぞれ名前を付けます。次に、[次へ] をクリックします。
4) 増幅プログラムを設定します。反応容量を 30 μL に設定します。
|
サイクルステップ |
温度(℃) |
時間 |
サイクル |
|
汚染された消化 |
37℃ |
5分 |
1 |
|
初期変性 |
95℃ |
5分 |
1 |
|
変性 |
95℃ |
15秒 |
45 |
|
アニーリング/エクステンション(蛍光収集) |
60℃ |
30秒 |
表4 増幅手順
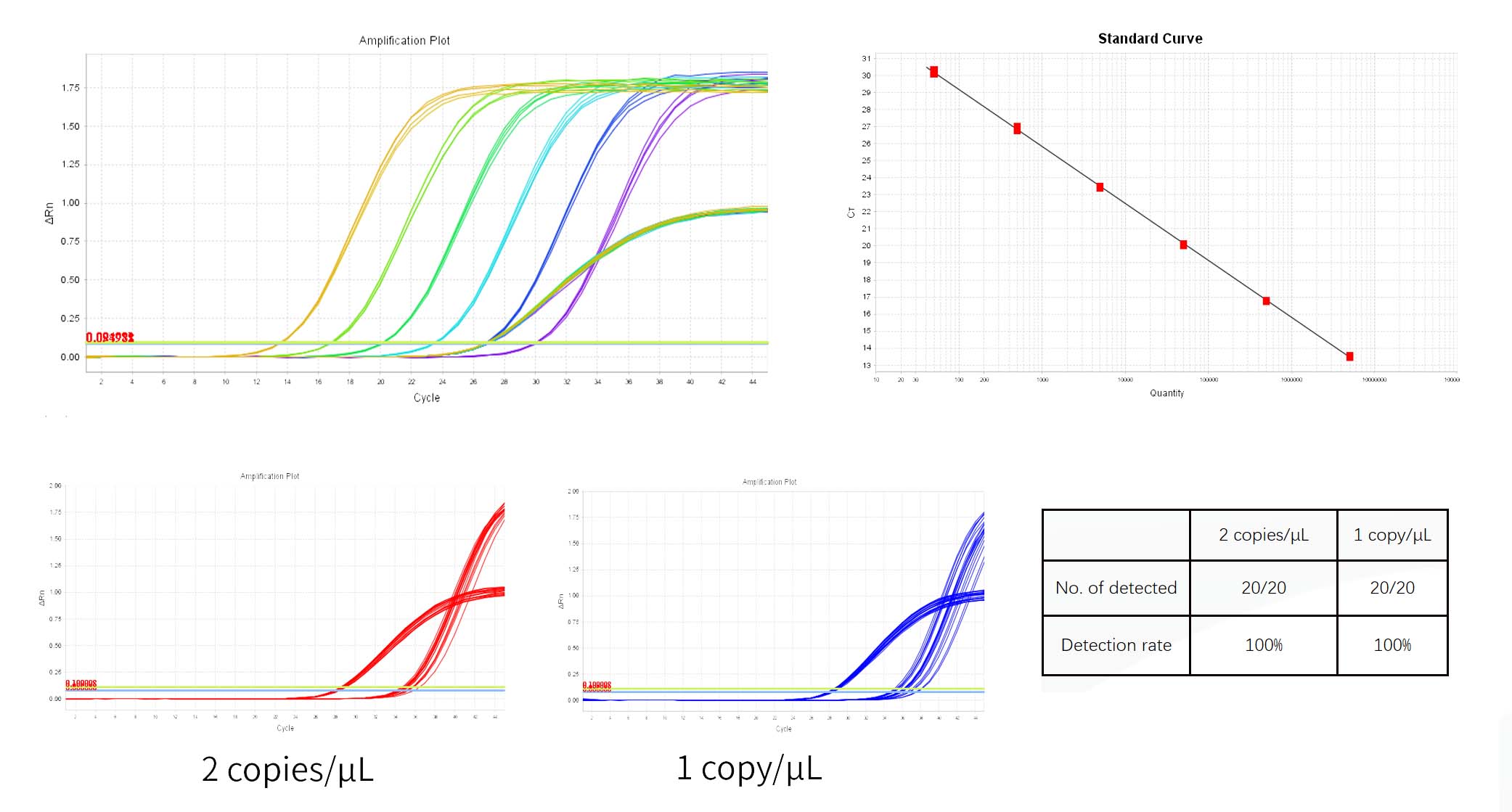
- qPCR結果の分析
1) システムは、分析の増幅プロット パネルでしきい値を自動的に示します。システムによって示されたしきい値は、ベースラインに近すぎる場合があり、その結果、複製ウェル間の Ct に大きな差が生じます。しきい値を適切な位置に手動で調整し、分析をクリックします。その後、マルチコンポーネント プロットで増幅曲線が正常かどうかを最初に確認できます。
2) [結果分析] タブで、標準曲線プロットを確認します。R2、効率、傾き、Y 切片の値を確認します。通常の標準曲線の場合、R²>0.99、90%≤Eff%≤110%、-3.6≤傾き≤-3.1 です。
3) 分析の「ウェル テーブルの表示」ペインでは、各サンプルの濃度が数量で表示され、単位はコピー/μL です。単位はアッセイ レポートで変換できます。
4) 結果分析のパラメータ設定は、使用する特定のモデルとソフトウェアのバージョンに基づいて行う必要があり、通常は機器によって自動的に解釈されます。
5) 測定対象サンプルTSの試験結果とサンプルスパイク回収率ERCに基づいてスパイク回収率を計算します。スパイクの回収率は50%~150%である必要があります。スパイク回収率計の計算式:
回収率(%)={サンプル添加アッセイ(例:コピー数/μL)-サンプルアッセイ(例:コピー数/μL)}×溶出量(μL)/ DNA添加量の理論値(例:コピー数)×100%。
6) 陰性対照NCSのCt値は、標準の最低濃度Ctの平均値よりも大きくなければなりません。
7) テンプレートフリーコントロールNTCは未確定またはCt値≥38である必要があります。
注記
- この製品は研究目的にのみご使用いただけます。
- 安全のため、白衣と使い捨て手袋を着用して作業してください。
3. この試薬を使用する前にこのマニュアルをよく読み、サンプルの取り扱い、反応システムの準備、サンプルの添加など、実験を標準化する必要があります。
4. 使用前に各成分が完全にボルテックスされ、低速で遠心分離されていることを確認します。
支払いとセキュリティ
お支払い情報は安全に処理されます。 クレジットカードの詳細を保存したり、クレジットカード情報にアクセスすることはありません
問い合わせ
あなたも好きかもしれません
よくある質問
この製品は研究目的のみに使用され、人間や動物の治療や診断に使用することを意図したものではありません。製品とコンテンツは、
特定のアプリケーションでは、追加のサードパーティの知的財産権が必要になる場合があります。