Heb je geleerd hoe je een PCR-vrije bibliotheek opbouwt?
Bij high-throughput sequencing vereist conventionele bibliotheekconstructie PCR-amplificatie. Enerzijds is het om DNA-sporenmonsters te versterken en de bibliotheekopbrengst te verhogen. Anderzijds kan het fluorescerende signalen versterken, waardoor het voor sequencers gemakkelijker wordt om fluorescerende signalen vast te leggen en te identificeren om de nauwkeurigheid van de sequentie te verbeteren. PCR is echter als een "tweesnijdend zwaard". Terwijl het het probleem van een laag initieel monstervolume oplost en het fluorescerende signaal versterkt, introduceert het ook amplificatiefouten en -biases en kan het de genoomsequentie "True face" niet perfect presenteren. Bovendien heeft PCR-polymerase een bepaalde amplificatiebias. Sommige regio's, met name hoge GC of lage GC, secundaire structuur en andere regio's, hebben een lage amplificatie-efficiëntie en zijn moeilijk te dekken. Het zal ook een groot aantal verkeerde InDels introduceren en een groot aantal duplicaties met zich meebrengen. , wat leidt tot verspilling van DNA-datavolume en hogere kosten voor sequentiebepaling. Daarom heeft de introductie van PCR-vrije technologie niet alleen de voordelen van PCR, maar overwint het ook de tekortkomingen van PCR. Het kan niet alleen hoogwaardige bibliotheken met hoge gevoeligheid leveren en de detectie van sporenmonsters realiseren, maar ook een hoge nauwkeurigheid hebben en de follow-up Validatie-werklast voor variantstudies verminderen. Dus wat is de PCR-vrije bibliotheek precies en wat zijn de voordelen ervan?
1. Wat is een PCR-vrije bibliotheek?
2. Wat zijn de voordelen van PCR-vrije technologie?
3. Gegevensweergave van PCR-vrij
4. Veelgestelde vragen
5. Productinformatie
6. Over het lezen
1. Wat is een PCR-vrije bibliotheek?
Next-generation sequencingtechnologie is de meest gebruikte technologie in high-throughput sequencingonderzoek in de moderne moleculaire biologie. Traditionele first-generation sequencing kan niet langer volledig voldoen aan de behoeften van onderzoekers. Genoom-resequencing van modelorganismen en genoom-sequencing van niet-modelorganismen vereisen meer kosten. Lage, hogere throughput, snellere sequencingtechnologie, en zo ontstond de second-generation sequencingtechnologie. Het kernprincipe van de second-generation sequencingtechnologie is sequencing-by-synthesis, dat wil zeggen het bepalen van de DNA-sequentie door het vastleggen van de nieuw gesynthetiseerde eind-gelabelde base-informatie. Het hoofdprincipe van second-generation sequencingtechnologie voor DNA-sequencing is om eerst het DNA te fragmenteren, het uiteinde van het gefragmenteerde DNA te repareren en vervolgens specifieke adapters aan beide kanten toe te voegen en vervolgens verschillende methoden te gebruiken om miljoenen ruimtelijk gefixeerde PCR's te genereren. Voor klonale arrays worden sequencinggegevens verkregen met behulp van primerhybridisatie en enzymatische extensiereacties om de fluorescerende labels die door elke extensiereactie zijn opgenomen, in beeld te brengen.
DNA-sequencing door next-generation sequencing omvat hoofdzakelijk twee processen: bibliotheekvoorbereiding en on-machine sequencing. Tijdens de bibliotheekvoorbereiding worden willekeurig onderbroken genomische fragmenten doorgaans versterkt door standaard PCR. Voor sommige speciale templates zijn er echter factoren zoals een complexe secundaire structuur of slechte thermische stabiliteit, die de amplificatievoorkeur van template PCR beïnvloeden, dus niet alle genomische sequenties kunnen op gelijke wijze worden weerspiegeld in de PCR-amplificatiebibliotheek. Vooral voor sommige templates met een hoog GC- of AT-gehalte is het soms moeilijk om de PCR-methode te gebruiken om te amplificeren voor bibliotheekconstructie. Er is geen verschil tussen de PCR-vrije bibliotheek en de conventionele PCR-bibliotheek bij sequencing op de machine, behalve dat PCR niet wordt uitgevoerd tijdens het bibliotheekconstructieproces. De PCR-vrije bibliotheek kan theoretisch de distributie van data-reads verbeteren en een uniformere genoomdekking genereren.De PCR-vrije bibliotheek is een aanvulling op de bibliotheekconstructiemethode. Omdat het direct wordt voorbereid in een onboard-bibliotheek zonder PCR-amplificatie, kan het de dekking van sommige hoge GC- of hoge AT-regio's verbeteren, waardoor de foutbases die door PCR worden geïntroduceerd, databias en sequentieherhaling worden verminderd.
De kernstappen van conventionele NGS-bibliotheekconstructie zijn genoomfragmentatie, eindreparatie, adaptertoevoeging, PCR en signaalamplificatie vóór sequentiebepaling. Dus, hoe zit het met PCR-vrije bibliotheekconstructie? Zoals de naam al doet vermoeden, is dit een bibliotheekbouwproces waarvoor geen PCR nodig is. De kernstappen van bibliotheekconstructie zijn genoomfragmentatie, eindmodificatie, linkertoevoeging en kwaliteitscontrole van de bibliotheek. Maar in feite kan dit alleen als PCR-vrij worden beschouwd in het bibliotheekbouwproces. De echte PCR-vrije zou een proces moeten zijn van bibliotheekconstructie tot sequentiebepaling zonder bibliotheekamplificatie (zoals single-molecule sequencingtechnologie) of een sequentietechnologie zonder accumulatie van PCR-amplificatiefouten (zoals MGI's DNBseq).

Figuur 1. Stroomdiagram van conventionele bibliotheekconstructie en PCR-vrije bibliotheekconstructie
2. Wat zijn de voordelen van PCR-vrije technologie?
In het routinematige bibliotheekconstructieproces kan het amplificatie-enzym fouten introduceren. Door cyclische amplificatie worden deze fouten geaccumuleerd en versterkt, waardoor de betrouwbaarheid van de DNA-sequentiereplicatie afneemt. Bovendien heeft DNA-polymerase ook een bepaalde amplificatiebias, met name voor regio's met grote verschillen in GC-inhoud of secundaire structuren, wat resulteert in een lage amplificatie-efficiëntie en slechte dekkingsuniformiteit; terwijl het verkeerde InDels introduceert, zal het ook een groot aantal duplicaties veroorzaken, wat leidt tot verspilling van DNA-datavolume en hogere sequentiekosten. De PCR-vrije technologie kan de hierboven genoemde problemen die worden geïntroduceerd door PCR-amplificatie in conventionele bibliotheekconstructie goed vermijden. In het proces van bibliotheekconstructie en sequentiebepaling zal een PCR-amplificatieproces een grotere impact hebben op de uniformiteit van de genoomdekking. Voor DNA-sjablonen hebben sommige complexe secundaire structuren en sommige hebben grote verschillen in thermische stabiliteit. Deze factoren zullen de efficiëntie van PCR-amplificatie beïnvloeden. Daarom kan in het PCR-amplificatieproces niet worden gegarandeerd dat alle genomische fragmenten dezelfde amplificatie-efficiëntie kunnen verkrijgen, maar er is een duidelijke amplificatiebias, zoals lage dekking in hoge GC- of lage GC-regio's van het genoom. Er is echter geen PCR in het hele proces van PCR-vrije sequencing. Vergeleken met PCR-bibliotheekconstructie is de dekking van hoge GC-regio's en AT/TA-herhalingsregio's van het genoom verbeterd. De specifieke voordelen zijn als volgt.
2.1 Gestroomlijnd bibliotheekvoorbereidingsproces, tijdsbesparing
Dankzij PCR-vrije bibliotheekvoorbereiding zijn PCR-amplificatie en andere stappen niet meer nodig. Hierdoor wordt het bibliotheekvoorbereidingsproces gestroomlijnd en bespaart u tijd.
2.2 Lagere kosten voor bibliotheekvoorbereiding
In het conventionele bibliotheekvoorbereidingsproces worden dure high-fidelity enzymen gebruikt voor PCR-amplificatie om de authenticiteit van de sequentie te garanderen. Tegelijkertijd elimineert PCR-Free de PCR-amplificatiestap, waardoor de kosten voor bibliotheekvoorbereiding worden verlaagd.
2.3 Verminder de amplificatiebias
DNA-amplificatie-enzymen hebben bepaalde amplificatiebiases, met name voor templates met significante verschillen in GC-inhoud of secundaire structuur. Daarom kan PCR-Free effectief de amplificatiebias vermijden die wordt veroorzaakt door de polymerase.
2.4 Geen PCR-duplicatie
PCR-Free kan dubbele uitlezingen verminderen en de unieke mapping-percentages en het effectieve gegevensgebruik verhogen.
2.5 Verminderde foutmarge
PCR-amplificatie leidt eerder tot replicatiefouten bij insertie en deletie, wat resulteert in een hogere foutmarge bij Indel-sequencing, terwijl PCR-Free het ontstaan en de accumulatie van dergelijke fouten effectief kan voorkomen.
3. Gegevensweergave van PCR-vrij
Hieff NGS™ Ultima Pro PCR-vrije DNA-bibliotheekvoorbereidingskit V2 (Cat# 12196ES) heeft een betere bibliotheekconversieratio dan concurrerende producten.
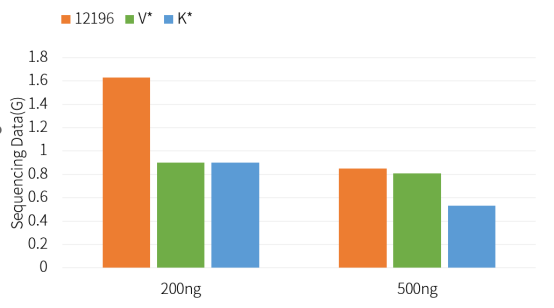
Figuur 2. Hoeveelheid off-machine sequentiegegevens voor PCR-vrije bibliotheekconstructie
4. Veelgestelde vragen
V: Welke soorten monsters zijn geschikt voor PCR-vrije bibliotheken?
A: Voor sommige monsters met complexe secundaire structuren, een hoge GC en PCR-voorkeur kan een PCR-vrije bibliotheek worden gebruikt om te construeren.
V: Wat is de algemene fragmentgrootte van de PCR-vrije bibliotheek?
A: De PCR-vrije bibliotheek zelf heeft geen speciale vereisten voor de fragmentgrootte. De PCR-productbibliotheek, bibliotheek is samengesteld op basis van de grootte van het PCR-product. Voor de genomische bibliotheek wordt aanbevolen om rond de 350 bp te zijn, zodat de kwaliteit van de bibliotheekgegevens relatief goed is.
5. Productinformatie
Het product dat
Tabel 1. Productinformatie
| Product Naam | Cat.nr. | Maat |
| Hieff NGS™ Ultima Pro PCR-vrije DNA-bibliotheekvoorbereidingskit V2 | 12196ES24/96 | 24/96T |
6. Over het lezen
De oplossing voor de NGS-bibliotheekvoorbereiding uit DNA-monsters
Hoeveel weet u over NGS-gerelateerde technologie?
