Beschrijving
De Hieff NGS™ OnePot Pro DNA Library Prep Kit V4 is een enzymatische bibliotheekvoorbereidingskit van de volgende generatie, ontworpen voor gebruik met Illumina en MGI high-throughput sequencing platforms. Vergeleken met traditionele bibliotheekvoorbereidingsmethoden, maakt deze kit gebruik van hoogwaardige fragmentatie-enzymen, waardoor omslachtige sonificatieprocessen overbodig worden. De fragmentatie- en eindreparatiemodules worden gecombineerd in één stap, waardoor zowel de tijd als de kosten van bibliotheekvoorbereiding aanzienlijk worden verminderd. Deze kit is geschikt voor een breed scala aan monstertypen, waaronder planten- en dierengenomen, microbiële genomen en meer, met een monsterinvoerbereik van 1 ng tot 1 μg. De enzymatische fragmentatie zorgt voor uniforme fragmentgroottes over verschillende soorten, met minimale variatie tussen soorten. Bovendien kan deze kit worden gecombineerd met Illumina of MGI adapters en primers voor sequencing op Illumina of MGI high-throughput platforms.
Specificaties
| Cat.Nr. | 12972ES08 / 12972ES24 /12972ES96 |
| Maat | 8 D / 24 D/96 T |
Componenten
| Componenten nr. | Naam | 12972ES08 | 12972ES24 | 12972ES96 |
| 12972-A | 80 μL | 240 μL | 960 μL | |
| 12972-B | Smearase™ Enzym 4.0 | 80 μL | 240 μL | 960 μL |
| 12972-C | Ligatieversterker 4.0 | 240 μL | 720 μL | 3×960 μL |
| 12972-D | Snel DNA-ligase 4.0 | 80 μL | 240 μL | 2×480 μL |
| 12972-E | 2×Ultima HF-versterkingsmix | 200 μL | 600 μL | 3×800 μL |
Opslag
Dit product moet worden bewaard bij -25~-15℃ gedurende 1 jaar.
Notities
1. Over de werking
1. Werk met labjassen en wegwerphandschoenen,voor uw veiligheid.
2. Laat de componenten ontdooien op kamertemperatuur. Na ontdooien, meng grondig door te vortexen, draai de buis kort rond en zet ze op ijs voor later gebruik.
3. Bij het voorbereiden van de reactieoplossing van elke stap, wordt aanbevolen om een pipet te gebruiken om goed te mengen of voorzichtig te schudden. Krachtig schudden kan leiden tot een afname van de bibliotheekoutput.
4. Het wordt sterk aanbevolen om gefilterde pipetpunten te gebruiken om kruisbesmetting te voorkomen. Zorg ervoor dat u pipetpunten vervangt bij het verwerken van verschillende monsters.
5. Onjuiste handelingen kunnen zeer waarschijnlijk aerosolverontreinigingen veroorzaken, wat de nauwkeurigheid van het resultaat beïnvloedt. Verplichte fysieke isolatie van PCR-reactiemenggebieden en PCR-productzuiveringsassaygebieden wordt aanbevolen. Uitgerust met apparatuur zoals gespecialiseerde pipetten voor bibliotheekconstructie.Voer routinematig schoonmaken uit voor elk gebied door de oppervlakken af te vegen met 0,5% natriumhypochloriet of 10% bleekmiddel
6. Dit product is uitsluitend bedoeld voor onderzoeksdoeleinden.
2. DNA-fragmentatie1. De kit is compatibel met 100 pg - 1000 ng input-DNA. Het wordt sterk aanbevolen om input-DNA van hoge kwaliteit te gebruiken met A260/A280 = 1,8-2,0.
2. De volgende experimenten kunnen worden beïnvloed als hoge concentraties zouten zoals het metaalchelerende middel worden geïntroduceerd met het invoer-DNA. We raden aan om het DNA-monster te elueren in ddH2O voor fragmentatie.
3. Zie tabel 6 voor de fragmentatietijd van standaard DNA-monsters.De kit heeft een lage fragmentatiebias en biedt een uniforme GC-dekking voor DNA-monsters met een breed scala aan GC-samenstellingen. Pas de fragmentatietijd aan op basis van uw experimentele vereisten.
4. Voor een nauwkeurige fragmentatie bereidt u de reactie voor op ijs.
3. Adapterligatie
1. Klanten kunnen kiezen uit Illumina of MGI Long Adapter (Barcoded Adapter) kits en korte Adapter kits, afhankelijk van hun experimentele vereisten.
2. Het werd aanbevolen om hoogwaardige, commerciële adapters te selecteren. Als u zelfgemaakte adapters selecteert, vertrouw dan een bedrijf toe met ervaring in NGS-primersynthese en merk op dat er strikte contaminatiecontrole nodig is. Daarnaast wordt aanbevolen om DNA-annealingoplossing te bereiden in een schone werkbank en slechts één type adapter per keer te gebruiken om kruisbesmetting te voorkomen.
3. Laat de adapters ontdooien op ijs of bij 4°C. Bij gebruik op kamertemperatuur mag de laboratoriumtemperatuur niet hoger zijn dan 25°C om te voorkomen dat de adapters denatureren.
4. De kwaliteit en concentratie van de adapters hebben direct invloed op de ligatie-efficiëntie en de opbrengst van de bibliotheek. Een te hoge concentratie adapters bevordert de vorming van adapterdimeren, terwijl een te lage adapter de ligatiesnelheid en de opbrengst van de bibliotheek vermindert. Corresponderende verdunningen met TE-buffer volgens de hoeveelheid invoer-DNA bij gebruik van adapter. Tabel 2 -3 geeft de aanbevolen adapterverdunningsmethoden weer voor verschillende invoer-DNA-hoeveelheden met behulp van deze kit.
Tabel 1 De aanbevolen Illumina-adapterhoeveelheid voor verschillende invoer-DNA
| DNA invoeren | Adapterverdunning (volume van adapter: totaal volume) | Concentratie |
| 1 ng | 30-Vouw (1 : 30) | 0,5 μM |
| 10 ng | 7.5-Vouw (1 : 7.5) | 2 μM |
| 100 gram | 3-Vouw (1 : 3) | 5 μM |
| 1000 gram | 1.5-Vouw (1 : 1.5) | 10 μM |
Tabel 2 De aanbevolen MGI-adapterhoeveelheid voor verschillende invoer-DNA
| DNA invoeren | Adapterverdunning (volume van adapter: totaal volume) | Concentratie |
| 1 ng | 20-Vouw (1 : 20) | 0,5 μM |
| 10 ng | 5-Vouw (1 : 5) | 2 μM |
| 100 gram | 2-Vouw (1 : 2) | 5 μM |
| 1000 gram | onverdund | 10 μM |
4. Op kralen gebaseerde DNA-opruiming en grootteselectie
1. De stap voor selectie van de DNA-fragmentgrootte kan worden uitgevoerd na adapterligatie of na bibliotheekamplificatie.
2. Het wordt aanbevolen om de grootteselectie direct na de adapterligatie uit te voeren als de hoeveelheid DNA-invoer meer dan 50 ng bedraagt; Anders dient u de grootteselectie pas na de versterking uit te voeren.
3. De Ligation Enhancer bevat een hoge concentratie PEG, wat een significante impact kan hebben op de nauwkeurige maatselectie. Als maatselectie dus direct na adapterligatie moet worden uitgevoerd, wordt het sterk aanbevolen om een stap voor het opruimen van kralen toe te voegen vóór de maatselectie. De stap voor maatselectie kan direct worden uitgevoerd als deze wordt uitgevoerd vóór de eindreparatie/dA-tailing of na de bibliotheekversterking.
4. De magnetische kralen moeten voor gebruik op kamertemperatuur worden gebracht, anders neemt de opbrengst af en wordt het effect van de grootteselectie beïnvloed.
5. De magnetische kralen moeten vóór gebruik goed worden gemengd door middel van vortexen of pipetteren.
6. Zuig de kralen niet op bij het overbrengen van de supernatant, zelfs sporen van de kralen kunnen de volgende reacties beïnvloeden.
7. De 80% ethanol moet vers worden bereid, anders heeft dit invloed op de winningsefficiëntie.
8. Voor een nauwkeurige grootteselectie wordt aanbevolen om te beginnen met een volume van meer dan 100 μL. Indien minder, wordt aanbevolen om het volume op te voeren tot 100 μL met ultrazuiver water.
9. De magnetische kralen moeten bij kamertemperatuur worden gedroogd voordat het product wordt geëlueerd. Onvoldoende droogte zal er gemakkelijk voor zorgen dat ethanolresten de daaropvolgende reacties beïnvloeden; overmatige droogte zal ervoor zorgen dat de magnetische kralen barsten en de zuiveringsopbrengst verminderen. Normaal gesproken is drogen bij kamertemperatuur gedurende 3-5 minuten voldoende om de kralen volledig te laten drogen.
10. Indien nodig worden de gezuiverde of op grootte geselecteerde DNA-monsters geëlueerd in 0,1× TE-buffer kan 1-2 weken bewaard worden bij 4°C of een maand bij -20°C.
5. Bibliotheekversterking
1. Of er wel of geen bibliotheekamplificatie moet worden uitgevoerd, hangt af van de hoeveelheid DNA-invoer, typen adapters, de sequentiedatatoepassingen, enz. De amplificatiestap is vereist als er gedeeltelijke adapters worden gebruikt. Bij gebruik van adapters van volledige lengte wordt aanbevolen om amplificatie uit te voeren als het invoer-DNA < 200 ng is; anders is amplificatie niet nodig.
2. Amplificatiecyclusnummers moeten strikt worden gecontroleerd. Onvoldoende amplificatie kan leiden tot een lage bibliotheekopbrengst; Overamplificatie kan leiden tot verhoogde bias, fouten, gedupliceerde read en chimere producten. Tabel 3 geeft aanbevolen cyclusnummers weer die gericht zijn op een bibliotheekopbrengst van 1 μg.
Tabel 3 Het aanbevolen aantal cycli om te genereren 1.000 ng bibliotheekopbrengst
| DNA invoeren | Aantal cycli dat nodig is om 1 μg bibliotheekopbrengst te genereren |
| 1000 gram | 2 - 4 |
| 500 gram | 2 - 4 |
| 250 gram | 4 - 6 |
| 100 gram | 5 - 7 |
| 50 gram | 7 - 9 |
| 1 ng | 12 - 14 |
Opmerking:
1. Tabel 3 toont het aantal lusparameters met behulp van hoogwaardige invoer-DNA-testen van ongeveer 200 bp. De FFPE-DNA-kwaliteit varieert sterk en wanneer de DNA-kwaliteit slecht is of de bibliotheeklengte groot is, moet het aantal cycli dienovereenkomstig worden verhoogd om voldoende bibliotheken te verkrijgen.
2.IAls de grootte moet worden geselecteerd tijdens het bibliotheekopbouwproces, wordt een hoger cyclusnummer voor bibliotheekversterking aanbevolen. Anders wordt een lager cyclusnummer aanbevolen.
3.IIndien onvolledige adapters worden gebruikt, dienen er minimaal 2 cycli te worden versterkt om een complete adapter te vormen.
6. Analyse van de bibliotheekkwaliteit
1. De kwaliteit van de geconstrueerde bibliotheken wordt over het algemeen geanalyseerd door de concentraties en grootteverdelingen te meten.
2. De concentraties in bibliotheken kunnen worden gemeten met behulp van op fluorescentie gebaseerde methoden zoals Qubit en PicoGreen of qPCR.
3. Het wordt NIET aanbevolen om op absorptie gebaseerde kwantificeringsmethoden zoals NanoDrop te gebruiken.
4. Het wordt aanbevolen om de qPCR-methode te gebruiken voor bibliotheekkwantificering: fluorescentiegebaseerde methoden zoals Qubit en PicoGreen kunnen de incomplete dsDNA-structuren (inserts zonder adapter of met slechts één van de uiteinden geligeerd met adapter) niet onderscheiden van de complete bibliotheken. De qPCR-methode zal alleen de complete bibliotheken met beide uiteinden geligeerd met adapters (de sequentiële bibliotheken) amplificeren en meten, waardoor een nauwkeurigere meting voor het laden wordt geboden.
5. De grootteverdeling van bibliotheken kan worden geanalyseerd met behulp van Agilent Bioanalyzer of andere apparaten die gebaseerd zijn op de principes van capillaire elektroforese of microfluïdica.
7. Andere materialen
1. DNA-zuiveringsmagnetische kralen: Hieff NGSTM DNA-selectiekralen (
2. Adapters: Complete adapter voor Illumina:
3. Kwaliteitsanalyse van de bibliotheek: Agilent 2100 Bioanalyzer DNA 1000 Chip/High Sensitivity Chip of andere gelijkwaardige producten; kwantitatieve reagentia voor de bibliotheek.
4. Overige materialen: absolute ethanol, steriel ultrapuur water, pipetpunten met lage retentie, PCR-buis, magnetische standaards, thermocycler, enz.
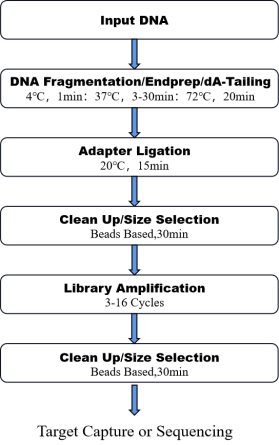
8. Werkstroom
Figuur 1.De workflow van Eenpot Pro DNA Bibliotheekvoorbereidingskit
Betaling en beveiliging
Uw betalingsinformatie wordt veilig verwerkt. We slaan geen creditcardgegevens op en hebben geen toegang tot uw creditcardinformatie.
Navraag
Misschien vind je het ook leuk
FAQ
Het product is alleen bedoeld voor onderzoeksdoeleinden en is niet bedoeld voor therapeutisch of diagnostisch gebruik bij mensen of dieren. Producten en inhoud worden beschermd door patenten, handelsmerken en auteursrechten die eigendom zijn van
Voor bepaalde toepassingen zijn mogelijk aanvullende intellectuele eigendomsrechten van derden vereist.