Beschrijving
CHO Host Cell DNA Residue Detection Kit wordt gebruikt voor de kwantitatieve analyse van CHO DNA-residuen van de gastheercel in tussenliggende monsters, halffabricaten en eindproducten van verschillende biologische producten.
Deze kit maakt gebruik van de Taqman-fluorescentiesonde en de polymerasekettingreactie (PCR)-methode, die een minimale detectielimiet van het fg-niveau heeft en specifiek en snel de resterende CHO kan detecteren cel-DNA. De kit moet samen met de Residual DNA Sample Preparation Kit (Cat# 18461ES) worden gebruikt.
Product Componenten
| Nee. | Naam | 41332ES50 (50T) | 41332ES60 (100T) |
| 41332-A | CHO qPCR Mengen | 0,75 ml | 1.5 ml |
| 41332-B | CHO Primer&Sonde Mengen | 250 μL | 500 μL |
| 41332-C | DNA-verdunning Buffer | 2×1.8 ml | 4×1.8 ml |
| 41332-D | CHO DNA-controle (30 ng/μL) | 25 μL | 50 μL |
Verzending en opslag
1. Verzonden op droogijs en bewaard bij -20°C voor 2 jaar
Dit product moet gedurende 2 jaar bij een temperatuur van -25~-15℃ bewaard worden.
Zowel 41332-A als 41332-B moeten op een lichtbeschermde plaats worden bewaard.
Toepasbare instrumentmodellen
Omvatten onder meer:
Bio-Rad: CFX96 optische module.
Thermowetenschappelijk: ABI 7500; ABI Quant Studio 5.
Instructies
- CHO DNA-standaardverdunning en standaardcurvevoorbereiding
De CHO DNA-controle werd gradiëntverdund met behulp van de DNA-verdunningsbuffer die in de kit werd meegeleverd*, en de verdunning
concentratie is 3 ng/μL, 300 pg/μL, 30 pg/μL, 3 pg/μL, 300 fg/μL, 30 fg/μL, 3 fg/μL.
Zie onderstaande gedetailleerde instructies:
- Ontdooi de CHO DNA-controle en DNA-verdunningsbuffer op ijs. Na volledig ontdooien, zachtjes vortexen om te mengen en centrifugeren op lage snelheid gedurende 10 seconden.
- Neem zeven schone 1,5 ml-buisjes, gemarkeerd met Std0, Std1, Std2, Std3, Std4, Std5, Std6.
- Voeg 90 μL DNA-verdunningsbuffer en 10 μL CHODNA-controle toe aan de 1,5 ml microfugebuis met het label Std0, namelijk verdun tot 3 ng/μL. Meng en centrifugeer vervolgens gedurende 10 seconden. Verpak de verdunde DNA-standaard in subverpakkingen en deze kan op korte termijn (niet langer dan 3 maanden) worden bewaard bij -25~-15℃**Vermijd herhaaldelijk invriezen en ontdooien.
- Voeg 90 μL DNA-verdunningsbuffer toe aan andere buizen***, volg dan de onderstaande procedure voor de seriële verdunningen****.
| Buis | Verdunningsverhouding | Standaardconcentratie |
| Standaard 1 | 10 μL Std0 + 90 μL DNA-verdunningsbuffer | 300 pg/μL |
| Standaard 2 | 10 μL Std1 + 90 μL DNA-verdunningsbuffer | 30 pg/μL |
| Std3 | 10 μL Std2 + 90 μL DNA-verdunningsbuffer | 3 pg/μL |
| Standaard 4 | 10 μL Std3 + 90 μL DNA-verdunningsbuffer | 300 fg/μL |
| Standaard 5 | 10 μL Std4 + 90 μL DNA-verdunningsbuffer | 30 fg/μL |
| Std6 | 10 μL Standaard 5 + 90 μL DNA-verdunningsbuffer | 3 fg/μL |
Tabel 1 Standaardgradiëntverdunning
*Voor elke concentratie zijn drie replicaatputjes nodig. Het detectiebereik is 3 fg/μL~300pg/μL, maar dit bereik kan worden uitgebreid.
**Om het aantal keren dat het DNA opnieuw moet worden ingevroren en ontdooid te beperken en besmetting te voorkomen, wordt aanbevolen om de DNA-controle de eerste keer in alikwoten bij -25~-15℃ te bewaren.
***Eenmaal ontdooid kan de DNA-verdunningsbuffer gedurende 7 dagen bij 2-8°C worden bewaard. Indien u het langere tijd niet gebruikt, bewaar het dan bij een temperatuur van -25~-15℃.
****Zorg ervoor dat het sjabloon volledig gemengd is. Schud het mengsel voorzichtig gedurende 15 seconden tot 1 minuut voor elke gradiëntverdunning.
- Extraction Recovery Control (ERC) voorbereiding
Stel de concentratie van CHO DNA in ERC in zoals nodig (het ERC-monster werd bijvoorbeeld bereid met 30 pg CHO DNA), als volgt:
- Voeg 100 μL testmonster toe aan een schone 1,5 ml-buis, voeg vervolgens 10 μL 3pg/μL CHO DNA-standaard (Std3) toe en meng goed, gemarkeerd als ERC.
- Voer de DNA-extractie van het ERC-monster uit samen met de testmonsters om het gezuiverde ERC-monster te bereiden.
- Voorbereiding van negatieve controleoplossing (NCS)
Stel de negatieve controle in het experiment in; de specifieke bewerkingsstappen zijn als volgt:
1) Voeg 100 μL monstermatrix (of DNA-verdunningsbuffer) toe aan een schone 1,5 ml-buis en markeer deze als NCS.
2) Voer de DNA-extractie van het NCS-monster uit samen met de testmonsters om het gezuiverde NCS-monster voor te bereiden.
- Geen Template Control (NTC) voorbereiding
Stel de 'geen sjabloon'-controle in het experiment in; de specifieke bewerkingsstappen zijn als volgt:
1) NTC vereist geen voorbehandeling van het monster en kan worden geconfigureerd in de fase van qPCR-detectie van resterend DNA.
2) Het NTC-monster in elke buis of put is 20 μL Mix (d.w.z. 15 μL CHO qPCR Mix + 4 μL CHO Primer&probe Mix + 1 μL IC) + 10 μL DNA Dilution Buffer. Het wordt aanbevolen om drie replicaatputten te configureren.
- PCR-reactiesysteem
| Onderdeel | Volume (μL) |
| CHO qPCR-mix* | 15 |
| CHO Primer&Probe-mix | 5 |
| DNA-sjabloon | 10 |
| Totaal volume** | 30 |
Tabel2 Reactiesysteem
*Bereken het totale PCR-reactievolume door het aantal reacties: qPCR-mix = (het aantal reacties + 2) × (15 + 4 + 1) μL (inclusief de verliezen van twee reactieputjes). In het experiment worden meer dan drie replicaties voor elk monster aanbevolen.
**Na het afsluiten van de buis of het verzegelen van de plaat, centrifugeer de reactiebuis of plaat op lage snelheid gedurende 10 seconden. Na voldoende schudden en mengen gedurende 5 seconden, herhaal de centrifuge om de vloeistof van het deksel of de wand naar de bodem te verzamelen. Vermijd bellen tijdens de werking.
Zie onderstaande tabel voor de aanbevolen plaatopstelling:
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | NTC |
| TS-1 | TS-1 | TS-1 |
| Standaard 1 | Standaard 1 | Standaard 1 |
|
|
|
| B | NTC |
| TS2 | TS2 | TS2 |
| Standaard 2 | Standaard 2 | Standaard 2 |
|
|
|
| C | NTC |
| TS-3 | TS-3 | TS-3 |
| Standaard 3 | Standaard 3 | Standaard 3 |
|
|
|
| D |
|
|
|
|
|
| Standaard 4 | Standaard 4 | Standaard 4 |
|
|
|
| Ik | NCS |
| ERC-1 | ERC-1 | ERC-1 |
| Standaard 5 | Standaard 5 | Standaard 5 |
|
|
|
| F | NCS |
| ERC2 | ERC2 | ERC2 |
| Standaard 6 | Standaard 6 | Standaard 6 |
|
|
|
| G | NCS |
| ERC-3 | ERC-3 | ERC-3 |
|
|
|
|
|
|
|
| H |
|
|
|
|
|
|
|
|
|
|
|
|
Tabel 3 Computer-on referentiebord
De plaatindeling omvat: 6 Std (de standaardcurve van 6 standaardconcentraties), 1 NTC (geen templatecontrole), 1 NCS (negatieve controleoplossing), 3 TS (testmonsters), 3 ERC (extractieherstelcontrole).Drie replicaatputjes voor elk monster.
- Instellen richtlijnen voor een PCR-instrument(2-stapsmethode) (bijv. Thermo ABI 7500 qPCR-instrument, softwareversie 2.0)
De volgende instructies zijn alleen van toepassing op het Thermo ABI 7500 qPCR-instrument (softwareversie 2.0). Als u een ander instrument gebruikt, raadpleeg dan de handleiding van het desbetreffende instrument voor installatierichtlijnen.
1) Genereer een nieuw experiment en kies de sjabloon voor absolute kwantificering of een door de gebruiker gedefinieerde sjabloon.
2) Maak 1 detectiesonde, genaamd "CHO-DNA", selecteer reporterfluorofoor als "FAM" en quenchfluorofoor als "Geen"; maak nog 1 detectiesonde, noem "IC" en selecteer de reporterfluorofoor als "CY5" en de quenchingfluorofoor als "Geen". De referentiefluorescentie is ROX" (de referentiefluorescentie kan gebaseerd zijn op het instrumentmodel, enz., selecteer of u deze moet toevoegen).
3) Voeg in het paneel 'Samples' alle informatie over de samples op hun beurt toe. Selecteer vervolgens de wells, kies het doel en de samples dienovereenkomstig. Stel de taak van CHO in DNA-standaard als standaard, en wijs de waarden 300000, 30000, 3000, 300, 30, 3 toe (de eenheid van DNA-concentratie in elke put is fg/μL) in de kolom Hoeveelheid en noem de putjes Standaard 1, Standaard 2, Standaard 3, Standaard 4, Standaard 5, Std 6, overeenkomstig. Stel de taak van NTC in als NTC. Stel de NCS, TS en ERC in als Onbekend, en noemde ze overeenkomstig de bovenstaande Plaatindeling. Klik vervolgens op Volgende.
4) Stel het amplificatieprogramma in: stel het reactievolume in op 30 μL.
| Cyclus Stap | Temperatuur (℃) | Tijd | Cycli |
| Initiële denaturatie | 95℃ | 5 minuten | 1 |
| Denaturatie | 95℃ | 15 seconden | 40 |
| Gloeien/verlenging (fluorescentieverzameling) | 60℃ | 30 seconden |
Tabel 4 Amplificatieprocedure
- Analyse van qPCR-resultaten
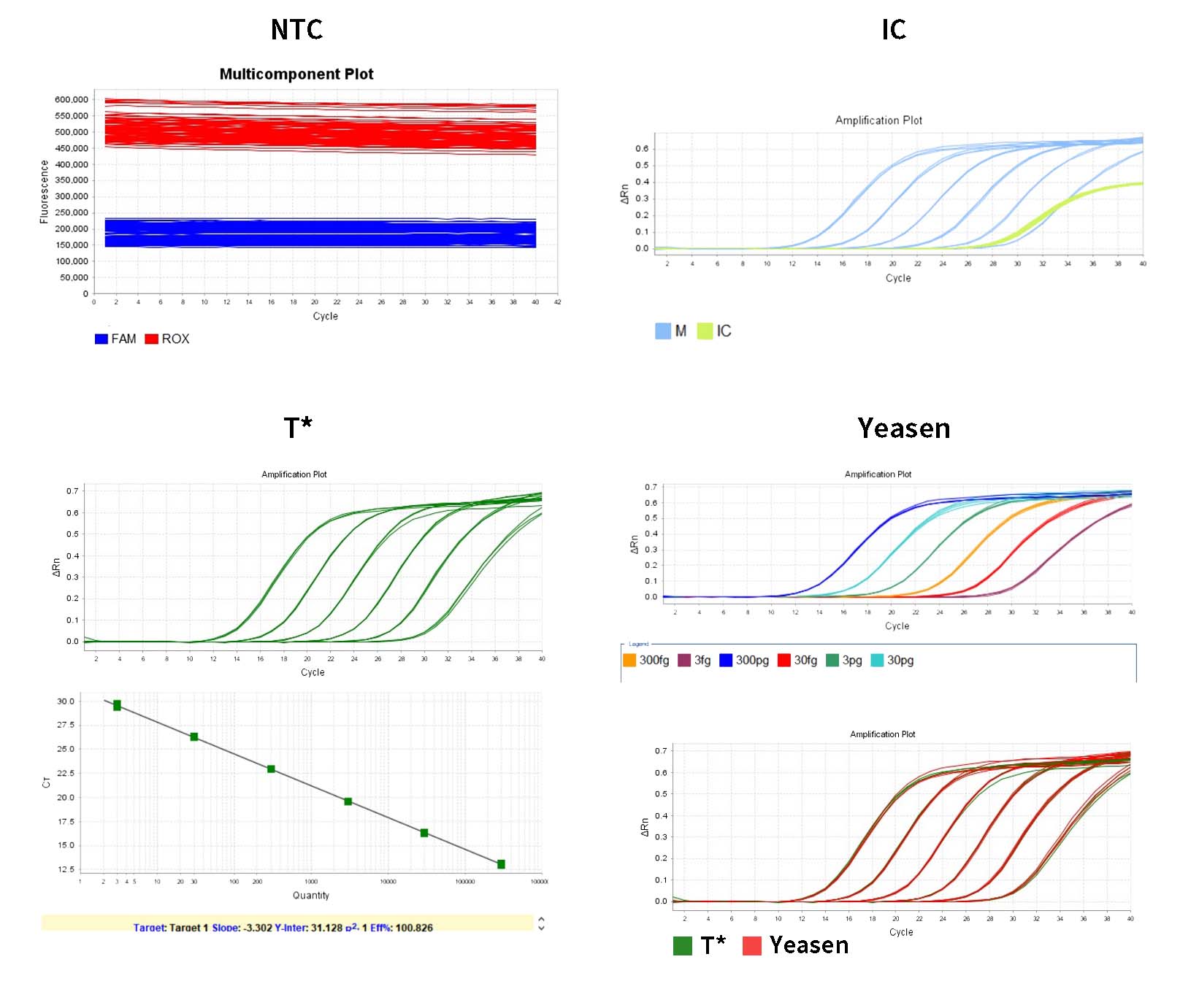
1) Het systeem geeft automatisch de Threshold in het Amplification Plot-paneel van Analysis. De Threshold die door het systeem wordt gegeven, ligt soms te dicht bij de basislijn, wat resulteert in een groot verschil in Ct tussen replicatenputten. U kunt de Threshold handmatig aanpassen naar een geschikte positie en op Analyze klikken. Vervolgens kunt u eerst controleren of de amplificatiecurve normaal is in Multicomponent Plot.
2) Bekijk in het tabblad Resultaatanalyse de Standard Curve-plot. Controleer de waarden voor de R2, Efficiëntie, Helling en Y-intercept. Voor een normale standaardcurve, R²>0,99, 90%≤Eff%≤110%, -3,6≤Helling≤-3,1.
3) In het deelvenster 'Bekijk puttentabel' in Analyse worden de concentraties van elk monster weergegeven in Hoeveelheid. De eenheid is fg/μL. De eenheden kunnen in het analyserapport worden omgezet.
4) De parameterinstellingen van de resultaatanalyse moeten gebaseerd zijn op het specifieke model en de gebruikte softwareversie en kunnen doorgaans automatisch door het instrument worden geïnterpreteerd.
5) Bereken de spike recovery rate op basis van de testresultaten van de te meten monster TS en de sample spike recovery ERC. De recovery rate van spikes moet tussen 50%~150% liggen. Formule voor spiked recovery rate meter: Recovery (%) = {Sample spiked assay (eg.pg/μL) - Sample assay (eg.pg/μL)} x Elutievolume (μL) / Theoretische waarde van de hoeveelheid DNA-toevoeging (bijv. pg) x 100%。
6) De Ct-waarde van de negatieve controle NCS moet groter zijn dan het gemiddelde van de laagste concentratie Ct van de standaard.
- Template vrije controle NTC moet Onbepaald zijn of Ct-waarde ≥3
Notities
- Dit product is uitsluitend bedoeld voor onderzoeksdoeleinden.
- Voor uw eigen veiligheid draagt u een labjas en wegwerphandschoenen.
3. Lees deze handleiding zorgvuldig door voordat u dit reagens gebruikt. Het experiment moet gestandaardiseerd zijn, inclusief de monsterbehandeling, de voorbereiding van het reactiesysteem en de toevoeging van het monster.
4. Zorg ervoor dat elk onderdeel volledig wordt gevortexed en op lage snelheid wordt gecentrifugeerd voordat u het gebruikt.
Betaling en beveiliging
Uw betalingsinformatie wordt veilig verwerkt. We slaan geen creditcardgegevens op en hebben geen toegang tot uw creditcardinformatie.
Navraag
Misschien vind je het ook leuk
FAQ
Het product is alleen bedoeld voor onderzoeksdoeleinden en is niet bedoeld voor therapeutisch of diagnostisch gebruik bij mensen of dieren. Producten en inhoud worden beschermd door patenten, handelsmerken en auteursrechten die eigendom zijn van
Voor bepaalde toepassingen zijn mogelijk aanvullende intellectuele eigendomsrechten van derden vereist.