Har du lärt dig hur man bygger ett PCR-fritt bibliotek?
Vid sekvensering med hög genomströmning kräver konventionell bibliotekskonstruktion PCR-amplifiering. Å ena sidan är det att amplifiera spår-DNA-prover och ökar biblioteksutbytet. Å andra sidan kan den förstärka fluorescerande signaler, vilket gör det lättare för sekvenserare att fånga och identifiera fluorescerande signaler för att förbättra sekvenseringsnoggrannheten. PCR är dock som ett "tveeggat svärd". Samtidigt som den löser problemet med låg initial provvolym och förstärker den fluorescerande signalen, introducerar den också amplifieringsfel och fördomar och kan inte perfekt presentera genomsekvensen "True face". Dessutom har PCR-polymeras en viss amplifieringsbias. Vissa regioner, särskilt hög GC eller låg GC, sekundär struktur och andra regioner, har låg amplifieringseffektivitet och är svåra att täcka. Det kommer också att introducera ett stort antal felaktiga InDels och ge ett stort antal dupliceringar. , vilket orsakar ett slöseri med DNA-datavolym och ökar kostnaden för sekvensering. Därför har införandet av PCR-fri teknologi inte bara fördelarna med PCR utan övervinner också bristerna med PCR. Det kan inte bara ge högkvalitativa bibliotek med hög känslighet och realisera detektering av spårprover, utan också ha hög noggrannhet och minska uppföljningsarbetet för validering av variantstudier. Så vad exakt är det PCR-fria biblioteket och vilka är dess fördelar?
1. Vad är ett PCR-fritt bibliotek?
2. Vilka är fördelarna med PCR-fri teknologi?
3. Datavisning av PCR-Free
4. Vanliga frågor
5. Produktinformation
6. Angående läsning
1. Vad är ett PCR-fritt bibliotek?
Nästa generations sekvenseringsteknologi är den mest använda tekniken inom högkapacitets sekvenseringsforskning inom modern molekylärbiologi. Traditionell första generationens sekvensering kan inte längre fullt ut tillgodose forskarnas behov. Genomresekvensering av modellorganismer och genomsekvensering av icke-modellorganismer kräver mer kostnad. Låg, högre genomströmning, snabbare sekvenseringsteknik, vilket ger upphov till andra generationens sekvenseringsteknik. Kärnprincipen för den andra generationens sekvenseringsteknologi är sekvensering-för-syntes, det vill säga att bestämma DNA-sekvensen genom att fånga den nyligen syntetiserade ändmärkta basinformationen. Huvudprincipen för andra generationens sekvenseringsteknologi för DNA-sekvensering är att först fragmentera DNA:t, reparera änden av det fragmenterade DNA:t och sedan lägga till specifika adaptrar på båda sidor och sedan använda olika metoder för att generera miljontals rumsligt fixerade PCR. För klonala arrayer erhålls sekvenseringsdata med användning av primerhybridisering och enzymatiska förlängningsreaktioner för att avbilda de fluorescerande märkningarna som ingår i varje förlängningsreaktion.
DNA-sekvensering genom nästa generations sekvensering innefattar huvudsakligen två processer: biblioteksberedning och sekvensering på maskin. Under biblioteksberedningen amplifieras slumpmässigt avbrutna genomiska fragment typiskt genom standard PCR. Men för vissa speciella mallar finns det faktorer som komplex sekundär struktur eller dålig termisk stabilitet, som påverkar amplifieringspreferensen för mall-PCR, så inte alla genomiska sekvenser kan återspeglas lika i PCR-amplifieringsbiblioteket. Speciellt för vissa mallar med hög GC eller hög AT-halt är det ibland svårt att använda PCR-metoden för att amplifiera för bibliotekskonstruktion. Det finns ingen skillnad mellan det PCR-fria biblioteket och det konventionella PCR-biblioteket vid sekvensering på maskinen, förutom att PCR inte utförs under bibliotekskonstruktionsprocessen. Det PCR-fria biblioteket kan teoretiskt förbättra distributionen av dataavläsningar och generera mer enhetlig genomtäckning.Det PCR-fria biblioteket är ett komplement till bibliotekskonstruktionsmetoden. Eftersom det förbereds direkt i ett ombordbibliotek utan PCR-amplifiering, kan det förbättra täckningen av vissa regioner med hög GC eller hög AT, och därigenom minska de felbaser som introduceras av PCR, databias och sekvensupprepning.
Kärnstegen i konventionell NGS-bibliotekskonstruktion är genomfragmentering, ändreparation, adaptertillägg, PCR och signalförstärkning före sekvensering. Så, hur är det med PCR-fri bibliotekskonstruktion? Som namnet antyder är det en biblioteksbyggande process som inte kräver PCR. Kärnstegen för bibliotekskonstruktion är genomfragmentering, slutmodifiering, länkaddition och bibliotekskvalitetskontroll. Men i själva verket kan detta bara betraktas som PCR-fritt i bibliotekets byggprocess. Den verkliga PCR-fria bör vara en process från bibliotekskonstruktion till sekvensering utan biblioteksamplifiering (som enmolekyls sekvenseringsteknologi) eller en sekvenseringsteknik utan ackumulering av PCR-amplifieringsfel (som MGI:s DNBseq).

Figur 1. Flödesschema över konventionell bibliotekskonstruktion och PCR-fri bibliotekskonstruktion
2. Vilka är fördelarna med PCR-fri teknologi?
I den rutinmässiga bibliotekskonstruktionsprocessen kan amplifieringsenzymet introducera fel. Genom cyklisk amplifiering kommer dessa fel att ackumuleras och amplifieras, vilket minskar tillförlitligheten för DNA-sekvensreplikation. Dessutom har DNA-polymeras också en viss amplifieringsbias, speciellt för regioner med stora skillnader i GC-innehåll eller sekundära strukturer, vilket resulterar i låg amplifieringseffektivitet och dålig täckningslikformighet; samtidigt som felaktiga InDels introduceras, kommer det också att medföra ett stort antal Duplicering orsakar slöseri med DNA-datavolym och ökar sekvenseringskostnaderna. Den PCR-fria teknologin kan mycket väl undvika de ovan nämnda problemen som introduceras av PCR-amplifiering i konventionell bibliotekskonstruktion. I processen för bibliotekskonstruktion och sekvensering, om det finns en PCR-amplifieringsprocess, kommer det att ha en större inverkan på enhetligheten i genomtäckningen. För DNA-mallar har vissa komplexa sekundära strukturer och vissa har stora skillnader i termisk stabilitet. Dessa faktorer kommer att påverka effektiviteten av PCR-amplifiering. I PCR-amplifieringsprocessen kan det därför inte garanteras att alla genomiska fragment kan erhålla samma amplifieringseffektivitet, men det finns en uppenbar amplifieringsbias, såsom låg täckning i områden med hög GC eller låg GC i genomet. Det finns dock ingen PCR i hela processen med PCR-fri sekvensering. Jämfört med PCR-bibliotekskonstruktion förbättras täckningen av regioner med hög GC och AT/TA-repeterande regioner i genomet. De specifika fördelarna är följande.
2.1 Effektiviserad biblioteksförberedelseprocess, vilket sparar tid
PCR-fri biblioteksförberedelse eliminerar behovet av PCR-amplifiering och andra steg, vilket effektiviserar bibliotekets förberedelseprocess och sparar tid.
2.2 Minskad biblioteksförberedelsekostnad
I den konventionella biblioteksframställningsprocessen används dyra högtrogna enzymer för PCR-amplifiering för att säkerställa sekvensäkthet. Samtidigt eliminerar PCR-Free PCR-amplifieringssteget, vilket minskar kostnaderna för biblioteksberedning.
2.3 Minska förstärkningsbias
DNA-amplifieringsenzymer har vissa amplifieringsbiaser, speciellt för mallar med signifikanta skillnader i GC-innehåll eller sekundär struktur. Därför kan PCR-Free effektivt undvika amplifieringsbias som orsakas av polymeraset.
2.4 Ingen PCR-duplicering
PCR-Free kan minska Duplicate Reads och öka Unique Mapping-hastigheter och effektivt datautnyttjande.
2.5 Minskat fel Pare
PCR-amplifiering är mer sannolikt att introducera replikeringsfel av Insertion & Deletion, vilket resulterar i en högre felfrekvens för Indel-sekvensering, medan PCR-Free effektivt kan undvika generering och ackumulering av sådana fel.
3. Datavisning av PCR-Free
Hieff NGS™ Ultima Pro PCR Free DNA Library Prep Kit V2 (Cat# 12196ES) har en bättre bibliotekskonverteringsfrekvens än konkurrerande produkter.
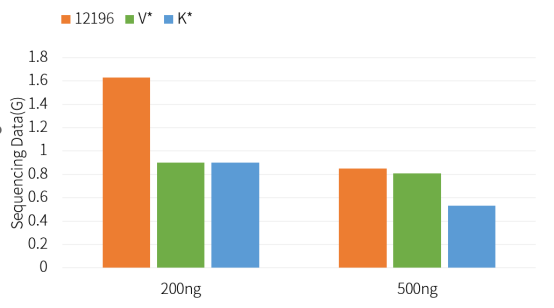
Figur 2. Kvantitet off-machine sekvenseringsdata för PCR-fri bibliotekskonstruktion
4. Vanliga frågor
F: Vilka typer av prover är lämpliga för PCR-fria bibliotek?
S: Vissa prover med komplexa sekundära strukturer, hög GC och PCR-preferens kan välja ett PCR-fritt bibliotek att konstruera.
F: Vad är den allmänna fragmentstorleken för det PCR-fria biblioteket?
S: Det PCR-fria biblioteket i sig har inga speciella krav på fragmentstorleken. PCR-produktbiblioteket, biblioteket konstrueras enligt storleken på PCR-produkten. För det genomiska biblioteket rekommenderas det att vara runt 350 bp, så att kvaliteten på biblioteksdata är relativt bra.
5. Produktinformation
Den produkt som Yeasen kan tillhandahålla visas i tabell 1.
Tabell 1. Produktinformation
| Produkt Namn | Katt. Inga. | Storlek |
| Hieff NGS™ Ultima Pro PCR Free DNA Library Prep Kit V2 | 12196ES24/96 | 24/96T |
6. Angående läsning
Lösningen för NGS-biblioteksberedningen från DNA-prover
Om NGS-relaterad teknik, hur mycket vet du?
