Beskrivning
Hieff NGSTM EvoMax RNA Library Prep Kit (dUTP) är en färdigblandad, aktinomycin D gratis och strängspecifik total RNA-sekvenseringsbibliotek prep kit kompatibel med Illumina och MGI-plattformar. Denna produkt finns i två typer: rör eller tätningssats, och detta kit är mer bekväm oavsett om det är en automatiserad vätskehanteringsenhet eller manuell manipulation . Satsen innehåller RNA-fragmenteringsreagenser, reagens för omvänd transkription, strängspecifika ds-cDNA-syntesreagens och biblioteksförstärkningsreagens. Den kan kopplas till mRNA-reningssatsen eller rRNA-borttagningssatsen till göra mRNA- eller lncRNA-forskning. Denna produkt optimerar modulen för omvänd transkription för att erhålla ett bibliotek med hög kedjespecificitet utan aktinomycin D, vilket säkerställer säkerheten för försökspersonerna i större utsträckning. Alla reagenser utsattes för strikt kvalitetskontroll och funktionell validering för att maximera stabiliteten och repeterbarheten hos biblioteket förberedelse.
Specifikationer
| Kat.nr. | 12340ES24/12340ES96/12340ES97/12340ES98 |
| Storlek | 24 T / 96 T / 96 T (automation) / 96 T (plåt) |
Komponenter
| Komponentnr. | Namn | 12340ES24 | 12340ES96 | 12340ES97 (automatisering) | 12340ES98 (Plåt)** |
| 12340-A | Frag/Prime Buffer | 450 μL | 2×900 μL | 2×1064 μL | 8×266 μL |
| 12340-B | Första reaktionsmodul 2.0 | 192 μL | 768 μL | 960 μL | 8×120 μL |
| 12340-C | 2nd Reaction Module (dUTP) | 840 μL | 3×1120 μL | 3×1280 μL | 8×480 μL |
| 12340-D | Ligeringsreaktionsmodul | 840 μL | 3×1120 μL | 3×1280 μL | 8×480 μL |
| 12340-E | 2×Super Canace® II High-Fidelity Mix | 600 μL | 2×1200 μL | 2×1360 μL | 8×340 μL |
| * | Primer Mix* | / | / | / | / |
Obs: * Notationen anger att komponenten inte ingår i satsen. Primermix krävs om de kompletta adaptrarna används, annars är det inte't. Detta kit är kompatibelt med Illumina- och MGI-plattformar, men behöver ytterligare primermix (Cat # 13334 Primer Mix för MGITM och Cat # 13335 Primer Mix for Illumina®) för Illumina® eller MGI-plattform om de kompletta adaptrarna används.
Figur
Layouten för plattreagensgruppen.

Figur 1: reagenslayout för förseglingsplattans bibliotekskit

Figur 2. Förenklade komponenter av EvoMax RNA Library Prep Kit.
Lagring
Denna produkt bör förvaras vid -25~-15 ℃ för 1 år.
Instruktioner
1. Den rekommenderade mängden att tillsätta i ett 20 μL-system är 0,1-1 enheter (U), och inmatad kvantitet kan justeras baserat på de faktiska resultaten.
2. Enligt experimentets krav kan den slutliga koncentrationen av dUTP justeras mellan 0,2 ~ 0,6 mM och 0,2 mM dTTP kan tillsättas selektivt.
3. Reaktionstiden vid 25~37℃ kan justeras inom 5 ~ 10 min enligt experimentkraven.
Anteckningar
1. Drift
1) Använd labbrockar och engångshandskar,för din säkerhet.
2) Tina komponenterna i rumstemperatur. Blanda noggrant genom att vända upp och ner flera gånger, snurra ner kort och lägg på is för användning.
3) Det rekommenderas att utföra varje stegsreaktion i en termocykler med uppvärmt lock. Termocyklern bör förvärmas till den inställda temperaturen före användning.
4) Tillförsel fria från RNase-kontamination och rengöring av experimentområdet regelbundet är nödvändigt. ThermoFishers RNAZapTM Spray för borttagning av nukleinsyra med hög effektivitet rekommenderades för att avlägsna RNas-kontamination.
5) Felaktiga åtgärder kan med stor sannolikhet orsaka aerosolkontamination, vilket påverkar resultatets noggrannhet. Obligatorisk fysisk isolering av PCR-reaktionsblandningsregioner och PCR-produktreningsanalysregioner rekommenderas. Utrustad med utrustning som specialiserade pipetter för biblioteksbyggande.
6) Detta reagens är för engångsanvändning. Fleranvändning är strängt förbjudet.
7) Denna produkt är endast avsedd för forskning.
2. Adapterligering
1) Illumina eller MGI Long Adapter (Barcoded Adapter) kit och kort Adapter kit finns tillgängliga för kunderna att välja enligt deras experimentella krav.
2) Att välja kommersiella adaptrar av hög kvalitet rekommenderades. Om egentillverkade adaptrar väljs, anförtro ett företag med erfarenhet av NGS-primersyntes och påpeka behovet av strikt kontamineringskontroll. Dessutom rekommenderas att förbereda DNA-glödgningslösning på en ren bänk och endast använda en typ av adapter varje gång för att förhindra korskontaminering.
3) Tina adaptrarna på isen eller vid 4°C; vid drift i rumstemperatur bör laboratorietemperaturen inte överstiga 25°C för att förhindra att adaptrarna denatureras.
4) Koncentrationen av adaptern påverkar direkt ligeringseffektiviteten och biblioteksutbytet. Adaptervolymen som läggs till satsen är fixerad till 5 μl. Adaptrarna rekommenderas att spädas med 0,1×TE-buffert och de utspädda adaptrarna kan förvaras vid 4°C i 48 timmar. Tabell 1 listar den rekommenderade adaptermängden för olika mängder ingående RNA.
Tabell 1-1 Den rekommenderade mängden Illumina-adapter för olika ingående RNA
| Indata totalt RNA | Illumina® Adapter lagerkoncentration |
| 10 ng | 1 μM |
| 100 ng | 1,5 μM |
| 500 ng | 3 μM |
| ≥1 μg | 5 μM |
Tabell 1-2 Den rekommenderade MGI-adaptermängden för olika inmatade RNA
| Indata totalt RNA | MGI® Adapter lagerkoncentration |
| 10~99 ng | 1 μM |
| 100~499 ng | 2 μM |
| 500~4000 ng | 5 μM |
* Adapteranvändning kan justeras efter olika typer av totala RNA-prover och inmatad mängd
3.Biblioteksförstärkning
1) På basis av den första generationens DNA-polymeras, har högfidelitets-DNA-polymeraset i kitet avsevärt förbättrat sin amplifieringslikformighet och uppvisar ingen amplifieringsbias.
2) Antalet amplifieringscykel bör vara strikt kontrollerade. Otillräcklig amplifiering kan leda till lågt biblioteksutbyte; Överamplifiering kan introducera ökad bias, fel, duplicerad läsning, chimära produkter och ackumulering av expansionsmutationer. Tabell 2 listar de rekommenderade cykelnumren för PCR-amplifiering.
3) Det rekommenderade antalet cykler som rekommenderas i Tabell 2 kan uppfylla de allra flesta krav på biblioteksförberedelser. Om ditt prov är av dålig kvalitet (som FFPE-prover med allvarlig nedbrytning), kan antalet cykler ökas på lämpligt sätt, beroende på den faktiska situationen. Var uppmärksam på att notera att mRNA varierar från olika arter och vävnader, antalet amplifieringscykler bör justeras.
Tabell 2 Indata Total RNA-volym och amplifieringscykler rekommenderade Tabell *
| Indata totalt RNA | Antal cykler |
| 10 ng | 16 |
| 100 ng | 14 |
| 500 ng | 12 |
| 1 μg | 11 |
[Notera]: *Utbytet av biblioteket är inte bara relaterat till ingångskvantiteten och antalet amplifieringscykler, utan påverkas också av kvaliteten på prover, fragmenteringsförhållanden och sorteringsförhållanden. I processen med biblioteksbyggandet, välj de lämpligaste förhållandena enligt den faktiska situationen.
4. Pärlbaserad DNA-rensning och storleksval
1) Det finns flera steg i bibliotekskonstruktionsprocessen som kräver DNA-rening av magnetiska pärlor. Vi rekommenderar Hieff NGS™ DNA-selektionspärlor (Yeasen Cat#12601) eller AMPure® XP magnetiska pärlor (Beckman Cat#A63880) för DNA-rening och storleksval.
2) Den magnetiska pärlan bör balanseras till rumstemperatur före användning, annars avkastningen kommer att minska och storleksvalseffekten påverkas.
3) De magnetiska pärlorna ska blandas väl genom virvel eller pipettering före användning.
4) Aspirera inte pärlorna när du överför supernatanten, även spårmängder av pärlorna kan påverka följande reaktioner.
5) Etanolen på 80 % bör vara nyberedd, annars kommer det att påverka återvinningseffektiviteten.
6) De magnetiska pärlorna bör torkas i rumstemperatur innan produkten elueras. Otillräcklig torrhet leder lätt till att etanolrester påverkar efterföljande reaktioner; överdriven torrhet gör att de magnetiska pärlorna spricker och minskar reningsutbytet. Normalt räcker det att torka i rumstemperatur i 3-5 minuter för att pärlorna ska torka helt.
7) Om det behövs kan de renade eller storleksvalda DNA-proverna eluerade i TE-buffert förvaras vid 4°C i 1-2 veckor eller vid -20°C i en månad.
5.Bibliotekskvalitetsanalys
I allmänhet kan kvaliteten på det konstruerade biblioteket utvärderas genom koncentrationsdetektion och längdfördelningsdetektion.
6. Annat material
1) mRNA-anrikningssats: Hieff NGS® mRNA Isolation Master Kit V2 (Yeasen Cat # 12629).
2) rRNA-borttagningskit: Hieff NGS® MaxUp Human rRNA-utarmningskit (rRNA & ITS / ETS) (Yeasen Cat # 12257) eller annat rRNA-borttagningskit.
3) RNA-rening av magnetiska pärlor: Hieff NGS® RNA Cleaner (Yeasen Cat # 12602) eller andra likvärdiga produkter.
4) DNA-renade magnetiska pärlor: Hieff NGS® DNA Selection Beads (Yeasen Cat # 12601) eller AMPure® XP Beads (A63880) eller andra likvärdiga produkter.
5) RNA kvalitetskontroll: Agilent 2100 Bioanalyzer RNA 6000 Nano / Pico Chip eller andra likvärdiga produkter.
6) Adaptrar: Komplett adapter för Illumina®-användning (Cat # 13519-13520 eller annan likvärdig) eller Komplett Adapter för MGI®-användning (Cat # 13360-13362 eller annan likvärdig).
7. Bibliotekskvalitetsinspektion
Agilent 2100 Bioanalyzer DNA 1000 Chip / High Sensitivity Chip eller andra likvärdiga produkter; bibliotekskvantifieringsreagenser.
8. Övrigt material
Vattenfri etanol, sterilt ultrarent vatten, pistolhuvud med låg adsorption, PCR-rör, magnetisk ram, PCR-instrument, etc.
Flödesschema

Figur 1: Byggprocessen för RNA-bibliotek
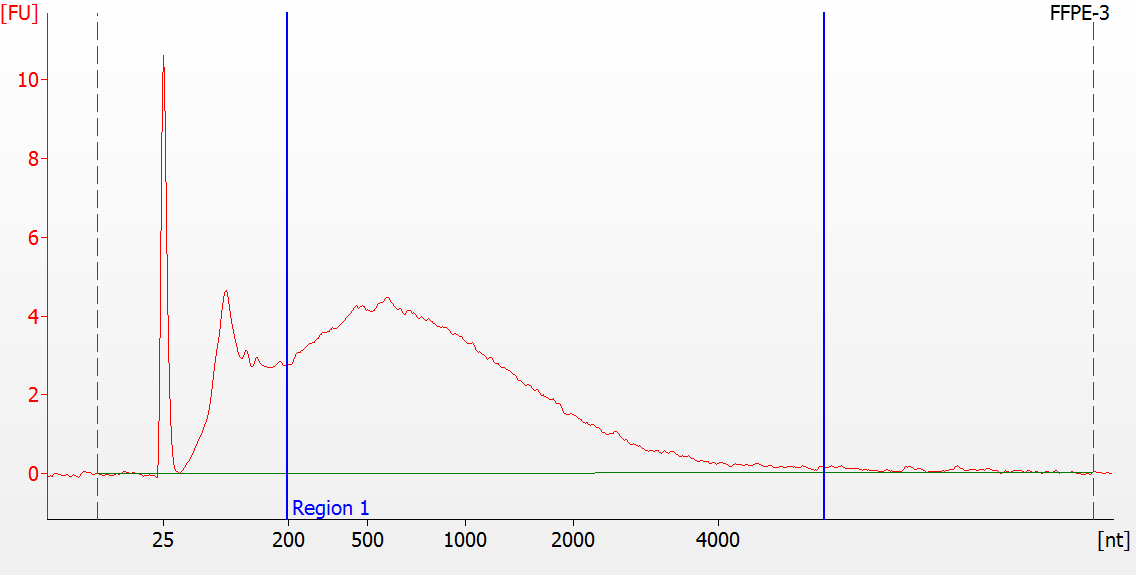
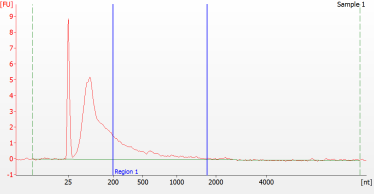
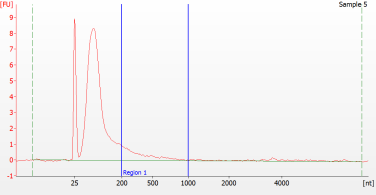
RNA Library förbereder för FFPE-prover av olika kvalitet
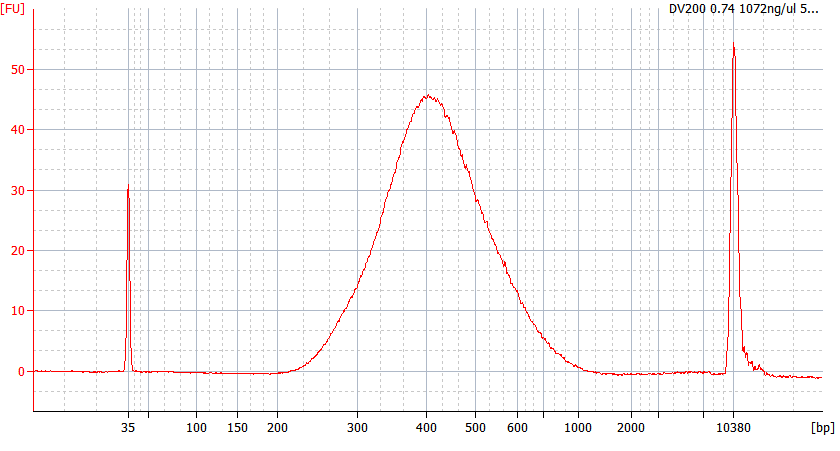
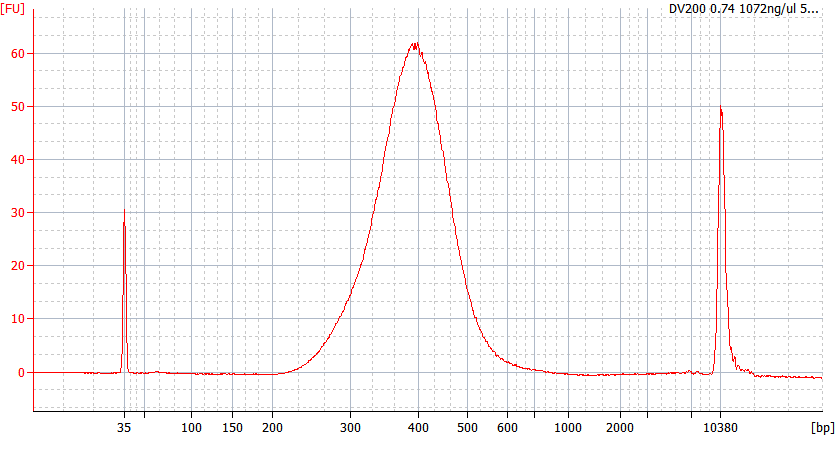
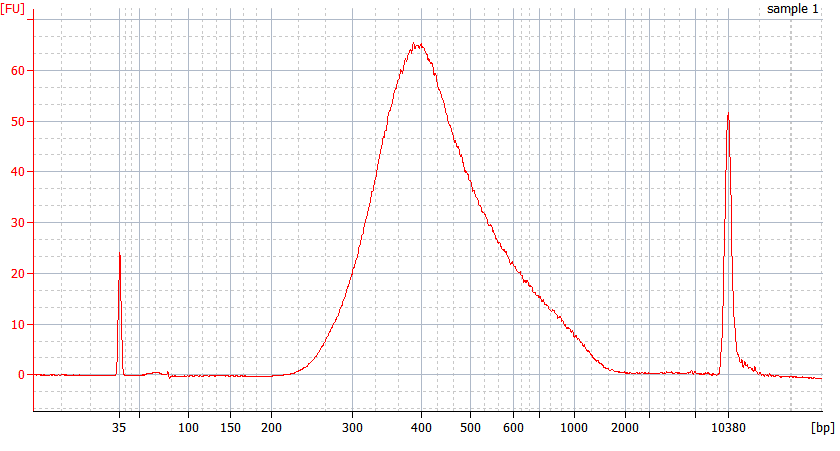
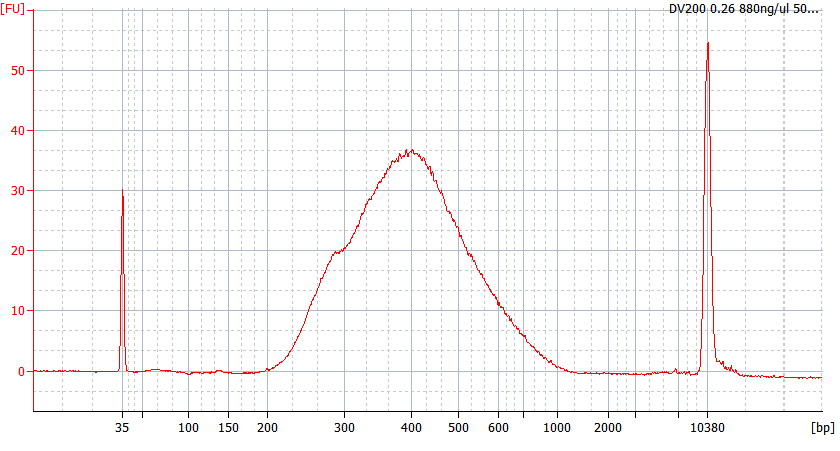
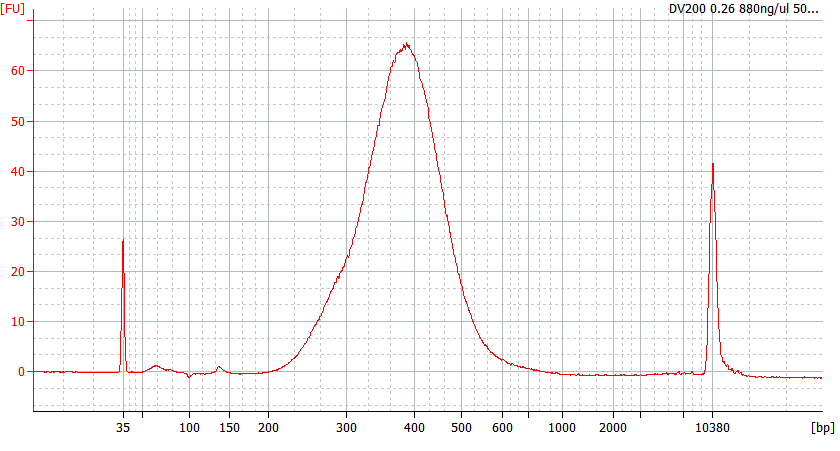
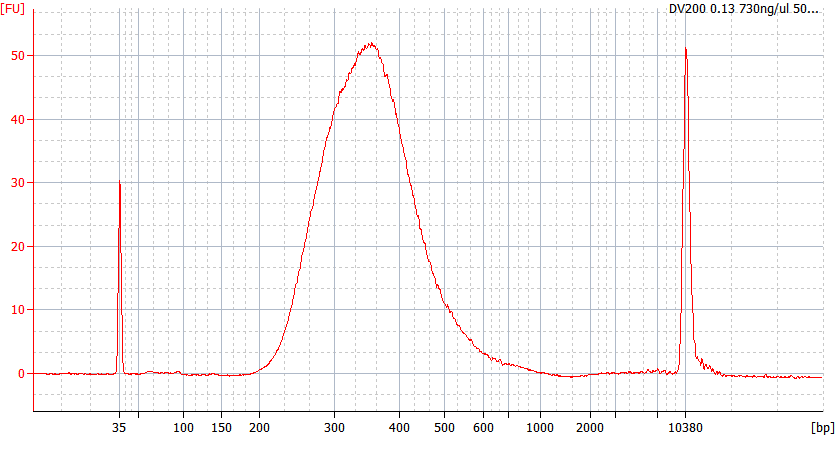
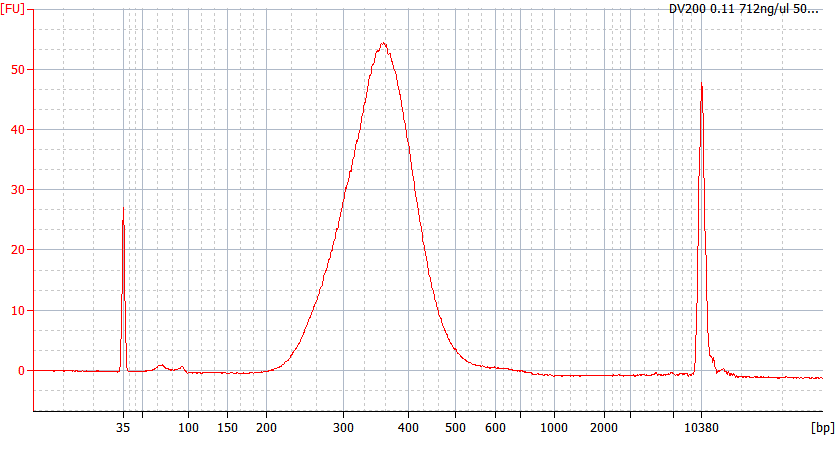
För allvarligt nedbrutet FFPE-RNA (DV 200 <50%) och låginmatade prover rekommenderar vi ett dubbelreningsprotokoll efter adapterligering för att minska biblioteksförlusten.
Toppmönster av FFPE-prover av olika kvalitet
Betalning och säkerhet
Din betalningsinformation behandlas säkert. Vi lagrar inte kreditkortsuppgifter och har inte heller tillgång till din kreditkortsinformation.
Förfrågan
Du kanske också gillar
Vanliga frågor
Produkten är endast avsedd för forskningsändamål och är inte avsedd för terapeutisk eller diagnostisk användning hos människor eller djur. Produkter och innehåll skyddas av patent, varumärken och upphovsrätt som ägs av Yeasen Biotechnology. Varumärkessymboler anger ursprungsland, inte nödvändigtvis registrering i alla regioner.
Vissa applikationer kan kräva ytterligare immateriella rättigheter från tredje part.
Yeasen är dedikerad till etisk vetenskap, och anser att vår forskning bör behandla kritiska frågor samtidigt som den säkerställer säkerhet och etiska standarder.