Beskrivning
Hieff NGS™ DNA-selektionspärlor framställs baserat på SPRI-principen (Solid Phase Reverse Immobilization) och kan användas för DNA-rening och storleksval under beredningen av nästa generations sekvenseringsbibliotek (NGS). Hieff NGS™ DNA Selection Beads är kompatibel med olika DNA- och RNA-biblioteksförberedande kit och är ett bra alternativ till AMPure pärlor.
Komponenter
| Komponentnr. | Namn | 12601ES08 | 12601ES56 | 12601ES75 |
| 12601 | Hieff NGS™ DNA-selektionspärlor | 5 ml | 60 ml | 450 ml |
Specifikationer
| Produktlinje | DNA ren och urval pärlor |
| Utgångsmaterial | DNA |
| Kompatibilitet | DNA |
| Isoleringsteknik | Magnetisk pärla |
| Slutlig produkttyp | DNA |
| För användning med (applikation) | DNA celan up, DNA storlek selektion |
Frakt och förvaring
Pärlorna skickas med isförpackningar och kan förvaras vid 2°C-8°C i ett år.
Instruktioner
- 1. Förberedelse
Utjämna urvalspärlorna vid rumstemperatur i minst 30 minuter före användning.
- 2. Val av DNA-storlek
Operationsflödet för storleksval visas i figur 1 och protokollet är som följer.
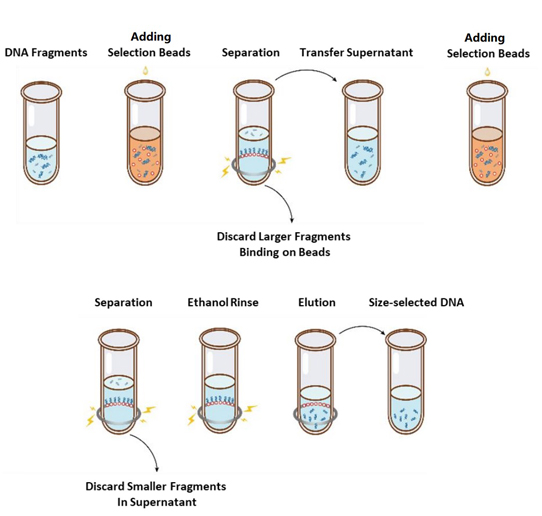
Figur 1. Flödesschemat för urval av DNA-storlek
2.1 Blanda pärlorna noggrant genom att vortexa eller pipettera upp och ner varje gång före användning.
2.2 Lägg till den första omgången av urvalspärlor till provet (se tabell 1). Blanda noggrant genom att vortexa eller pipettera upp och ner minst 10 gånger.
2.3 Inkubera i rumstemperatur i 5 min.
2.4 Snurra ner röret kort och placera det på magnetstativ. När lösningen är klar (ca 5 min), överför supernatanten till ett nytt PCR-rör.
2.5 Lägg till den andra omgången av urvalspärlor till provet från steg 2.4 enligt tabell 1. Blanda noggrant genom att vortexa eller pipettera upp och ner minst 10 gånger.
2.6 Inkubera vid rumstemperatur i 5 min.
2.7 Snurra ner röret kort och placera det på magnetstativ. När lösningen är klar (ca 5 min), aspirera supernatanten och kassera.
2.8 Håll röret i det magnetiska stativet och tillsätt 200 μL nyberedd 80 % etanol för att utan att störa pärlorna, inkubera i rumstemperatur i 30 sekunder. Aspirera etanolen och kassera.
2.9 Upprepa steg 2.8 en gång för totalt två tvättar.
2.10 Ta bort resterande etanol med 10 µL pipettspetsar. Håll röret i det magnetiska stativet, lufttorka urvalspärlorna med locket öppet tills sprickor precis uppstår (ca 5 min).
Obs: Torka inte valpärlorna för mycket. Detta kan resultera i lägre återställnings-DNA-mål.
2.11 Ta bort röret från magnetstativet. Tillsätt en lämplig mängd ddH2O (≥20 µL) och blanda noggrant genom att vortexa eller pipettera upp och ner minst 10 gånger.
2.12 Inkubera i rumstemperatur i 5 min.
Snurra ner röret kort och placera det på magnetstativet. När lösningen är klar (cirka 5 minuter), överför 20 μL av supernatanten till ett nytt rör.
- 3. Rekommenderade villkor för urval av DNA-storlek
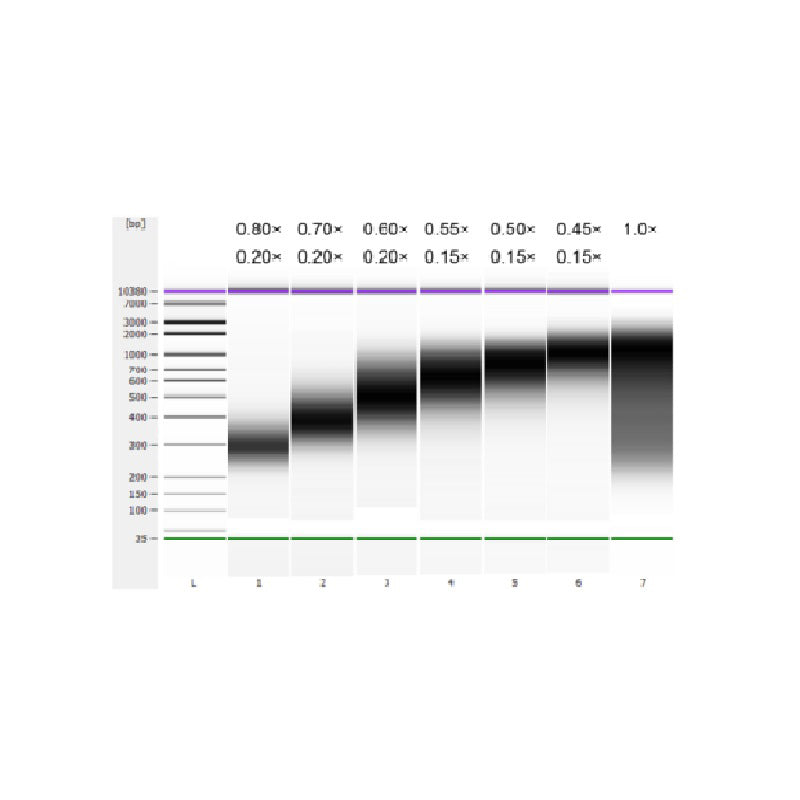
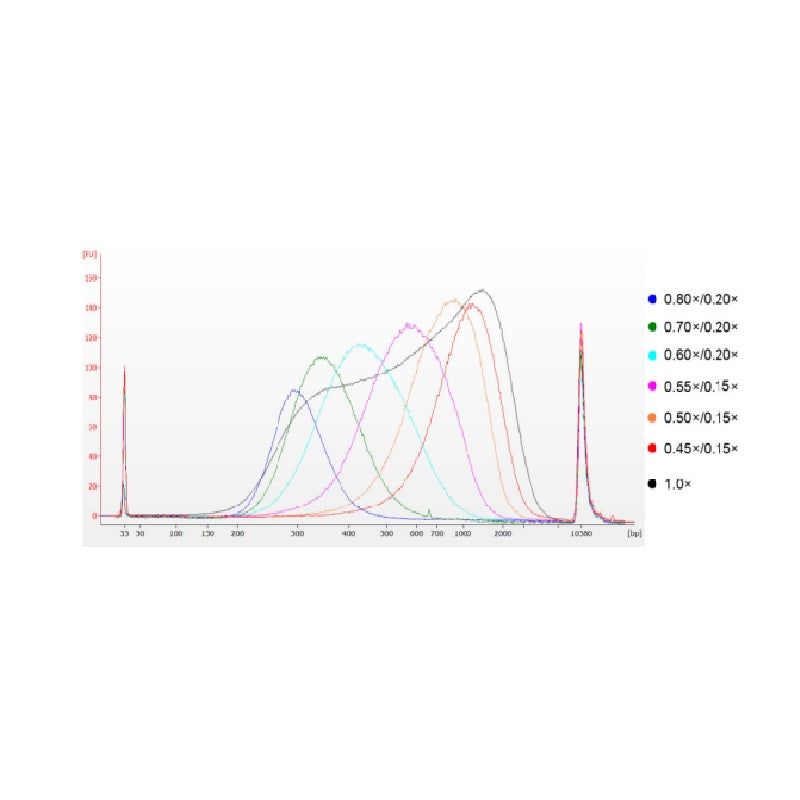
Kalvtymus-DNA:t fragmenterades genom sonikering för att framställa ett fragment på 100-1 000 bp, och två omgångar av storleksval utfördes enligt tabell 1. Resultaten analyserades med användning av Agilent 2100 Bioanalyzer (Figur 2).
Tabell 1. Rekommenderat villkor för val av DNA-storlek
| Längd på DNA-fragment | 250-350 bp | 320-420 bp | 450-550 bp | 550-700 bp | 700-900 bp | 800-1 000 bp |
| Förhållande av pärlor: DNA för 1:a omgången | 0,80× | 0,70× | 0,60× | 0,55× | 0,50× | 0,45× |
| Förhållande av pärlor: DNA för 2:an Runda | 0,20× | 0,20× | 0,20× | 0,15× | 0,15× | 0,15× |
Obs: "×" i tabellen anger volymen av prov-DNA. Till exempel, om insättningslängden på biblioteket är 250 bp och provets DNA-volym är 100 μL, är volymen magnetiska pärlor som används i den första sorteringsrundan 0,80×100 μL=80 μL; volymen av magnetiska pärlor som används i den andra sorteringsrundan är 0,20× 100 μL=20 μL.
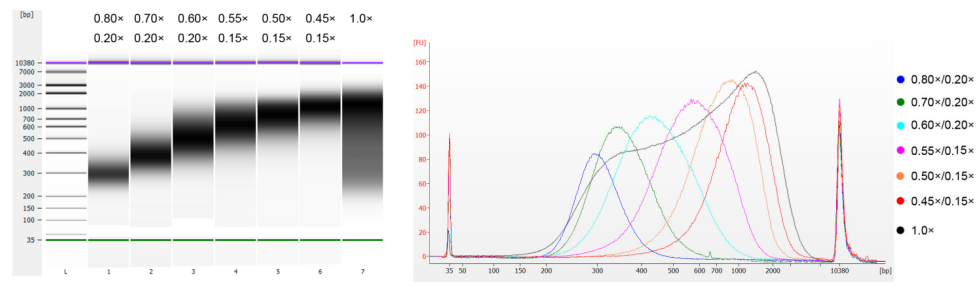
Figur 2. Agilent 2100 högkänsligt DNA-chipelektroferogram
Anmärkningar:
1. För din säkerhet och hälsa, vänligen bär laboratorierockar och engångshandskar vid användning.

Citerad från "Sekvensspecifik integration av familj 1-kasposas från Candidatus Nitrosopumilus koreensis AR1. Nucleic Acids Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725"

Citerad från "Recent infektion av Wolbachia förändrar mikrobiella samhällen i vilda Laodelphax striatellus populationer. Mikrobiom. 2020;8(1):104. Publicerad 2020 juli 2. doi:10.1186/s40168-020-00878-x"
[1] Wang X, Yuan Q, Zhang W, et al. Sekvensspecifik integration av familj 1-kasposas från Candidatus Nitrosopumilus koreensis AR1. Nucleic Acids Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725(IF:16.971)
[2] Duan XZ, Sun JT, Wang LT, et al. Ny infektion av Wolbachia förändrar mikrobiella samhällen i vilda Laodelphax striatellus-populationer. Mikrobiom. 2020;8(1):104. Publicerad 2020 juli. doi:10.1186/s40168-020-00878-x(IF:11.607)
[3] Song B, Almatrafi E, Sang F, et al.Hantera Fenton-behandlat sediment med biokol och fårgödselkompost: Effekter på de evolutionära egenskaperna hos bakteriesamhället. J Environ Manage. 2022;316:115218. doi:10.1016/j.jenvman.2022.115218(IF:6.789)
[4] Huang C, Mei Q, Lou L, et al. Ulcerös kolit som svar på fekal mikrobiotatransplantation via modulering av tarmmikrobiota och Th17/Treg-cellbalans. Celler. 2022;11(11):1851. Publicerad 2022 5 juni. doi:10.3390/cells11111851(IF:6.600)
[5] Ghosh S, Yang X, Wang L, Zhang C, Zhao L. Aktiv fas av prebiotisk utfodring förändrar tarmmikrobiota, inducerar viktoberoende lindring av leversteatos och serumkolesterol hos möss som matats med fettrik diet. Comput Struct Biotechnol J. 2020;19:448-458. Publicerad 2020 24 dec. doi:10.1016/j.csbj.2020.12.011(IF:6.018)
[6] Gao X, Yu B, Yu J, et al. Utvecklingsprofilering av matsmältning av kolhydrater hos smågrisar. Främre Microbiol. 2022;13:896660. Publicerad 2022 29 april. doi:10.3389/fmicb.2022.896660(IF:5.640)
[7] Li P, Zhang Y, Yan F, Zhou X. Characteristics of a Bacteriophage, vB_Kox_ZX8, Isolated From Clinical Klebsiella oxytoca och dess terapeutiska effekt på möss bakteriemi. Främre Microbiol. 2021;12:763136. Publicerad 2021 3 dec. doi:10.3389/fmicb.2021.763136(IF:5.640)
[8] Lin Z, Luo P, Huang D, Wu Y, Li F, Liu H. Multi-omics-baserad strategi för toxicitetsanalys av akrylamid i Saccharomyces cerevisiae-modell. Chem Biol Interact. 2021;349:109682. doi:10.1016/j.cbi.2021.109682(IF:5.194)
[9] Sun X, Lv W, Wang Y, et al. Mrgprb2-genen spelar en roll i de anafylaktoida reaktioner som induceras av Houttuynia cordata-injektion. J Etnopharmacol. 2022;289:115053. doi:10.1016/j.jep.2022.115053(IF:4.360)
[10] Ma H, Lai B, Zan C, Di X, Zhu X, Wang K. GLO1 bidrar till läkemedelsresistens hos Escherichia coli genom att inducera PER-typ av β-laktamaser med utökat spektrum. Infect Drug Resist. 2022;15:1573-1586. Publicerad 2022 5 april doi:10.2147/IDR.S358578(IF:4.003)
[11] Zhong Y, Zhao W, Tang Z, et al. Jämförande transkriptomisk analys av de olika utvecklingsstadierna av äggstockar hos röd träskkräfta Procambarus clarkii. BMC Genomics. 2021;22(1):199. Publicerad 2021 21 mars. doi:10.1186/s12864-021-07537-x(IF:3.969)
[12] Lian C, Yang H, Lan J, et al. Jämförande analys av kloroplastgenom avslöjar fylogenetiska samband och intraspecifik variation i medicinalväxten Isodon rubescens. PLoS One. 2022;17(4):e0266546. Publicerad 2022 6 april. doi:10.1371/journal.pone.0266546(IF:3.240)
[13] Diao G, Huang J, Zheng X, et al. Prostaglandin E2 har en dubbel roll för att reglera migrationen av dendritiska celler. Int J Mol Med. 2021;47(1):207-218. doi:10.3892/ijmm.2020.4801(IF:3.098)
[14] Bing XL, Zhao DS, Peng CW, Huang HJ, Hong XY. Likheter och rumsliga variationer av bakterie- och svampsamhällen i populationer av åkerrisplanthoppare (Hemiptera: Delphacidae). Insect Sci. 2020;27(5):947-963. doi:10.1111/1744-7917.12782(IF:2.791)
[15] Li X, Zhou S, Zhang J, Zhou Z, Xiong Q. Riktningsförändringar i tarmbakteriegemenskapen hos svart soldatfluga (Hermetia illucens) larver. Djur (Basel). 2021;11(12):3475. Publicerad 6 december 2021. doi:10.3390/ani11123475(IF:2.752)
[16] Yang J, Peng Y, Kong W. Identifiering och fullständig genomsekvens av mullbärskryptiska virus 1. Arch Virol. 2022;167(2):687-690. doi:10.1007/s00705-021-05350-1(IF:2.574)
[17] Chang Y, Xia X, Sui L, et al. Endofytisk kolonisering av entomopatogena svampar ökar resistens mot växtsjukdomar genom att förändra det endofytiska bakteriesamhället. J Basic Microbiol. 2021;61(12):1098-1112. doi:10.1002/jobm.202100494(IF:2.281)
[18] Ding CY, Ma YM, Li B, et al. Identifiering och funktionell analys av differentiellt uttryckta gener i Myzus persicae (Hemiptera: Aphididae) som svar på transanetol. J Insect Sci. 2022;22(1):3. doi:10.1093/jisesa/ieab094(IF:1.857)
Betalning och säkerhet
Din betalningsinformation behandlas säkert. Vi lagrar inte kreditkortsuppgifter och har inte heller tillgång till din kreditkortsinformation.
Förfrågan
Du kanske också gillar
Vanliga frågor
Produkten är endast avsedd för forskningsändamål och är inte avsedd för terapeutisk eller diagnostisk användning hos människor eller djur. Produkter och innehåll skyddas av patent, varumärken och upphovsrätt som ägs av Yeasen Biotechnology. Varumärkessymboler anger ursprungsland, inte nödvändigtvis registrering i alla regioner.
Vissa applikationer kan kräva ytterligare immateriella rättigheter från tredje part.
Yeasen är dedikerad till etisk vetenskap, och anser att vår forskning bör behandla kritiska frågor samtidigt som den säkerställer säkerhet och etiska standarder.