Beskrivning
Replikationskompetent Lentivirus (RCL) Detection Kit används att kvantitativt upptäcka replikerande lentivirus som kan förekomma i en olika cellprodukter associerade med lentiviral vektorer potentiella risker.
Detta kit designar specifika primers för VSV-G-gensekvens för lentivirala höljeproteiner. Och den antar taqman fluorescerande sond och polymeraskedjereaktionsmetoden (PCR), som har 1 kopior/μL nivådetektionsgräns och kan specifikt och snabbt upptäcka den replikationskompetenta lentivirusrisken. Satsen behöver att användas tillsammans med Residual DNA Sample Preparation Kit (katnummer 18461ES).
Komponenter
| Komponentnr. | Namn | 41311ES50 | 41311ES60 |
| 41311-A | RCL qPCR Mix | 0,75 ml | 1.5 ml |
| 41311-B | RCL Primer & Probe Mix | 200 μL | 400 μL |
| 41311-C | DNA-spädning Buffert | 2×1,8 ml | 4×1,8 ml |
| 41311-D | RCL DNA-kontroll(5×10E8 kopior/μL)) | 25 μL | 50 μL |
| 41311-E | IC* | 50 μL | 100 μL |
* IC: Inre kontrollera.
Lagring
Denna produkt bör förvaras vid -25~-15 ℃ för 2 år.
Både 41311-A och 41311-B bör förvaras skyddade från ljus.
Tillämplig instrument modeller
Inkludera men inte begränsat till: Bio-Rad: CFX96 optisk modul; Thermo Scientific: ABI 7500; ABI Quant Studio 5; ABI Step OnePlus.
Instruktioner
- RCL DNA Standard utspädning och Standard kurva förberedelse
RCL DNA-kontrollen gradientspäddes med hjälp av DNA-spädningsbufferten som medföljde i satsen* och spädningen koncentrationen är 5×10E7 kopior/μL, 5×10E6 kopior/μL, 5×10E5 kopior/μL, 5×10E4 kopior/μL, 5×10E3 kopior/μL, 5×10E2 kopior/μL, 5×10E1 kopior/μL.
Se detaljerade instruktioner nedan:
1) Tina RCL DNA-kontrollen och DNA-spädningsbufferten på is. Efter helt upptinad, vortex försiktigt till blanda, och centrifugera vid låg hastighet i 10 sekunder.
2) Ta ut sju rena 1,5 ml rör, märkta med Std0, Std1, Std2, Std3, Std4, Std5, Std6.
3) Lägg till 90 μL DNA-spädningsbuffert och 10 μL RCL DNA-kontroll till 1,5 mL mikrofugröret märkt Std0, nämligen späd till 5×10E7 kopior/μL. Blanda och centrifugera sedan i 10 sekunder. Underpaketera den utspädda DNA-standarden och det kan vara lagras på kort sikt (högst 3 månader) vid -25~-15℃** . Undvik upprepad frys-upptining.
4) Lägg till 90 μL DNA-spädningsbuffert i andra rör*** , följ sedan proceduren nedan för serieutspädningarna**** .
| Rör | Spädningsförhållande | Standardkoncentration |
| Std1 | 10 μL Std0 + 90 μL DNA-spädning Buffert | 5×10E6 kopior/μL |
| Std2 | 10 μL Std1 + 90 μL DNA-spädning Buffert | 5×10E5 kopior/μL |
| Std3 | 10 μL Std2 + 90 μL DNA-spädning Buffert | 5×10E4 kopior/μL |
| Std4 | 10 μL Std3 + 90 μL DNA-spädning Buffert | 5×10E3 kopior/μL |
| Std5 | 10 μL Std4 + 90 μL DNA-spädning Buffert | 5×10E2 kopior/μL |
| Std6 | 10 μL Std5 + 90 μL DNA Utspädning Buffert | 5×10E1 kopior/μL |
Tabell 1 Standardgradientspädning
*Tre replikera brunnar är nödvändig för varje koncentration.Den upptäckt räckvidd är 5×10E1 kopior/μL~5×10E6 kopior/μL och detta räckvidd burk vara utökas vid behov.
** Till minska de antal av upprepa frysa-tina och undvika förorening, det är rekommenderad till lagra de DNA kontrollera i alikvoter på -25~-15 ℃ för de första tid.
*** En gång tinat, DNA utspädning buffert kunde vara lagras på 2-8°C för 7 dagar, om inte begagnad för a lång tid, snälla lagra på -25~-15 ℃ .
**** Göra säker de mall är helt blandad, försiktigt skaka de blandning för 15 sekunder till 1 min för varje lutning utspädning.
- Extraktionsåterställning Kontrollera (ERC) förberedelse
Ställ in koncentrationen av RCL-DNA i ERC efter behov (ERC-provet preparerades med 5×10E4 kopierar RCL DNA som ett exempel), enligt följande:
1) Lägg till 100 μL testprov i ett rent 1,5 ml rör, tillsätt sedan 10 μL 5×10E3 kopior/μL RCL DNA Standard (Std4) och blanda väl, markerad som ERC.
2) Utför DNA-extraktionen av ERC-provet tillsammans med testproverna för att förbereda det renade ERC-provet.
- Negativ kontroll Lösning (NCS) förberedelse
Ställ in den negativa kontrollen i experimentet, de specifika operationsstegen är som följer:
1) Lägg till 100 μL provmatris (eller DNA-spädningsbuffert) i ett rent 1,5 ml rör, märkt sedan som NCS.
2) Utför DNA-extraktionen av NCS prov tillsammans med testproverna för att förbereda den renade NCS prov.
- Ingen mall Kontrollera (NTC) förberedelse
Ställ in ingen mallkontroll i experimentet, de specifika operationsstegen är som följer:
1) NTC kräver ingen provförbehandling och kan konfigureras vid qPCR-detektion av kvarvarande DNA innehåll.
2) NTC-provet i varje rör eller brunn är 20 μL Mix (dvs. 15 μL RCL qPCR Mix + 4 μL RCL Primer & sond Blanda + 1 μL IC) + 10 μL DNA-spädningsbuffert. Det rekommenderas att konfigurera tre replikatbrunnar.
- PCR reaktion system
| Komponent | Volym (μL) |
| RCL qPCR Mix* | 15 |
| RCL Primer & Probe Mix | 4 |
| IC | 1 |
| DNA-mall | 10 |
| Total volym** | 30 |
Tabell 2 Reaktionssystem
* Kalkylera de total PCR reaktion volym av de antal av reaktioner: qPCR Blanda =(den antal av reaktioner+2) × (15+4+1) μL (inklusive de förluster av två reaktionsbrunnar). Mer än tre replikat för varje prov rekommenderas i experimentet.
** Efter täckning de rör eller tätning de plåt, centrifug de reaktion rör eller tallrik på låg hastighet för 10 sekunder. Efter tillräcklig skakning och blandning för 5 sekunder, upprepa centrifug till samla de flytande från de lock eller vägg till de botten. Undvika bubblor under drift.
Se nedanstående tabell för den rekommenderade plattinställningen:
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | NTC |
| TS 1 | TS 1 | TS 1 |
| Std 1 | Std 1 | Std 1 |
|
|
|
| B | NTC |
| TS 2 | TS 2 | TS 2 |
| Std 2 | Std 2 | Std 2 |
|
|
|
| C | NTC |
| TS 3 | TS 3 | TS 3 |
| Std 3 | Std 3 | Std 3 |
|
|
|
| D |
|
|
|
|
|
| Std 4 | Std 4 | Std 4 |
|
|
|
| E | NCS |
| ERC 1 | ERC 1 | ERC 1 |
| Std 5 | Std 5 | Std 5 |
|
|
|
| F | NCS |
| ERC 2 | ERC 2 | ERC 2 |
| Std 6 | Std 6 | Std 6 |
|
|
|
| G | NCS |
| ERC 3 | ERC 3 | ERC 3 |
|
|
|
|
|
|
|
| H |
|
|
|
|
|
|
|
|
|
|
|
|
Tabell 3 Computer-on hänvisning styrelse
Plattlayouten inkluderar: 6 Std (standardkurvan med 6 standardkoncentrationer), 1 NTC (ingen mallkontroll), 1 NCS (negativ kontrolllösning), 3 TS (testprover), 3 ERC (extraktionsåtervinningskontroll).Tre replikatbrunnar för varje prov.
- Riktlinjer för installation för a PCR Instrument
Följande instruktioner gäller endast för Termo ABI 7500 qPCR-instrument (programvara version 2.0). Om du använder en annat instrument, se tillämplig instrumentguide för installationsriktlinjer.
1) Skapa ett nytt experiment, välj mallen för absolut kvantifiering eller användardefinierad.
2) Skapa 1 detektionssond, benämnd "RCL-DNA", välj reporterfluorofor som "FAM" och släck fluorofor som "ingen"; skapa ytterligare en detektionssond, namnge "IC" och välj reporterfluoroforen som "CY5" och släckning fluorofor som "ingen". Referensfluorescensen är ROX" (referensfluorescensen kan baseras på instrumentmodell etc., välj om du måste lägga till det).
3) I I panelen "Sampler", lägg till all information om exempel i tur och ordning. Välj sedan brunnarna, välj målet och proverna på motsvarande sätt. Uppsättning uppgiften att RCL DNA-standard som standard, och tilldela värden 5000000, 500000, 50 000, 5000, 500, 50 (enheten för DNA-koncentration i varje brunn är kopior/μL) i kolumnen Kvantitet och namnge brunnar Std 1, Std 2, Std 3, Std 4, Std 5, Std 6, på motsvarande sätt. Ställ in NTC:s uppgift som NTC. Ställ in NCS, TS och ERC som Okänd, och namngav dem enligt ovanstående plattlayout på motsvarande sätt. Klicka sedan på nästa.
4) Ställ in amplifieringsprogrammet: ställ in reaktionsvolymen på 30 μL.
| Cykelsteg | Temperatur (℃) | Tid | Cyklar |
| Förorenad matsmältning | 37℃ | 5 min | 1 |
| Initial denaturering | 95 ℃ | 5 min | 1 |
| Denaturering | 95 ℃ | 15 sek |
45 |
| Glödgning/förlängning (Fluorescenssamling) | 60 ℃ | 30 sek |
Tabell 4 Amplifieringsprocedur
- Analys av qPCR resultat
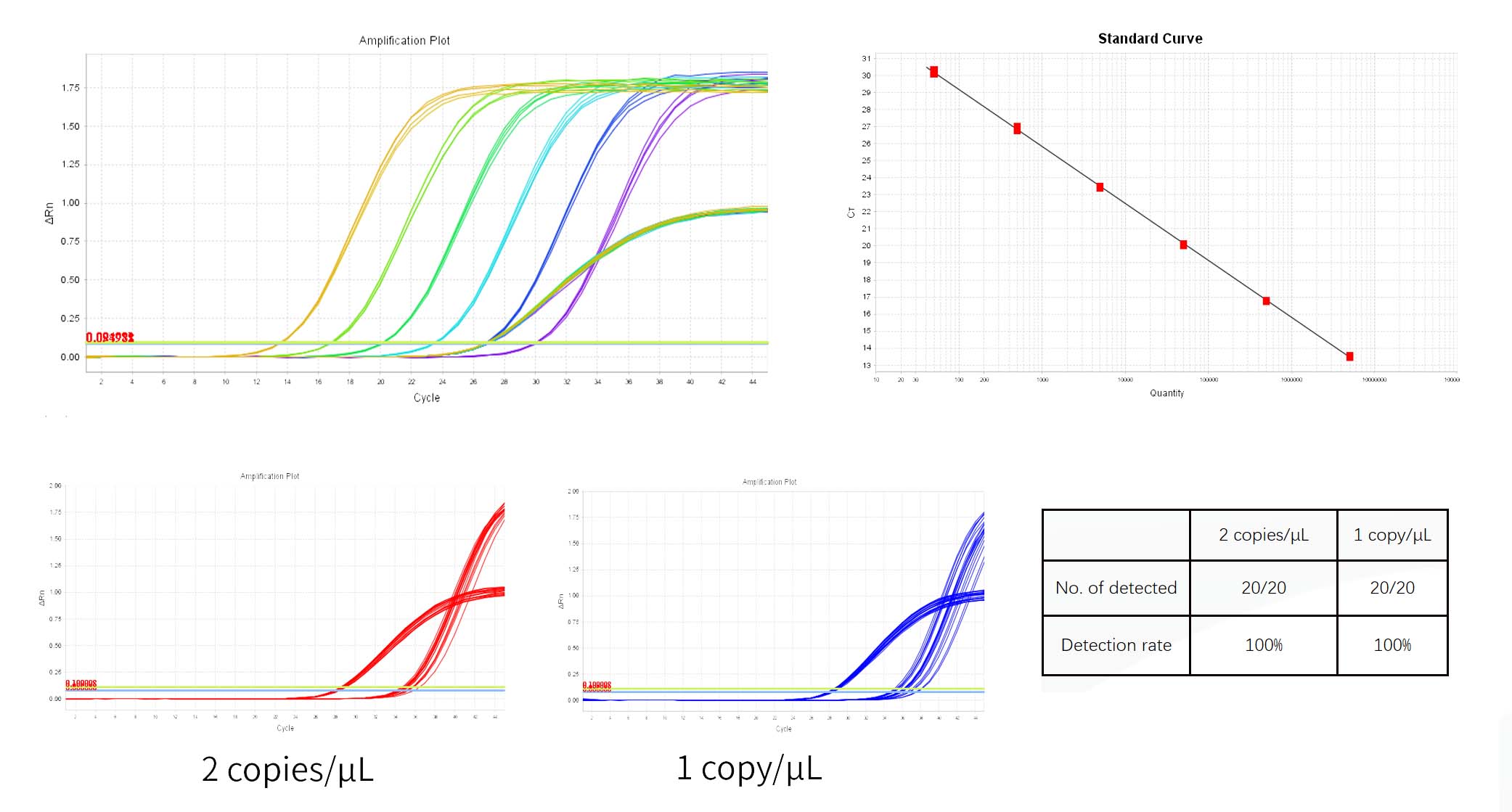
1) Systemet kommer automatiskt att ge tröskeln i Förstärkning Plot panel av Analys. Den angivna tröskeln av systemet är för nära baslinjen ibland, vilket resulterar i en stor skillnad i Ct mellan replikatbrunnar. Du kan justera manuellt tröskeln till en lämplig position och klicka Analysera. Då kan du först kontrollera om förstärkningskurvan är normal i Multicomponent Plot.
2) I Resultatet Fliken Analys, granska standardkurvans plot. Verifiera värden för R2, effektivitet, lutning och Y-skärning. För en normal standardkurva, R²>0,99, 90%≤Eff%≤110%, -3,6≤Lutning≤-3,1.
3) I 'Se brunnsbordsruta in Analys, koncentrationerna av varje prov visas i kvantitet, enheten är kopior/μL kan enheterna konverteras i analysrapporten.
4) Parameterinställningarna för Resultatanalysen måste baseras på den specifika modellen och mjukvaran version används och kan i allmänhet automatiskt tolkas av instrumentet.
5) Beräkna spikåtervinningsgraden baserat på testresultaten för provet TS som ska mätas och provet spike återvinning ERC, återvinningsgraden för spikar krävs att vara mellan 50% ~ 150%. Formel för mätare med spikåtervinningsgrad:
Återhämtning (%) = {Sample spiked assay (eg.copies/μL) - Sample assay (eg.copies/μL)} x Elueringsvolym (μL) / Teoretisk värde av DNA-tillsatsmängd (t.ex. kopior) x 100 %.
6) Ct värdet av den negativa kontrollen NCS bör vara större än medelvärdet av den lägsta koncentrationen Ct av de standard.
7) Mall fri kontroll NTC bör vara Obestämd eller Ct värde ≥38.
Anteckningar
- Denna produkt är endast avsedd för forskning.
- Använd labbrockar och engångshandskar för din säkerhet.
3. Läs denna bruksanvisning noggrant innan du använder den detta reagens, och experimentet bör standardiseras, inklusive provhantering, beredning av reaktionssystem och provtillsats.
4. Se till att varje komponent är helt vortexiserad och centrifugerad vid låg hastighet före användning.
Betalning och säkerhet
Din betalningsinformation behandlas säkert. Vi lagrar inte kreditkortsuppgifter och har inte heller tillgång till din kreditkortsinformation.
Förfrågan
Du kanske också gillar
Vanliga frågor
Produkten är endast avsedd för forskningsändamål och är inte avsedd för terapeutisk eller diagnostisk användning hos människor eller djur. Produkter och innehåll skyddas av patent, varumärken och upphovsrätt som ägs av Yeasen Biotechnology. Varumärkessymboler anger ursprungsland, inte nödvändigtvis registrering i alla regioner.
Vissa applikationer kan kräva ytterligare immateriella rättigheter från tredje part.
Yeasen är dedikerad till etisk vetenskap, och anser att vår forskning bör behandla kritiska frågor samtidigt som den säkerställer säkerhet och etiska standarder.