DNase I และการประยุกต์ใช้ในชีวการแพทย์
Deoxyribonuclease I (DNase I) เป็นเอ็นโดนิวคลีเอสชนิดหนึ่ง ซึ่งไม่เพียงแต่ใช้รักษาความสมบูรณ์ของ RNA เท่านั้น แต่ยังใช้วิเคราะห์รอยเท้าของ DNA การสร้างไลบรารี DNA แบบสุ่ม การลดความเหนียวในไลเสทของเซลล์หรือสารสกัดโปรตีน เป็นต้น กล่าวโดยสรุป DNase I สามารถใช้ได้ในเกือบทุกแอปพลิเคชันที่ต้องมีการแยก DNA ด้วยเอนไซม์ ต่อไปนี้เป็นคำอธิบายโดยละเอียดเกี่ยวกับ DNase I และการใช้งานเฉพาะของมัน
1. DNAse I คืออะไร?
2. DNase I สำหรับการเตรียมการสกัด RNA ที่ปราศจาก DNA
3. DNase I สำหรับการถอดรหัสในหลอดทดลองเพื่อกำจัดเทมเพลตดีเอ็นเอ
4. DNase I สำหรับการกำจัด rRNA
5. DNase I สำหรับการติดฉลาก DNA
6. การใช้งานอื่น ๆ
7. คำแนะนำการเลือกผลิตภัณฑ์ DNase I
1. DNAse I คืออะไร?
ดีออกซีไรโบนิวคลีเอส I (ดีเอ็นเอส I) เป็นเอ็นโดนิวคลีเอสแบบไม่จำเพาะที่สามารถย่อยดีเอ็นเอสายเดี่ยวหรือสายคู่ ซึ่งมีอยู่ในเนื้อเยื่อและของเหลวในร่างกายที่แตกต่างกัน เอ็นโดนิวคลีเอสสามารถไฮโดรไลซ์พันธะฟอสโฟไดเอสเทอร์เพื่อผลิตโมโนและโอลิโกดีออกซีไรโบนิวคลีโอไทด์ที่มีหมู่ฟอสเฟต 5' และหมู่ฟอสเฟต 3' ช่วง pH การทำงานที่เหมาะสมของ DNase I คือ 7-8 กิจกรรมของเอ็นโดนิวคลีโอไทด์ขึ้นอยู่กับ Ca2+ และสามารถเปิดใช้งานได้โดยไอออนโลหะที่มีประจุไฟฟ้าสองขั้ว เช่น Mn2+, Mg2+, Zn2+ เป็นต้น ในสภาพที่มี Mg2+ เอ็นโดนิวคลีโอเอส I จะเฉือนบริเวณใดๆ ของดีเอ็นเอสายคู่แบบสุ่ม ในสภาพที่มี Mn2+ เอ็นโดนิวคลีโอเอส I สามารถเฉือนดีเอ็นเอสายคู่ที่ตำแหน่งเดียวกันเพื่อสร้างปลายทู่หรือปลายเหนียวของนิวคลีโอไทด์ 1-2 ปลายยื่นออกมา
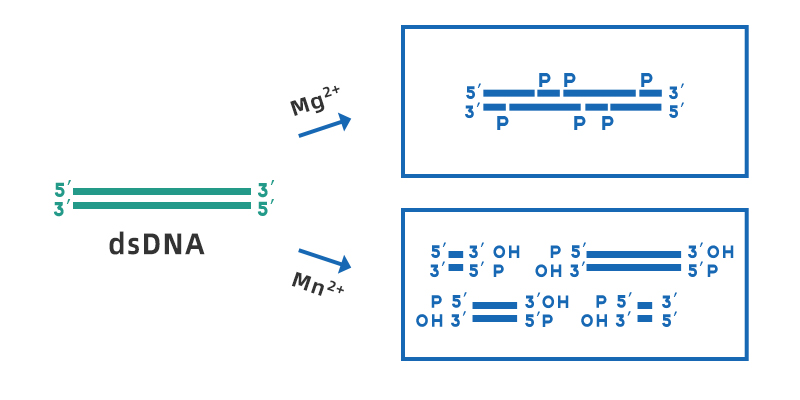
รูปที่ 1 แผนผังแสดงการแยก dsDNA โดย DNase I ในสภาวะที่มี Mg2+ และ Mn2+
แม้ว่าโดยทั่วไปแล้ว การแยก DNase I จะถือว่าเป็นการแยกแบบไม่จำเพาะ แต่ DNase I มีแนวโน้มที่จะทำปฏิกิริยากับชิ้นส่วนลำดับบางชิ้น เช่น บริเวณร่องเล็ก และมีแนวโน้มที่จะทำปฏิกิริยากับลำดับพิวรีน-ไพริมิดีนมากกว่า อย่างไรก็ตาม เมื่อ DNase I ทำปฏิกิริยากับ dsDNA ที่ไม่เป็นเนื้อเดียวกัน เบสทั้งสี่จะถูกตัดออก และผลกระทบต่อเบสเฉพาะตัวหนึ่งจะไม่เกิน 3 เท่าเมื่อเทียบกับเบสอื่นๆ
2. DNase I สำหรับการเตรียมการสกัด RNA ที่ปราศจาก DNA
ในการทดลองทางชีววิทยา ขั้นตอนแรกคือการเตรียมกรดนิวคลีอิกเพื่อศึกษาหน้าที่ต่างๆ ของ RNA อย่างไรก็ตาม เนื่องจาก DNA และ RNA มักถูกปล่อยออกมาพร้อมกันในระหว่างกระบวนการไลซิสเซลล์ จึงไม่สามารถหลีกเลี่ยงการรบกวนของทั้งสองได้ ไม่ว่าจะใช้สารละลายสกัดชนิดใดก็ตาม ดังนั้นจึงจำเป็นต้องใช้เอนไซม์เฉพาะเพื่อขจัดการรบกวนดังกล่าว สำหรับการสกัด RNA คุณภาพสูง จะใช้ DNase I เพื่อขจัด DNA ที่เหลือออกจากตัวอย่าง
DNase I สามารถย่อยสลาย DNA สายคู่และสายเดี่ยวเป็นโอลิโกนิวคลีโอไทด์และนิวคลีโอไทด์เดี่ยวได้ และ DNA ในผลิตภัณฑ์เตรียม RNA ก็สามารถย่อยสลายได้อย่างมีประสิทธิภาพ จากนั้น DNase I จะถูกทำให้ไม่ทำงานโดยการให้ความร้อนด้วยบัฟเฟอร์หยุด ในระหว่างกระบวนการให้ความร้อน โครงสร้างกิ๊บของโมเลกุล RNA จะสามารถเปิดออกได้ ซึ่งทำให้ RNA เข้าสู่กระบวนการถอดรหัสย้อนกลับได้โดยตรง
คุณภาพของ RNA จะส่งผลโดยตรงต่อข้อมูลการทดลองในระดับสูง โดยทั่วไปแล้ว ไม่สามารถหลีกเลี่ยงสารตกค้างของ gDNA ได้อย่างสมบูรณ์ในระหว่างการสกัด RNA ดังนั้นโดยทั่วไปจึงแนะนำให้บำบัดตัวอย่าง RNA ด้วย DNase I เพื่อย่อย gDNA ที่เหลือก่อนการประยุกต์ใช้ในขั้นตอนต่อไป (เช่น การวิเคราะห์การแสดงออกของ mRNA การวิเคราะห์ทรานสคริปโทม เป็นต้น) ขั้นตอนย่อย gDNA สามารถดำเนินการได้ในระหว่างการสกัด RNA หลังการสกัด RNA หรือก่อนการถอดรหัสย้อนกลับของ RNAตามการวางตำแหน่งผลิตภัณฑ์ ผลิตภัณฑ์ที่จัดให้โดย
ตารางที่ 1: รายการผลิตภัณฑ์ที่เกี่ยวข้องกับการกำจัด DNA จากการสกัด RNA หรือ ก่อนการถอดรหัสย้อนกลับ
| การวางตำแหน่งผลิตภัณฑ์ | ชื่อสินค้า | แมว # |
| การสกัด RNA | TRIeasy™ สารสกัด RNA ทั้งหมด [สอบถาม- | 10606ES |
| 19221ES | ||
| ชุด MolPure™ Plant Plus RNA -สอบถาม- | 19292ES | |
| ชุดทดสอบไวรัส DNA/RNA MolPure™ -สอบถาม- | 19321ES | |
| การกำจัด gDNA | 10325ES | |
| การถอดเสียงย้อนกลับ | Hifair™Ⅲ1st Strand cDNA Synthesis SuperMix สำหรับ qPCR (gDNA digester plus) | 11141ES |
| คิวพีซีอาร์ | 11184ES |
3. DNase I สำหรับการถอดรหัสในหลอดทดลองเพื่อกำจัดเทมเพลตดีเอ็นเอ
การถอดรหัสในหลอดทดลอง (In vitro transcription: IVT) จะใช้ DNA เป็นแม่แบบเป็นหลัก บวกกับซับสเตรตและบัฟเฟอร์ที่เกี่ยวข้องเพื่อรับ RNA ผ่านการถอดรหัสในหลอดทดลอง การทดลองถอดรหัสในหลอดทดลอง มักใช้ RNA โพลิเมอเรส เช่น T7, T3 และ SP6 สำหรับการสังเคราะห์ RNA RNA ที่สังเคราะห์อาจมีสารตกค้างของ DNA การกำจัดสารตกค้างของ DNA จะเป็นประโยชน์ต่อการพัฒนาการทดลองต่อเนื่อง ตัวอย่างเช่น ในขั้นตอนการพัฒนาวัคซีน mRNA การกำจัดสารตกค้างเป็นขั้นตอนที่สำคัญ ซึ่งสามารถลดความยากของการทำให้บริสุทธิ์ต่อเนื่องและเพิ่มความบริสุทธิ์ของผลิตภัณฑ์ได้ โดยทั่วไป เทมเพลต DNA จะถูกกำจัดโดยใช้ Recombinant DNase I (ปราศจาก RNase)ตามกระบวนการสังเคราะห์ mRNA ผลิตภัณฑ์ที่จัดทำโดย
| กระบวนการสังเคราะห์ mRNA | ชื่อสินค้า | แมว# |
| การเตรียมเทมเพลต | Hieff Canace™ Plus ดีเอ็นเอโพลีเมอเรสที่มีความเที่ยงตรงสูง -สอบถาม- | 10148ES |
| 10922ES | ||
| 10125ES | ||
| ฟันนี่คัท™BsaI -สอบถาม- | 15005ES | |
| ฟันนิคัท™ XbaI -สอบถาม- | 15033ES | |
| BspQI[สอบถาม] -สอบถาม- | 16215ES | |
| การถอดรหัสในหลอดทดลอง | 10623ES | |
| 10624ES | ||
| โพลิเมอเรส RNA T7 (50 U/μL)-สอบถาม- | 10618ES | |
| 10133ES | ||
| 10620ES | ||
| 10621ES | ||
| ลบเทมเพลต DNA | 10611ES | |
| การดัดแปลง mRNA | 10614ES | |
| 10612ES | ||
| 10132ES | ||
| 10619ES | ||
| การทำให้บริสุทธิ์ mRNA | 12602ES |
4. DNase I สำหรับการกำจัด rRNA
ในสิ่งมีชีวิต rRNA มีอยู่มากมายและมีความอนุรักษ์นิยมมาก ซึ่งไม่มีความสำคัญมากนักในการรับข้อมูลทางชีววิทยา ดังนั้น rRNA จึงมักถูกกำจัดออกก่อนในการสร้างคลัง RNA และการจัดลำดับปัจจุบัน วิธีการกำจัด rRNA คือการย่อย RNase H เป็นหลัก ขั้นตอนหลักของการกำจัด rRNA ที่ใช้เอนไซม์แสดงไว้ในรูปที่ 2: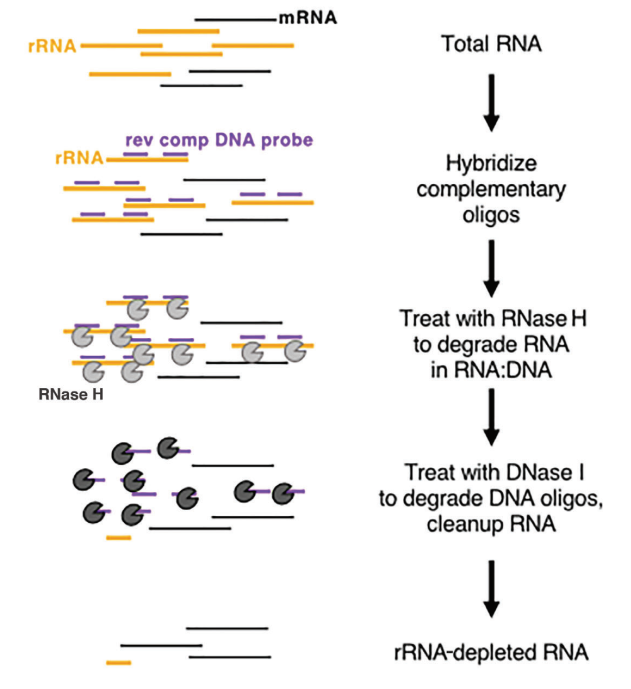
รูปที่ 2: แผนผังหลักการของการกำจัด rRNA ตามเอนไซม์ (Baldwin, A. et al. 2021, Current Protocols)
ขั้นแรก ให้สกัด RNA ทั้งหมด จากนั้นทำไฮบริดไดซ์โพรบ DNA สายเดี่ยวกับ rRNA ออกแบบและสังเคราะห์โพรบ DNA สายเดี่ยวเฉพาะ rRNA จากนั้นใช้ RNase H เพื่อย่อยสลาย rRNA ไฮบริดไดซ์ และใช้ DNase I เพื่อย่อยสลายโพรบ DNA สุดท้าย ให้เหลือเทมเพลต RNA ที่ไม่ใช่ rRNA ผลิตภัณฑ์ที่เกี่ยวข้องกับการกำจัด rRNA จัดทำโดย
| กระบวนการสังเคราะห์ mRNA | ชื่อสินค้า | แมว# |
| มนุษย์/หนู/หนูตะเภา การลดลงของ rRNA | ไฮเอฟเอ็นจีเอส™ ชุด MaxUp rRNA Depletion Kit (มนุษย์/หนู/หนูทดลอง) MaxUp -สอบถาม- | 12253ES |
| การลดลงของ rRNA ในพืช | 12254ES | |
| การกำจัดไรโบโซมอลอาร์เอ็นเอและบริเวณ 45S ITS/ETS จากอาร์เอ็นเอทั้งหมดของมนุษย์ | ไฮเอฟเอ็นจีเอส™ ชุด MaxUp Human rRNA Depletion Kit (rRNA & ITS/ETS) | 12257ES |
| การสลายตัวของ rRNA | 12906ES | |
| การสลายตัวของโพรบ DNA | 10325ES |
5.DNase I สำหรับการติดฉลาก DNA
การแปลรหัสเป็นวิธีการติดฉลากโพรบกรดนิวคลีอิกที่ห้องปฏิบัติการใช้กันทั่วไปที่สุดวิธีหนึ่ง วิธีนี้ใช้กิจกรรมเอนไซม์ต่างๆ ของดีเอ็นเอโพลีเมอเรส I เพื่อรวมดีออกซีไรโบนิวคลีโอไซด์ไตรฟอสเฟตที่ติดฉลากเข้ากับสายดีเอ็นเอที่สังเคราะห์ขึ้นใหม่ ดังนั้น จึงสังเคราะห์โพรบดีเอ็นเอที่ติดฉลากอย่างสม่ำเสมอสำหรับกิจกรรมที่เฉพาะเจาะจงสูง ลักษณะเฉพาะของการแปลรหัสคือ รวดเร็ว เรียบง่าย ตั้งใจ ความจำเพาะสูง และโพรบที่ติดฉลากอย่างสม่ำเสมอ ซึ่งเหมาะสำหรับดีเอ็นเอสายคู่ที่ยาวกว่าวิธีการนี้ทำได้โดยการทำงานร่วมกันของ DNase I และ E. coli DNA Polymerase I ขั้นตอนหลักของการติดฉลาก DNA โดยการแปลนิกแสดงไว้ในรูปที่ 3:
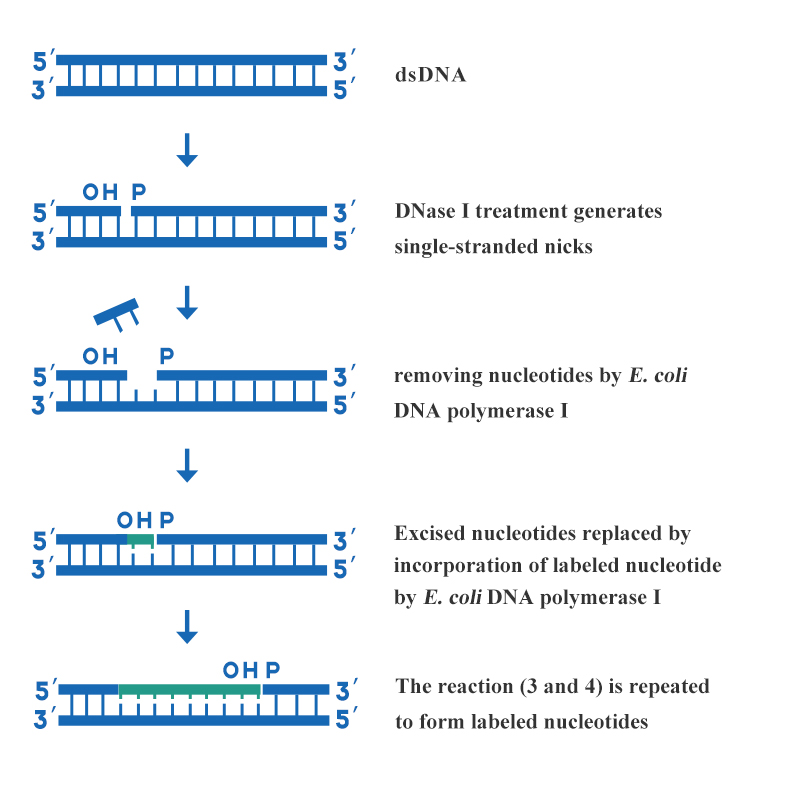
รูปที่ 3: แผนผังการติดฉลาก DNA โดยการแปลนิก
ความเข้มข้นที่เหมาะสมของ DNase I จะถูกใช้เพื่อสร้างช่องว่างสายเดี่ยวหลาย ๆ ช่องบนสาย DNA แต่ละสายที่จะติดฉลาก จากนั้นปลาย 3' ไฮดรอกซิลจะถูกสร้างขึ้นที่ช่องว่างนั้น ใช้การทำงานของเอ็กโซนิวคลีเอส 5'→3' ของ E. coli DNA Polymerase I เพื่อตัดนิวคลีโอไทด์ออกจากปลาย 5' ของรอยบาก และในเวลาเดียวกัน การทำงานของ 5'→3' ของ E. coli DNA Polymerase I จะแนะนำนิวคลีโอไทด์ที่ติดฉลากด้วยปลาย 3' ของช่องว่างเพื่อซ่อมแซมช่องว่าง เมื่อช่องว่างเคลื่อนไปตามสาย DNA นิวคลีโอไทด์ที่ติดฉลากจะถูกผสมเข้ากับสายที่สังเคราะห์ขึ้นใหม่ผลิตภัณฑ์ที่เกี่ยวข้องกับการติดฉลาก DNA จัดทำโดย
| การจัดวางตำแหน่งผลิตภัณฑ์ | ชื่อสินค้า | แมว# |
| สามัญ | ดีออกซีไรโบนิวคลีเอส I (DNase I) จากตับอ่อนของวัว -สอบถาม- | 10607ES/10608ES |
| RNase ฟรี | 10325ES | |
| อีโคไล แหล่งที่มา | 12903ES |
6. การใช้งานอื่น ๆ
ข้างต้นเป็นแอปพลิเคชันที่ใช้กันทั่วไปหลายตัว แอปพลิเคชันเพิ่มเติมของ DNase I ได้แก่ DNase I footprinting assay และ DNase I hypersensitive sites DNase I footprinting assay เป็นวิธีการตรวจจับที่สามารถระบุตำแหน่งการจับของโปรตีนที่จับ DNA บน DNA ได้อย่างแม่นยำ เมื่อโปรตีนจับกับชิ้นส่วน DNA โปรตีนจะปกป้องตำแหน่งการจับไม่ให้ได้รับความเสียหายจาก DNase I และชิ้นส่วน DNA จะถูกทิ้งไว้หลังจากการย่อยด้วยเอนไซม์ ("footprint") และสามารถกำหนดลำดับได้ ในภาพเจล ไม่มีแถบที่ DNA จับกับโปรตีน หากต้องการอ่านเพิ่มเติม คลิกลิงค์นี้ไซต์ที่ไวต่อเอนไซม์ DNase I มากเกินไปหมายถึงการแยกส่วนที่ไซต์เฉพาะจำนวนเล็กน้อยเมื่อโครมาตินได้รับการบำบัดด้วยเอนไซม์ DNase I ต่ำ และไซต์เฉพาะเหล่านี้เรียกว่าไซต์ที่ไวต่อเอนไซม์ DNase I มากเกินไป หลักการคือเมื่อยีนอยู่ในสถานะการถอดรหัสที่ทำงานอยู่ โครมาตินที่มียีนนั้นจะไวต่อการสลายตัวของเอนไซม์ DNase มากกว่าบริเวณที่ไม่ทำงานอย่างมีนัยสำคัญ หากต้องการอ่านเพิ่มเติม โปรดคลิกที่ลิงก์
7. คำแนะนำการเลือกผลิตภัณฑ์ DNase I
| ชื่อสินค้า(Cat#) | การจัดวางตำแหน่งผลิตภัณฑ์ | แอปพลิเคชั่นที่แนะนำ |
| DNase I จากตับอ่อนของวัว (CAT#10607,10608)-สอบถาม- | RNase ถูกถอดออก ไม่ตรวจพบ | ส่วนใหญ่ใช้ในการวิจัยโปรตีน: การกำจัด DNA ออกจากการเตรียมโปรตีน |
| Recombinant DNase I (ปราศจาก RNase)(แมว#10325) | RNase-Free สำหรับการวิจัย | เหมาะอย่างยิ่งสำหรับการใช้งานที่หลากหลาย: การกำจัด DNA ออกจากการเตรียม RNA และโปรตีน เช่น คลัง cDNA ที่ไวต่อ RNase หรือการเตรียมตัวอย่างสำหรับการทดลอง RT-PCR |
| ปราศจาก RNase เกรดเภสัชกรรม GMP | เหมาะอย่างยิ่งสำหรับการใช้งานที่หลากหลาย: การกำจัด DNA ออกจากการเตรียม RNA และโปรตีน เช่น คลัง cDNA ที่ไวต่อ RNase หรือการเตรียมตัวอย่างสำหรับการทดลอง RT-PCR |
ในส่วนของการอ่าน:
สารเคมีเกรด GMP สำหรับการสังเคราะห์ mRNA ในหลอดทดลอง
หลักการของการวางรอยเท้า DNase I และการประยุกต์ใช้ทางชีวการแพทย์
อ้างอิง
1. Baldwin A, Morris AR, Mukherjee N. วิธีการที่ง่าย คุ้มต้นทุน และปรับขนาดได้เพื่อลดปริมาณ RNA ไรโบโซมของมนุษย์สำหรับ RNA-seq[J] Current Protocols, 2021
2. Song C, Zhang S, Huang H. การเลือกวิธีการที่เหมาะสมสำหรับการระบุต้นกำเนิดการจำลองแบบในจีโนมของจุลินทรีย์[J] Frontiers in Microbiology, 2015, 6:1049
