Beskrivelse
Hieff NGS™ DNA-selektionsperler er fremstillet baseret på SPRI-princippet (Solid Phase Reverse Immobilization) og kan anvendes til DNA-oprensning og størrelsesudvælgelse under forberedelsen af næste generations sekventeringsbiblioteker (NGS). Hieff NGS™ DNA Selection Beads er kompatible med forskellige DNA- og RNA-biblioteksforberedelseskit og er et godt alternativ til AMPure perler.
Komponenter
| Komponenter nr. | Navn | 12601ES08 | 12601ES56 | 12601ES75 |
| 12601 | Hieff NGS™ DNA-selektionsperler | 5 ml | 60 ml | 450 ml |
Specifikationer
| Produktlinje | DNA rene og udvalgte perler |
| Udgangsmateriale | DNA |
| Kompatibilitet | DNA |
| Isoleringsteknologi | Magnetisk perle |
| Endelig produkttype | DNA |
| Til brug med (applikation) | DNA celan up, DNA størrelse selektion |
Forsendelse og opbevaring
Perlerne sendes med isposer og kan opbevares ved 2°C-8°C i et år.
Instruktioner
- 1. Forberedelse
Udlign udvælgelsesperlerne ved stuetemperatur i mindst 30 minutter før brug.
- 2. Valg af DNA-størrelse
Operationsflowet for størrelsesvalg er vist i figur 1, og protokollen er som følger.
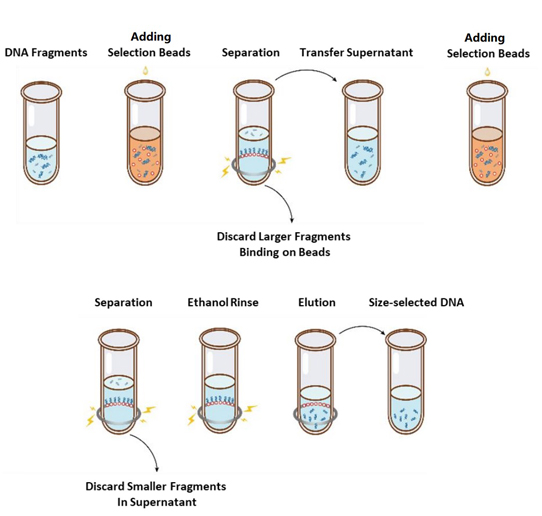
Figur 1. Flowchartet for DNA-størrelsesudvælgelse
2.1 Bland perlerne grundigt ved at vortexe eller pipettere op og ned hver gang før brug.
2.2 Tilføj den første runde af udvælgelsesperler til prøven (se tabel 1). Bland grundigt ved at vortexe eller pipettere op og ned mindst 10 gange.
2.3 Inkuber ved stuetemperatur i 5 min.
2.4 Drej røret kort ned og anbring det på magnetisk stativ. Når opløsningen er klar (ca. 5 min), overføres supernatanten til et nyt PCR-rør.
2.5 Tilføj den anden runde af selektionsperler til prøven fra trin 2.4 i henhold til tabel 1. Bland grundigt ved at vortexe eller pipettere op og ned mindst 10 gange.
2.6 Inkuber ved stuetemperatur i 5 min.
2.7 Drej røret kort ned og placer det på magnetisk stativ. Når opløsningen er klar (ca. 5 min), aspireres supernatanten og kasseres.
2.8 Hold røret i det magnetiske stativ og tilsæt 200 μL frisklavet 80 % ethanol til uden at forstyrre perlerne, inkuber ved stuetemperatur i 30 sek. Aspirer ethanolen og kasser den.
2.9 Gentag trin 2.8 én gang for i alt to vaske.
2.10 Fjern resterende ethanol med 10 µL pipettespidser. Hold røret i det magnetiske stativ, lufttørre udvælgelsesperlerne med låget åbent, indtil der lige er revner (ca. 5 min.).
Bemærk:Tør ikke udvalgsperlerne for meget. Dette kan resultere i et lavere gendannelses-DNA-mål.
2.11 Fjern røret fra det magnetiske stativ. Tilsæt en passende mængde ddH2O (≥20 µL) og bland grundigt ved at vortexe eller pipettere op og ned mindst 10 gange.
2.12 Inkuber ved stuetemperatur i 5 min.
Drej røret kort ned og placer det på det magnetiske stativ. Når opløsningen er klar (ca. 5 minutter), overføres 20 μL af supernatanten til et nyt rør.
- 3. Anbefalede betingelser for valg af DNA-størrelse
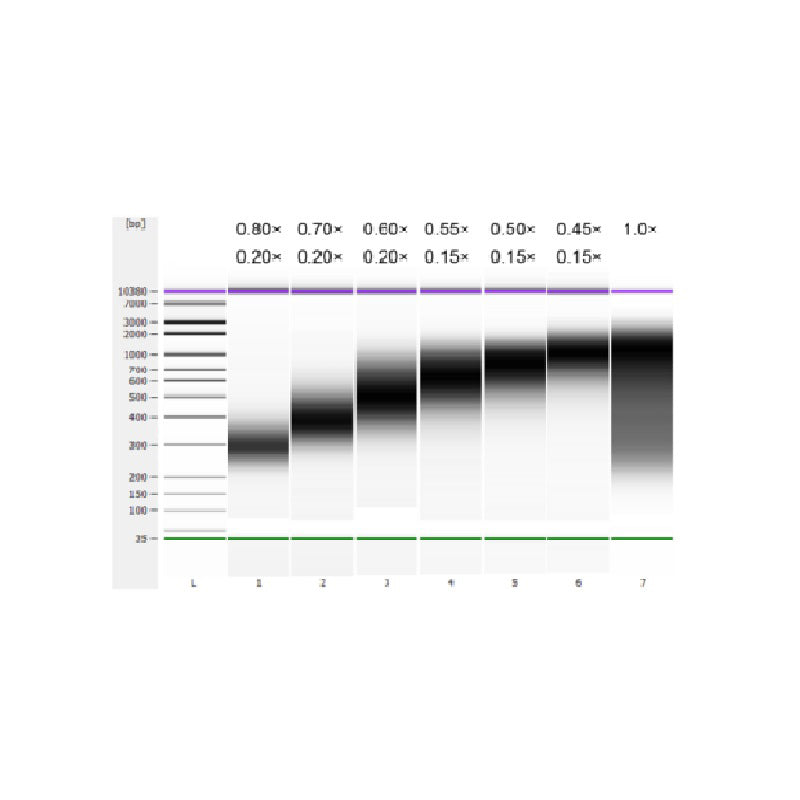
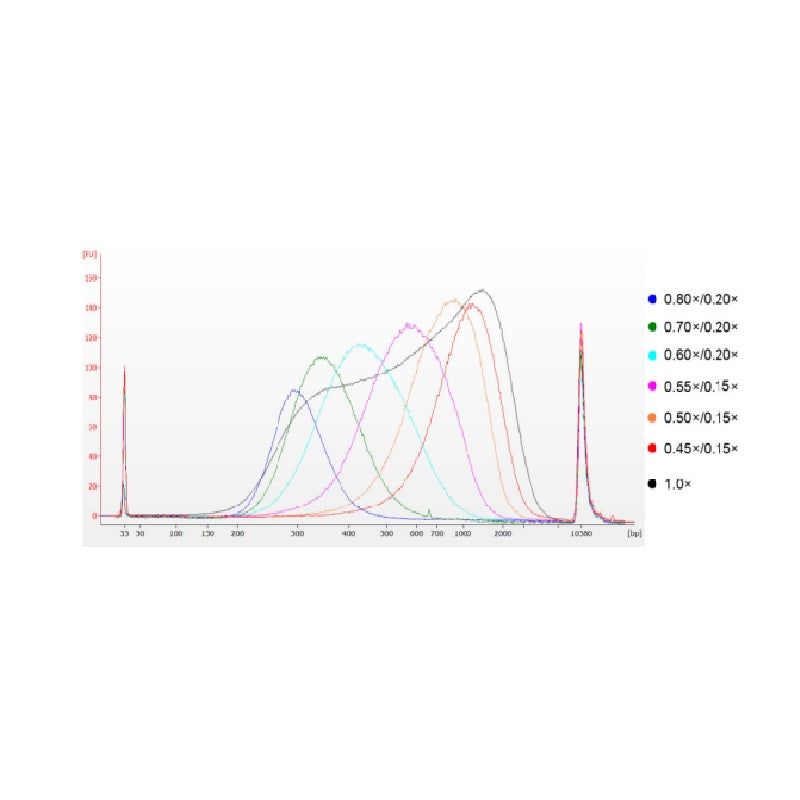
Kalvethymus-DNA'et blev fragmenteret ved sonikering for at fremstille et fragment på 100-1.000 bp, og to runder af størrelsesudvælgelse blev udført i henhold til tabel 1. Resultaterne blev analyseret under anvendelse af Agilent 2100 Bioanalyzer (figur 2).
Tabel 1. Anbefalet betingelse for valg af DNA-størrelse
| Længde af DNA-fragment | 250-350 bp | 320-420 bp | 450-550 bp | 550-700 bp | 700-900 bp | 800-1.000 bp |
| Forholdet mellem perler: DNA til 1. runde | 0,80× | 0,70× | 0,60× | 0,55× | 0,50× | 0,45× |
| Forholdet mellem perler: DNA for 2 Rund | 0,20× | 0,20× | 0,20× | 0,15× | 0,15× | 0,15× |
Bemærk: "×" i tabellen angiver volumen af prøve-DNA. For eksempel, hvis insertlængden af biblioteket er 250 bp, og prøvens DNA-volumen er 100 μL, er volumenet af magnetiske perler, der anvendes i den første sorteringsrunde, 0,80×100 μL=80 μL; volumenet af magnetiske perler brugt i anden sorteringsrunde er 0,20× 100 μL=20 μL.
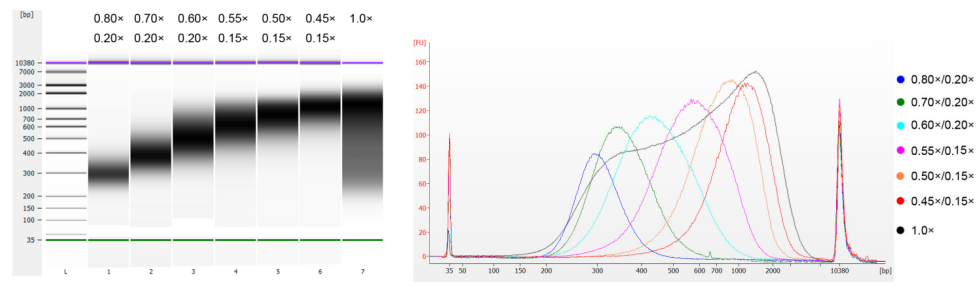
Figur 2. Agilent 2100 højfølsom DNA-chip elektroferogram
Bemærkninger:
1. For din sikkerhed og sundhed skal du bære laboratoriefrakker og engangshandsker til betjening.

Citeret fra "Sekvensspecifik integration af familie 1 casposase fra Candidatus Nitrosopumilus koreensis AR1. Nucleic Acids Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725"

Citeret fra "Nylig infektion af Wolbachia ændrer mikrobielle samfund i vilde Laodelphax striatellus-populationer. Mikrobiom. 2020;8(1):104. Udgivet 2020. jul. doi:10.1186/s40168-020-00878-x"
[1] Wang X, Yuan Q, Zhang W, et al. Sekvensspecifik integration af familie 1 casposase fra Candidatus Nitrosopumilus koreensis AR1. Nucleic Acids Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725(IF:16.971)
[2] Duan XZ, Sun JT, Wang LT, et al. Nylig infektion af Wolbachia ændrer mikrobielle samfund i vilde Laodelphax striatellus-populationer. Mikrobiom. 2020;8(1):104. Udgivet 2. juli 2020. doi:10.1186/s40168-020-00878-x(IF:11.607)
[3] Sang B, Almatrafi E, Sang F, et al.Håndtering af Fenton-behandlet sediment med biochar og fåregødningskompost: Effekter på bakteriesamfundets evolutionære karakteristika. J Environ Administrer. 2022;316:115218. doi:10.1016/j.jenvman.2022.115218(IF:6.789)
[4] Huang C, Mei Q, Lou L, et al. Colitis ulcerosa som svar på fækal mikrobiotatransplantation via modulering af tarmmikrobiota og Th17/Treg cellebalance. Celler. 2022;11(11):1851. Udgivet 5. juni 2022. doi:10.3390/cells11111851(IF:6.600)
[5] Ghosh S, Yang X, Wang L, Zhang C, Zhao L. Aktiv fase af præbiotisk fodring ændrer tarmmikrobiota, inducerer vægtuafhængig lindring af hepatisk steatose og serumkolesterol hos mus med højt fedtindhold. Comput Struct Biotechnol J. 2020;19:448-458. Udgivet 24. december 2020. doi:10.1016/j.csbj.2020.12.011(IF:6.018)
[6] Gao X, Yu B, Yu J, et al. Udviklingsprofilering af diætkulhydratfordøjelse hos smågrise. Front Microbiol. 2022;13:896660. Udgivet 2022 29. april doi:10.3389/fmicb.2022.896660(IF:5.640)
[7] Li P, Zhang Y, Yan F, Zhou X. Karakteristika for en bakteriophag, vB_Kox_ZX8, isoleret fra klinisk Klebsiella oxytoca og dens terapeutiske virkning på musbakteriæmi. Front Microbiol. 2021;12:763136. Udgivet 3. december 2021. doi:10.3389/fmicb.2021.763136(IF:5.640)
[8] Lin Z, Luo P, Huang D, Wu Y, Li F, Liu H. Multi-omics baseret strategi for toksicitetsanalyse af akrylamid i Saccharomyces cerevisiae model. Chem Biol Interact. 2021;349:109682. doi:10.1016/j.cbi.2021.109682(IF:5.194)
[9] Sun X, Lv W, Wang Y, et al. Mrgprb2-genet spiller en rolle i de anafylaktoide reaktioner induceret af Houttuynia cordata-injektion. J Ethnopharmacol. 2022;289:115053. doi:10.1016/j.jep.2022.115053(IF:4.360)
[10] Ma H, Lai B, Zan C, Di X, Zhu X, Wang K. GLO1 bidrager til lægemiddelresistens af Escherichia coli gennem inducering af PER-type af udvidet spektrum β-laktamaser. Infect Drug Resist. 2022;15:1573-1586. Udgivet 5. april 2022. doi:10.2147/IDR.S358578(IF:4.003)
[11] Zhong Y, Zhao W, Tang Z, et al. Komparativ transkriptomisk analyse af de forskellige udviklingsstadier af æggestokken hos rød sumpkrebs Procambarus clarkii. BMC Genomics. 2021;22(1):199. Udgivet 2021 21. marts doi:10.1186/s12864-021-07537-x(IF:3.969)
[12] Lian C, Yang H, Lan J, et al. Sammenlignende analyse af kloroplastgenomer afslører fylogenetiske forhold og intraspecifik variation i lægeplanten Isodon rubescens. PLoS One. 2022;17(4):e0266546. Udgivet 6. april 2022. doi:10.1371/journal.pone.0266546(IF:3.240)
[13] Diao G, Huang J, Zheng X, et al. Prostaglandin E2 tjener en dobbelt rolle i reguleringen af migrationen af dendritiske celler. Int J Mol Med. 2021;47(1):207-218. doi:10.3892/ijmm.2020.4801(IF:3.098)
[14] Bing XL, Zhao DS, Peng CW, Huang HJ, Hong XY. Ligheder og rumlige variationer af bakterie- og svampesamfund i populationer af markrisplanthopper (Hemiptera: Delphacidae). Insect Sci. 2020;27(5):947-963. doi:10.1111/1744-7917.12782(IF:2.791)
[15] Li X, Zhou S, Zhang J, Zhou Z, Xiong Q. Retningsændringer i tarmbakteriesamfundet hos sorte soldaterflue (Hermetia illucens) larver. Dyr (Basel). 2021;11(12):3475. Udgivet 6. december 2021. doi:10.3390/ani11123475(IF:2.752)
[16] Yang J, Peng Y, Kong W. Identifikation og komplet genomsekvens af morbærkryptisk virus 1. Arch Virol. 2022;167(2):687-690. doi:10.1007/s00705-021-05350-1(IF:2.574)
[17] Chang Y, Xia X, Sui L, et al. Endofytisk kolonisering af entomopatogene svampe øger plantesygdomsresistens ved at ændre det endofytiske bakteriesamfund. J Basic Microbiol. 2021;61(12):1098-1112. doi:10.1002/jobm.202100494(IF:2.281)
[18] Ding CY, Ma YM, Li B, et al. Identifikation og funktionel analyse af differentielt udtrykte gener i Myzus persicae (Hemiptera: Aphididae) som svar på trans-anethol. J Insect Sci. 2022;22(1):3. doi:10.1093/jisesa/ieab094(IF:1.857)
Betaling og sikkerhed
Dine betalingsoplysninger behandles sikkert. Vi gemmer ikke kreditkortoplysninger og har heller ikke adgang til dine kreditkortoplysninger.
Forespørgsel
Du kan også lide
FAQ
Produktet er kun til forskningsformål og er ikke beregnet til terapeutisk eller diagnostisk brug hos mennesker eller dyr. Produkter og indhold er beskyttet af patenter, varemærker og ophavsrettigheder ejet af Yeasen Biotechnology. Varemærkesymboler angiver oprindelseslandet, ikke nødvendigvis registrering i alle regioner.
Visse applikationer kan kræve yderligere tredjeparts intellektuelle ejendomsrettigheder.
Yeasen er dedikeret til etisk videnskab og mener, at vores forskning bør adressere kritiske spørgsmål og samtidig sikre sikkerhed og etiske standarder.