Beskrivelse
E. coli Værtscelle DNA-restdetektionskit bruges til den kvantitative analyse af E.coli værtscelle-DNA-rest i mellemprøver, halvfabrikata og færdigvarer af forskellige biologiske produkter.
Dette sæt anvender Taqman fluorescerende probe og polymerase chain reaction (PCR) metoden, som har fg niveau minimum detektionsgrænse og kan specifikt og hurtigt detektere den resterende E. coli celle-DNA. Sættet skal bruges sammen med Residual DNA Sample Preparation Kit (Cat# 18461ES).
Specifikationer
| Kat.nr. | 41308ES50-DA / 41308ES60-EN |
| Størrelse | 50 TI / 100 T-EN |
Komponenter
| Komponenter nr. | Navn | 41308ES50-DA | 41308ES60-EN |
| 41308-A | E. coli qPCR-blanding | 0,75 ml | 1.5 ml |
| 41308-B | E. coli Primer & Probe Mix | 250 μL | 500 μL |
| 41308-C | DNA-fortyndingsbuffer | 2×1,8 ml | 4×1,8 ml |
| 41308-D | E. coli DNA kontrol (30 ng/μL) | 25 μL | 50 μL |
Opbevaring
Dette produkt skal opbevares ved -25~-15 ℃ i 2 år.
Både 41308-A og 41308-B skal opbevares beskyttet mod lys.
Gældende instrumentmodeller
Inkluder, men ikke begrænset til:
Bio-Rad: CFX96 optisk modul.
Thermo Scientific: ABI 7500; ABI Quant Studio 5.
Instruktioner
- E.coli. DNA-standardfortynding og standardkurvepræparation
E.coli DNA-kontrol blev gradientfortyndet ved hjælp af den DNA-fortyndingsbuffer, der medfølger i sættet*og fortyndingen
koncentrationen er 300 pg/μL, 30 pg/μL, 3 pg/μL, 300 fg/μL, 30 fg/μL.
Se detaljerede instruktioner nedenfor:
- Optø coliDNA-kontrollen og DNA-fortyndingsbufferen på is. Efter fuldstændig optøning, vortex forsigtigt for at blande, og centrifuger ved lav hastighed i 10 sekunder.
- Tag seks rene 1,5 ml rør, mærket med Std0, Std1, Std2, Std3, Std4, Std5.
- Tilføj 90 μL DNA-fortyndingsbuffer og 10 μL coliDNA Control til 1,5 mL mikrofugerøret mærket Std0, nemlig fortyndes til 3 ng/μL. Bland og centrifuger derefter i 10 sek. Underpak den fortyndede DNA-standard, og den kan opbevares på kort sigt (ikke mere end 3 måneder) ved -25~-15℃**. Undgå gentagne frys-optøninger.
- Tilføj 90 μL DNA-fortyndingsbuffer i andre rør***, og følg derefter proceduren nedenfor for de serielle fortyndinger****.
| Rør | Fortyndingsforhold | Standardkoncentration |
| Std1 | 10 μL Std0 + 90 μL DNA-fortyndingsbuffer | 300 pg/μL |
| Std2 | 10 μL Std1 + 90 μL DNA-fortyndingsbuffer | 30 pg/μL |
| Std3 | 10 μL Std2 + 90 μL DNA-fortyndingsbuffer | 3 pg/μL |
| Std4 | 10 μL Std3 + 90 μL DNA-fortyndingsbuffer | 300 fg/μL |
| Std5 | 10 μL Std4 + 90 μL DNA-fortyndingsbuffer | 30 fg/μL |
Tabel 1 Standard gradientfortynding
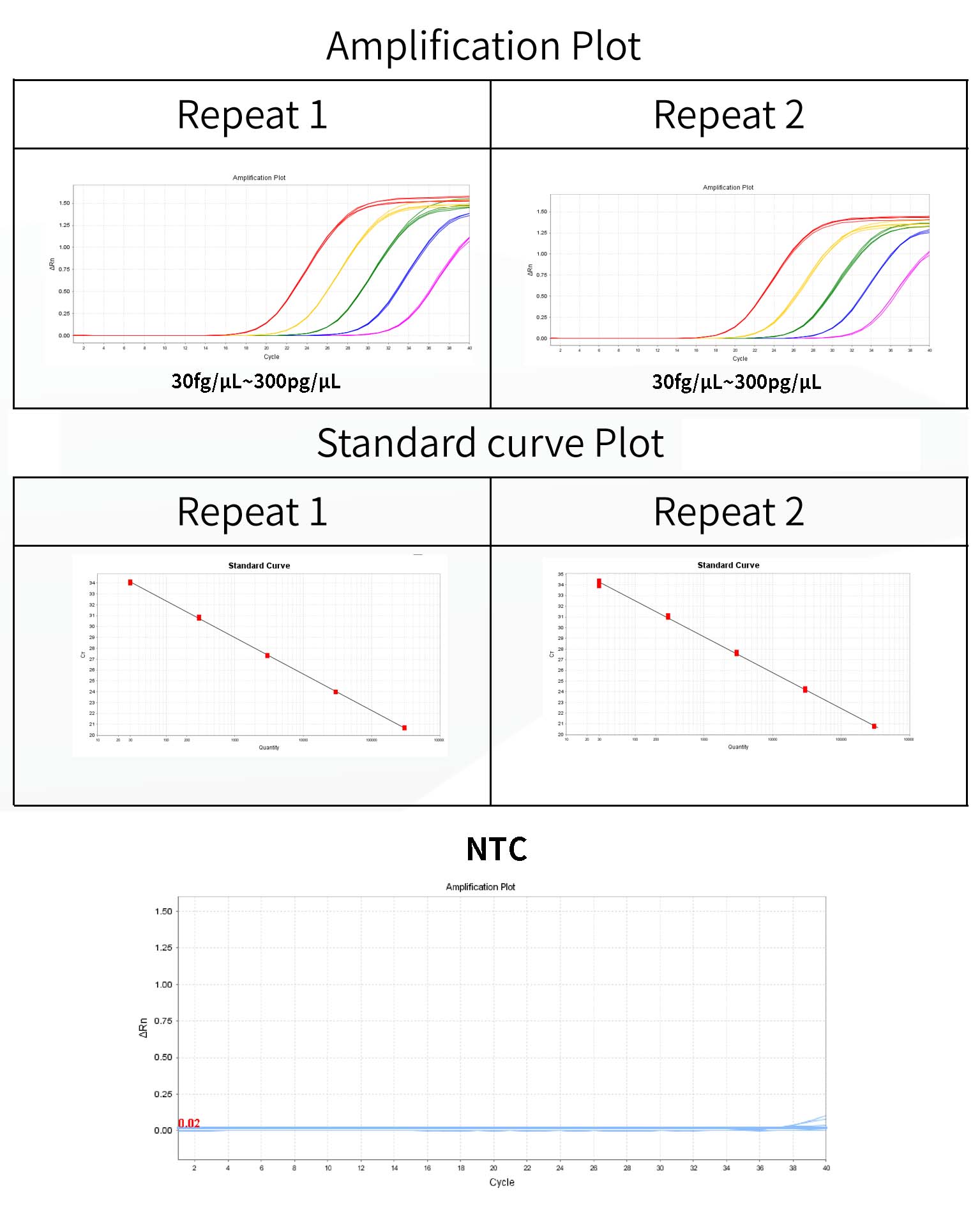
*Tre replikatbrønde er nødvendige for hver koncentration. Detektionsområdet er 30 fg/μL~300pg/μL, og dette område kan udvides.
**For at reducere antallet af gentagne frys-optøninger og undgå kontaminering, anbefales det at opbevare DNA-kontrollen i alikvoter ved -25~-15 ℃ for første gang.
***Når den er optøet, kan DNA-fortyndingsbuffer opbevares ved 2-8°C i 7 dage, hvis den ikke bruges i lang tid, skal den opbevares ved -25~-15℃.
****Sørg for, at skabelonen er fuldstændig blandet, ryst blandingen forsigtigt i 15 sekunder til 1 min for hver gradientfortynding.
- Extraction Recovery Control (ERC) forberedelse
Indstil koncentrationen af E.coli DNA i ERC efter behov (ERC prøven blev forberedt med 30 pg E.coli DNA som eksempel), som følger:
- Tilsæt 100 μL testprøve i et rent 1,5 mL rør, tilsæt derefter 10 μL 3pg/μL E.coli DNA Standard (Std3) og bland godt, markeret som ERC.
- Udfør DNA-ekstraktion af ERC-prøven sammen med testprøverne for at forberede den oprensede ERC-prøve.
- Forberedelse af negativ kontrolopløsning (NCS).
Indstil den negative kontrol i eksperimentet, de specifikke operationstrin er som følger:
1) Tilsæt 100 μL prøvematrix (eller DNA-fortyndingsbuffer) i et rent 1,5 mL rør, og marker derefter som NCS.
2) Udfør DNA-ekstraktion af NCS-prøven sammen med testprøverne for at forberede den oprensede NCS-prøve.
- Ingen skabelonkontrol (NTC) forberedelse
Indstil ingen skabelonkontrol i eksperimentet, de specifikke operationstrin er som følger:
1) NTC kræver ingen prøveforbehandling og kan konfigureres på stadiet med qPCR-detektion af resterende DNA.
2) NTC-prøven i hvert rør eller brønd er 20 μL Mix (dvs. 15 μL E.coli qPCR Mix + 5 μL E.coli Primer&probe Mix) + 10 μL DNA-fortyndingsbuffer. Det anbefales at konfigurere tre replikatbrønde.
- PCR reaktionssystem
| Komponent | Volumen (μL) |
| E. coli qPCR-blanding* | 15 |
| E.coli Primer & Probe Mix | 5 |
| DNA skabelon | 10 |
| Samlet volumen** | 30 |
Tabel 2 Reaktionssystem
*Beregn det samlede PCR-reaktionsvolumen ved antallet af reaktioner: qPCR Mix =(antal reaktioner+2) × (15+5) μL (inklusive tabene af to reaktionsbrønde). Mere end tre replikater for hver prøve anbefales i eksperimentet.
**Efter at have lukket røret eller forseglet pladen, centrifugeres reaktionsrøret eller pladen ved lav hastighed i 10 sekunder. Efter tilstrækkelig omrystning og blanding i 5 sekunder gentages centrifugeringen for at opsamle væsken fra låget eller væggen til bunden. Undgå bobler under drift.
Se nedenstående tabel for den anbefalede pladeopsætning:
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| EN | NTC |
| TS 1 | TS 1 | TS 1 |
| Std 1 | Std 1 | Std 1 |
|
|
|
| B | NTC |
| TS 2 | TS 2 | TS 2 |
| Std 2 | Std 2 | Std 2 |
|
|
|
| C | NTC |
| TS 3 | TS 3 | TS 3 |
| Std 3 | Std 3 | Std 3 |
|
|
|
| D |
|
|
|
|
|
| Std 4 | Std 4 | Std 4 |
|
|
|
| E | NCS |
| ERC 1 | ERC 1 | ERC 1 |
| Std 5 | Std 5 | Std 5 |
|
|
|
| F | NCS |
| ERC 2 | ERC 2 | ERC 2 |
|
|
|
|
|
|
|
| G | NCS |
| ERC 3 | ERC 3 | ERC 3 |
|
|
|
|
|
|
|
| H |
|
|
|
|
|
|
|
|
|
|
|
|
Tabel 3 Computer-on referencekort
Pladelayoutet inkluderer: 5 Std (standardkurven med 5 standardkoncentrationer), 1 NTC (ingen skabelonkontrol), 1 NCS (negativ kontrolopløsning), 3 TS (testprøver), 3 ERC (ekstraktionsgenvindingskontrol).Tre replikatbrønde for hver prøve.
- Opsætning retningslinjer for et PCR-instrument(2-trins metode) (f.eks. Thermo ABI 7500 qPCR-instrument, version 2.0-software)
Følgende instruktioner gælder kun for Thermo ABI 7500 qPCR-instrument (softwareversion 2.0). Hvis du bruger et andet instrument, se den relevante instrumentvejledning for opsætningsvejledninger.
1) Generer et nyt eksperiment, vælg skabelonen for absolut kvantificering eller brugerdefineret.
2) Opret 1 detektionsprobe, kaldet "E.coli-DNA", vælg reporterfluorofor som "FAM" og quench fluorofor som "Ingen". Referencefluorescensen er ROX" (referencefluorescensen kan baseres på instrumentmodellen osv., vælg om du skal tilføje den).
3) I ruden 'Prøver' skal du tilføje alle prøveoplysningerne efter tur. Vælg derefter brøndene, vælg målet og prøverne tilsvarende. Sæt opgaven med E.coli DNA-standard som standard, og tildel værdierne 300000, 30000, 3000, 300, 30 (enheden for DNA-koncentration i hver brønd er fg/μL) i kolonnen Mængde, og navngiv brøndene Std. 1, Std 2, Std 3, Std 4, Std 5, tilsvarende. Indstil NTC's opgave som NTC. Indstil NCS, TS og ERC som ukendt, og navngivet dem i henhold til ovenstående pladelayout tilsvarende. Klik derefter på næste.
4) Indstil amplifikationsprogrammet: Indstil reaktionsvolumen til 30 μL.
| Cyklustrin | Temperatur (℃) | Tid | Cykler |
| Indledende denaturering | 95℃ | 10 min | 1 |
| Denaturering | 95℃ | 15 sek | 40 |
| Udglødning/forlængelse (Fluorescenssamling) | 60℃ | 30 sek |
Tabel 4 Amplifikationsprocedure
- Analyse af qPCR-resultater
1) Systemet vil automatisk give Threshold In Amplification Plot panelet for analyse. Tærsklen givet af systemet er nogle gange for tæt på basislinjen, hvilket resulterer i en stor forskel i Ct mellem replikatbrønde. Du kan manuelt justere tærskelværdien til en passende position og klikke på Analyser. Derefter kan du indledningsvis kontrollere, om amplifikationskurven er normal i Multicomponent Plot.
2) På fanen Resultatanalyse skal du gennemgå standardkurve-plottet. Bekræft værdierne for R2, Effektivitet, Hældning og Y-afskæring. For en normal standardkurve, R²>0,99, 90%≤Eff%≤110%, -3,6≤Slope≤-3,1.
3) I ruden 'Se brøndtabel' i Analyse vises koncentrationerne af hver prøve i mængde, enheden er fg/μL, enhederne kan konverteres i analyserapporten.
4) Parameterindstillingerne for resultatanalysen skal være baseret på den specifikke model og den anvendte softwareversion og kan generelt automatisk fortolkes af instrumentet.
5) Beregn spidsgenvindingsraten baseret på testresultaterne af prøven TS, der skal måles, og prøven spidsgenvindings-ERC, genvindingsgraden for spidser skal være mellem 50%~150%.Formel til måling af spiked recovery rate: Recovery (%) = {Sample spiked assay (eg.pg/μL) - Sample assay (eg.pg/μL)} x Elueringsvolumen (μL) / Teoretisk værdi af DNA-tilsætningsmængde (f.eks. pg) x 100 %.
6) Ct-værdien af den negative kontrol NCS bør være større end gennemsnittet af den laveste koncentration af Ct i standarden.
- Skabelonfri kontrol NTC skal være Ubestemt eller Ct-værdi ≥3
Noter
- Dette produkt er kun til forskningsbrug.
- Brug venligst laboratoriefrakker og engangshandsker for din sikkerhed.
3. Læs venligst denne vejledning omhyggeligt, før du bruger dette reagens, og eksperimentet bør standardiseres, herunder prøvehåndtering, forberedelse af reaktionssystem og prøvetilsætning.
4.Sørg for, at hver komponent er fuldstændig vortexiseret og centrifugeret ved lav hastighed før brug.
Betaling og sikkerhed
Dine betalingsoplysninger behandles sikkert. Vi gemmer ikke kreditkortoplysninger og har heller ikke adgang til dine kreditkortoplysninger.
Forespørgsel
Du kan også lide
FAQ
Produktet er kun til forskningsformål og er ikke beregnet til terapeutisk eller diagnostisk brug hos mennesker eller dyr. Produkter og indhold er beskyttet af patenter, varemærker og ophavsrettigheder ejet af Yeasen Biotechnology. Varemærkesymboler angiver oprindelseslandet, ikke nødvendigvis registrering i alle regioner.
Visse applikationer kan kræve yderligere tredjeparts intellektuelle ejendomsrettigheder.
Yeasen er dedikeret til etisk videnskab og mener, at vores forskning bør adressere kritiske spørgsmål og samtidig sikre sikkerhed og etiske standarder.