¿Has aprendido a construir una biblioteca libre de PCR?
En la secuenciación de alto rendimiento, la construcción de bibliotecas convencionales requiere amplificación por PCR. Por un lado, es para amplificar muestras de ADN traza y aumentar el rendimiento de la biblioteca. Por otro lado, puede amplificar señales fluorescentes, lo que facilita que los secuenciadores capturen e identifiquen señales fluorescentes para mejorar la precisión de la secuenciación. Sin embargo, la PCR es como un "arma de doble filo". Si bien resuelve el problema del bajo volumen de muestra inicial y amplifica la señal fluorescente, también introduce errores y sesgos de amplificación, y no puede presentar perfectamente la "cara verdadera" de la secuencia del genoma. Además, la PCR polimerasa tiene un cierto sesgo de amplificación. Algunas regiones, especialmente las de GC alto o bajo, la estructura secundaria y otras regiones, tienen una baja eficiencia de amplificación y son difíciles de cubrir. También introducirá una gran cantidad de InDels incorrectos y traerá una gran cantidad de duplicaciones, lo que provocará un desperdicio de volumen de datos de ADN y aumentará el costo de la secuenciación. Por lo tanto, la introducción de la tecnología PCR-Free no solo tiene las ventajas de la PCR, sino que también supera las deficiencias de la PCR. No solo puede brindar bibliotecas de alta calidad con alta sensibilidad y realizar la detección de muestras traza, sino que también tiene alta precisión y reduce la carga de trabajo de validación de seguimiento para estudios de variantes. Entonces, ¿qué es exactamente la biblioteca PCR-Free y cuáles son sus ventajas?
1. ¿Qué es la biblioteca PCR-Free?
2. ¿Cuáles son las ventajas de la tecnología PCR-Free?
3. Visualización de datos de PCR-Free
4. Preguntas frecuentes
5. Información del producto
6. Respecto a la lectura
1. ¿Qué es la biblioteca PCR-Free?
La tecnología de secuenciación de próxima generación es la tecnología más utilizada en la investigación de secuenciación de alto rendimiento en la biología molecular moderna. La secuenciación tradicional de primera generación ya no puede satisfacer por completo las necesidades de los investigadores. La resecuenciación del genoma de organismos modelo y la secuenciación del genoma de organismos no modelo requieren un mayor costo. Tecnología de secuenciación de bajo rendimiento, mayor velocidad, dando origen a la tecnología de secuenciación de segunda generación. El principio central de la tecnología de secuenciación de segunda generación es la secuenciación por síntesis, es decir, determinar la secuencia de ADN capturando la información de base etiquetada en el extremo recién sintetizada. El principio principal de la tecnología de secuenciación de segunda generación para la secuenciación de ADN es fragmentar primero el ADN, reparar el extremo del ADN fragmentado y luego agregar adaptadores específicos en ambos lados y luego usar diferentes métodos para generar millones de PCR fijadas espacialmente. Para las matrices clonales, los datos de secuenciación se obtienen utilizando hibridación de cebadores y reacciones de extensión enzimática para obtener imágenes de las etiquetas fluorescentes incorporadas por cada reacción de extensión.
La secuenciación de ADN mediante secuenciación de próxima generación incluye principalmente dos procesos: preparación de la biblioteca y secuenciación en máquina. Durante la preparación de la biblioteca, los fragmentos genómicos interrumpidos aleatoriamente se amplifican típicamente mediante PCR estándar. Sin embargo, para algunas plantillas especiales, existen factores como la estructura secundaria compleja o la mala estabilidad térmica, que afectan la preferencia de amplificación de la PCR de plantilla, por lo que no todas las secuencias genómicas pueden reflejarse por igual en la biblioteca de amplificación por PCR. Especialmente para algunas plantillas con alto contenido de GC o AT, a veces es difícil utilizar el método de PCR para amplificar para la construcción de la biblioteca. No hay diferencia entre la biblioteca sin PCR y la biblioteca de PCR convencional cuando se secuencia en la máquina, excepto que la PCR no se realiza durante el proceso de construcción de la biblioteca. La biblioteca sin PCR puede mejorar teóricamente la distribución de las lecturas de datos y generar una cobertura genómica más uniforme.La biblioteca sin PCR es un complemento del método de construcción de bibliotecas. Dado que se prepara directamente en una biblioteca incorporada sin amplificación por PCR, puede mejorar la cobertura de algunas regiones con alto contenido de GC o AT, lo que reduce las bases de error introducidas por PCR, el sesgo de datos y la repetición de secuencias.
Los pasos básicos de la construcción de bibliotecas NGS convencionales son la fragmentación del genoma, la reparación de extremos, la adición de adaptadores, la PCR y la amplificación de señales antes de la secuenciación. Entonces, ¿qué pasa con la construcción de bibliotecas sin PCR? Como sugiere el nombre, es un proceso de construcción de bibliotecas que no requiere PCR. Los pasos básicos de la construcción de bibliotecas son la fragmentación del genoma, la modificación de extremos, la adición de enlaces y el control de calidad de la biblioteca. Pero, de hecho, esto solo puede considerarse sin PCR en el proceso de construcción de bibliotecas. El verdadero proceso sin PCR debería ser un proceso desde la construcción de la biblioteca hasta la secuenciación sin amplificación de la biblioteca (como la tecnología de secuenciación de moléculas individuales) o una tecnología de secuenciación sin acumulación de errores de amplificación por PCR (como DNBseq de MGI).

Figura 1. Diagrama de flujo de la construcción de bibliotecas convencionales y de la construcción de bibliotecas sin PCR
2. ¿Cuáles son las ventajas de la tecnología PCR-Free?
En el proceso rutinario de construcción de bibliotecas, la enzima de amplificación puede introducir errores. A través de la amplificación cíclica, estos errores se acumularán y amplificarán, reduciendo la fidelidad de la replicación de la secuencia de ADN. Además, la ADN polimerasa también tiene un cierto sesgo de amplificación, especialmente para regiones con grandes diferencias en el contenido de GC o estructuras secundarias, lo que resulta en una baja eficiencia de amplificación y una uniformidad de cobertura deficiente; mientras que la introducción de InDels incorrectos, también traerá una gran cantidad de duplicación que provoca el desperdicio de volumen de datos de ADN y aumenta los costos de secuenciación. La tecnología PCR-Free puede evitar bien los problemas mencionados anteriormente introducidos por la amplificación por PCR en la construcción de bibliotecas convencionales. En el proceso de construcción y secuenciación de bibliotecas, si hay un proceso de amplificación por PCR, tendrá un mayor impacto en la uniformidad de la cobertura del genoma. Para las plantillas de ADN, algunas tienen estructuras secundarias complejas y algunas tienen grandes diferencias en la estabilidad térmica. Estos factores afectarán la eficiencia de la amplificación por PCR. Por lo tanto, en el proceso de amplificación por PCR, no se puede garantizar que todos los fragmentos genómicos puedan obtener la misma eficiencia de amplificación, pero existe un sesgo de amplificación obvio, como una baja cobertura en regiones de GC alto o de GC bajo del genoma. Sin embargo, no hay PCR en todo el proceso de secuenciación libre de PCR. En comparación con la construcción de la biblioteca de PCR, se mejora la cobertura de las regiones de GC alto y las regiones de repetición AT/TA del genoma. Las ventajas específicas son las siguientes.
2.1 Proceso de preparación de bibliotecas simplificado, ahorrando tiempo
La preparación de la biblioteca sin PCR elimina la necesidad de amplificación por PCR y otros pasos, lo que agiliza el proceso de preparación de la biblioteca y ahorra tiempo.
2.2 Reducción de los costes de preparación de la biblioteca
En el proceso de preparación de bibliotecas convencional, se utilizan enzimas costosas de alta fidelidad para la amplificación por PCR a fin de garantizar la autenticidad de la secuencia. Al mismo tiempo, PCR-Free elimina el paso de amplificación por PCR, lo que reduce los costos de preparación de bibliotecas.
2.3 Reducir el sesgo de amplificación
Las enzimas de amplificación de ADN tienen ciertos sesgos de amplificación, especialmente en el caso de plantillas con diferencias significativas en el contenido de GC o la estructura secundaria. Por lo tanto, PCR-Free puede evitar eficazmente el sesgo de amplificación causado por la polimerasa.
2.4 No hay duplicación de PCR
PCR-Free puede reducir las lecturas duplicadas y aumentar las tasas de mapeo único y la utilización efectiva de datos.
2.5 Reducción de errores
Es más probable que la amplificación por PCR introduzca errores de replicación de inserción y deleción, lo que resulta en una mayor tasa de error de secuenciación de indel, mientras que la PCR libre puede evitar eficazmente la generación y acumulación de dichos errores.
3. Visualización de datos de PCR-Free
Kit de preparación de biblioteca de ADN libre de PCR Hieff NGS™ Ultima Pro V2 (Cat# 12196ES) tiene una mejor tasa de conversión de biblioteca que los productos de la competencia.
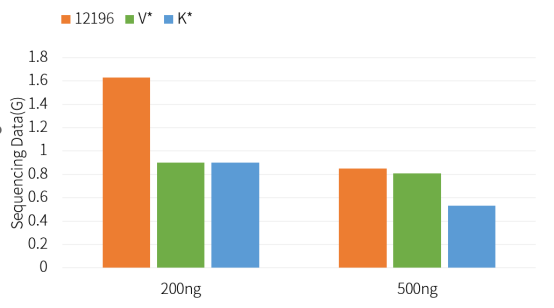
Figura 2. Cantidad de datos de secuenciación fuera de máquina para la construcción de bibliotecas sin PCR
4. Preguntas frecuentes
P: ¿Qué tipos de muestras son adecuadas para bibliotecas sin PCR?
R: Algunas muestras con estructuras secundarias complejas, alta GC y preferencia de PCR pueden elegir una biblioteca sin PCR para construir.
P: ¿Cuál es el tamaño general de los fragmentos de la biblioteca sin PCR?
R: La biblioteca libre de PCR en sí no tiene requisitos especiales en cuanto al tamaño de los fragmentos. La biblioteca de productos de PCR se construye de acuerdo con el tamaño del producto de PCR. Para la biblioteca genómica, se recomienda que sea de alrededor de 350 pb, de modo que la calidad de los datos de la biblioteca sea relativamente buena.
5. Información del producto
El producto que Yeasen puede proporcionar se muestra en la Tabla 1.
Tabla 1. Información del producto
| Producto Nombre | N.º de cat. | Tamaño |
| Kit de preparación de biblioteca de ADN libre de PCR Hieff NGS™ Ultima Pro V2 | 12196ES24/96 | 24/96T |
6. Respecto a la lectura
La solución para la preparación de bibliotecas NGS a partir de muestras de ADN
Sobre la tecnología relacionada con NGS, ¿cuánto sabes?
