Descripción
Hifair™ La Supermezcla de Síntesis de ADNc de 1.ª Hebra III para qPCR (gDNA Digester Plus) es una premezcla reajustada desarrollada con la transcriptasa inversa Hifair™ III. En comparación con la transcriptasa inversa Hifair™ II, la transcriptasa inversa Hifair™ III presenta una estabilidad térmica significativamente mayor y puede soportar temperaturas de reacción de hasta 60 °C. Además, es adecuada para la transcripción inversa de moldes de ARN con estructuras secundarias complejas. Además, la enzima mejora la afinidad con los moldes, lo que resulta ideal para la transcripción inversa de un número reducido de moldes y genes con bajo número de copias.
La premezcla contiene 5×gDNA Digester Mix y 4×Hifair™ Ⅲ SuperMix Plus. La mezcla digestora de ADNg 5×gDNA elimina la contaminación residual del ADN genómico en las plantillas de ARN para garantizar resultados de seguimiento más fiables. 4×Hifair™ Ⅲ SuperMix Plus contiene todos los componentes necesarios para la transcriptasa inversa (tampón, dNTP, Hifair™ Ⅲ Se añaden la transcriptasa inversa, el inhibidor de la ARNasa, la mezcla de cebadores aleatorios/oligo (dT)18, la plantilla de ARN y H2O sin ARNasa a la transcripción inversa y, simultáneamente, se termina el digestor de ADNg para garantizar la integridad del ADNc.
El producto es adecuado para la detección por RT-QPCR en dos pasos, está especialmente optimizado para qPCR y utiliza una mezcla de cebadores aleatorios/oligo (dT)18 proporcionalmente optimizada. Esto permite iniciar la síntesis de ADNc desde todas las regiones del transcrito de ARN con la misma eficiencia de transcripción inversa. La autenticidad y repetibilidad de los resultados de qPCR están garantizadas al máximo. Este producto de transcripción inversa es compatible con los métodos SYBR Green y TaqMan, y con Hifair™. Mezcla maestra UNICON™ qPCR SYBR Green, Mezcla maestra Hifair™ qPCR SYBR Green o Hifair™ La mezcla maestra de sondas qPCR TaqMan se puede seleccionar según fines experimentales y se utiliza en combinación para realizar análisis de expresión genética de alto rendimiento.
Características
- Transcripción inversa de alto rendimiento
- Aumentar la temperatura de reacción: el ADNc de primera cadena se puede sintetizar en un rango de temperatura de 55 a 60 °C.
- Eliminación eficiente de residuos genómicos
- Amplio rango de detección lineal
Aplicaciones
- Construcción de una biblioteca de ADNc
- Síntesis de ARN antisentido
Presupuesto
| Tipo de producto final | ADNc (primera cadena) |
| Método de PCR | RT-PCR |
| Tipo de reactivo | Transcripción inversa |
| Tipo de muestra | ARN |
| Temperatura óptima de reacción | 55℃-60℃ |
| Transcriptasa inversa | M-MLV |
Componentes
| Componentes No. | Nombre | 11141ES10 (10T) | 11141ES60 (100 toneladas) |
| 11141-A | H libre de ARNasa2Oh | 1 ml | 2×1 mL |
| 11141-B | Mezcla digestora de 5 gDNA | 30 μL | 300 μL |
| 11141-C | 4×Hifair™ Ⅲ SuperMix plus | 50 μL | 500 μL |
Envío y almacenamiento
El producto se envía con hielo seco y se puede almacenar a una temperatura entre -15 °C y -25 °C durante 18 meses.
Cifras
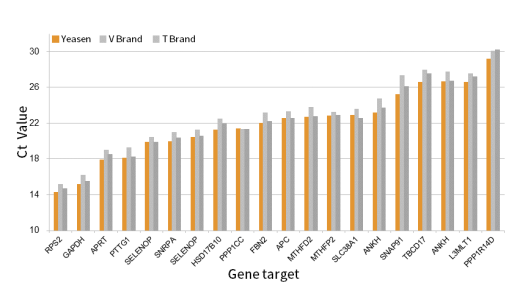
Figura 1. Excelente eficiencia de transcripción inversa, adecuada para genes con diferentes abundancias de expresión y diferentes contenidos de GC.
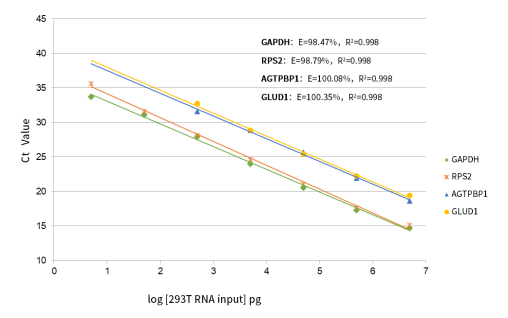
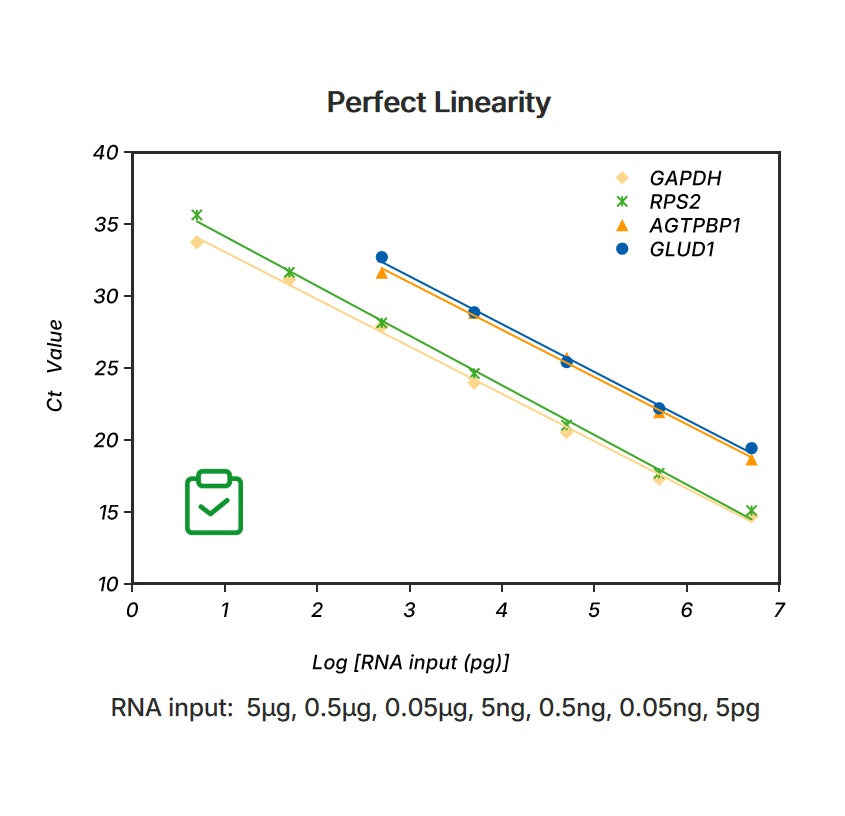
Figura 2.Rango de ensayo lineal ARN total 5 pg ~ 5 μg
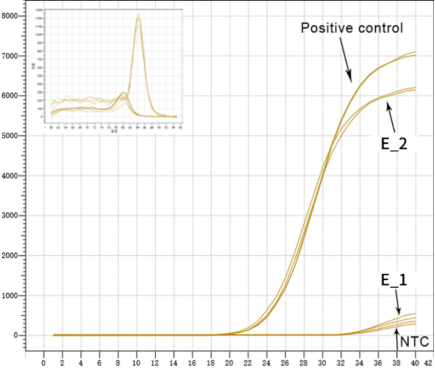
Figura 3. Eliminación eficiente de residuos genómicos

Citado de "Una enzima de ácido nucleico treosa que escinde ARN capaz de discriminar mutaciones puntuales. Nat Chem. Marzo de 2022;14(3):350-359. doi: 10.1038/s41557-021-00847-3".
Citado de "La proteína de repetición tetratricopeptídica OsTPR075 promueve el encabezamiento regulando el transporte de florígenos en el arroz". Plant Cell. 27 de septiembre de 2022;34(10):3632-3646. doi: 10.1093/plcell/koac190.
[1] Yuan B, Peng Q, Cheng J, et al. Estructura del complejo de la polimerasa del virus del Ébola. Nature. 2022;610(7931):394-401. doi:10.1038/s41586-022-05271-2(SI:69.504)
[2] Bi Q, Wang C, Cheng G, et al. El PDGFB derivado de la microglía promueve las corrientes neuronales de potasio para suprimir la tonicidad simpática basal y limitar la hipertensión. Inmunidad. 2022;55(8):1466-1482.e9. doi:10.1016/j.immuni.2022.06.018(SI:43.474)
[3] You XJ, Zhang S, Chen JJ, et al. Formación y eliminación de 1,N6-dimetiladenosina en ARN de transferencia de mamíferos. Nucleic Acids Res. 2022;50(17):9858-9872. doi:10.1093/nar/gkac770(SI:19.160)
[4] Ni X, Wu W, Sun X, et al. El análisis de las interacciones entre glioma y macrófagos M2 identifica a Gal-9/Tim-3 como una diana viable contra el glioblastoma sin PTEN. Sci Adv. 2022;8(27):eabl5165. doi:10.1126/sciadv.abl5165(SI:14.957)
[5] Chen Y, Shen J, Zhang L, et al. La translocación nuclear de OsMFT1, inhibida por OsFTIP1, promueve la tolerancia a la sequía en el arroz. Mol Plant. 2021;14(8):1297-1311. doi:10.1016/j.molp.2021.05.001(SI:13.164)
[6] Yang L, Chen Y, Xu L, et al. El módulo OsFTIP6-OsHB22-OsMYBR57 regula la respuesta a la sequía en el arroz. Mol Plant. 2022;15(7):1227-1242. doi:10.1016/j.molp.2022.06.003(SI:13.164)
[7] Song JM, Xie WZ, Wang S, et al. Dos genomas de referencia sin gaps y una visión global de la arquitectura del centrómero en el arroz. Mol Plant. 2021;14(10):1757-1767. doi:10.1016/j.molp.2021.06.018(SI:13.164)
[8] Lin QY, Zhang YL, Bai J, Liu JQ, Li HH. El eje VEGF-C/VEGFR-3 protege contra la disfunción cardíaca inducida por sobrecarga de presión mediante la regulación de la linfangiogénesis. Clin Transl Med. 2021;11(3):e374. doi:10.1002/ctm2.374(SI:11.492)
[9] Zhang L, Zhang F, Zhou X, et al. La proteína de repetición tetratricopeptídica OsTPR075 promueve el encabezamiento mediante la regulación del transporte de florígenos en el arroz [publicado en línea antes de su impresión, 28 de junio de 2022]. Plant Cell. 2022;koac190. doi:10.1093/plcell/koac190(SI:11.277)
[10] Mao Z, Xiao H, Shen P, et al. KRAS(G12D) puede ser diana de inhibidores potentes mediante la formación de un puente salino. Cell Discov. 2022;8(1):5. Publicado el 25 de enero de 2022. doi:10.1038/s41421-021-00368-w(SI:10.849)
[11] Li M, Chen J, Liu Y, et al. Diseño racional del dominio funcional de la ECA2 vectorizado con AAVrh10 para bloquear ampliamente la entrada celular de variantes del SARS-CoV-2. Antiviral Res. 2022;205:105383. doi:10.1016/j.antiviral.2022.105383(SI:10.103)
[12] Luo F, Yang W, Yin M, et al.Un genoma a nivel cromosómico del parásito sanguíneo humano Schistosoma japonicum identifica la base genómica del cambio de hospedador. Cell Rep. 2022;39(1):110638. doi:10.1016/j.celrep.2022.110638(SI:9.423)
[13] Wang X, Lin C, Wu S, et al. La cannabidivarina alivia la neuroinflamación al actuar sobre el correceptor TLR4 MD2 y mejora la analgesia mediada por morfina. Front Immunol. 2022;13:929222. Publicado el 10 de agosto de 2022. doi:10.3389/fimmu.2022.929222(SI:8.786)
[14] Jiang R, Su G, Chen X, et al. La esculetina inhibe la proliferación del cáncer de endometrio y promueve la apoptosis a través de hnRNPA1 para regular negativamente BCLXL y XIAP [publicado en línea antes de su impresión, 1 de septiembre de 2021]. Cancer Lett. 2021;521:308-321. doi:10.1016/j.canlet.2021.08.039(SI:8.679)
Pago y seguridad
Su información de pago se procesa de forma segura. No almacenamos detalles de la tarjeta de crédito ni tenemos acceso a la información de su tarjeta de crédito.
Consulta
También te puede gustar
Preguntas frecuentes
El producto es solo para fines de investigación y no está destinado a uso terapéutico o diagnóstico en humanos o animales. Los productos y el contenido están protegidos por patentes, marcas comerciales y derechos de autor propiedad de Yeasen Biotechnology. Los símbolos de marca comercial indican el país de origen, no necesariamente el registro en todas las regiones.
Algunas aplicaciones pueden requerir derechos de propiedad intelectual adicionales de terceros.
Yeasen se dedica a la ciencia ética y cree que nuestra investigación debe abordar cuestiones críticas al tiempo que garantiza la seguridad y los estándares éticos.