Descripción
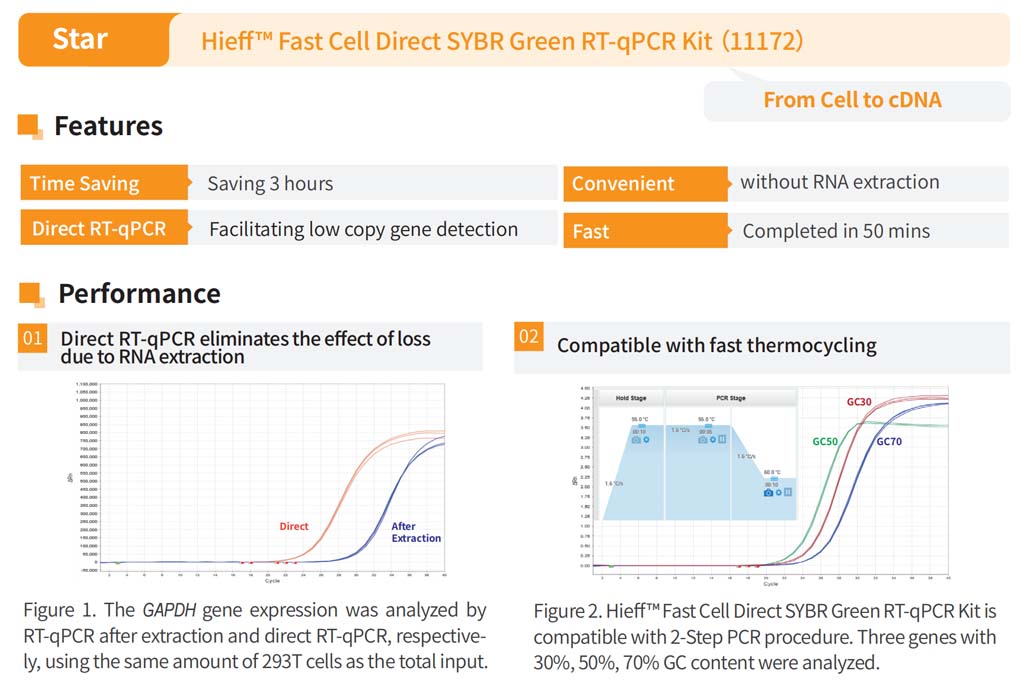
Hieff™ Celda rápida directa El kit RT-qPCR basado en el colorante SYBR Green es adecuado para la extracción de ARN de todo tipo de células animales (como células de la pared celular y células en suspensión, células de cultivo primario, diversas células madre, células iPS, etc.), sin necesidad de extraer ARN, y se puede utilizar directamente para el análisis de expresión de qPCR, que es corto, fácil de operar y tiene una baja tasa de error. Se necesitan solo 1,5 horas para completar los pasos desde la preparación de la plantilla hasta la reacción de transcripción inversa y el análisis de expresión genética.
El kit incluye reactivos de detección de fluorescencia y transcripción inversa, que pueden utilizarse para el análisis de la expresión genética sin necesidad de comprar reactivos adicionales.
Presupuesto
| Cat.No. | 11172ES40 / 11172ES60 |
| Tamaño | 40 T/100 yo |
Componentes
| Componentes No. | Nombre | 11172ES40 | 11172ES60 |
| 11172-A | Tampón de lisis FCD | 2 ml | 5 ml |
| 11172-B | Tampón de lavado FCD | 8 ml | 20 ml |
| 11172-C | Solución de parada de FCD | 100 μl | 250 μL |
| 11172-D | DNasa I | 80 μl | 200 μL |
| 11172-E | Mezcla de 4× Hifair™ FCD RT | 200 microlitros | 500 microlitros |
| 11172-F | Mezcla maestra SYBR qPCR FCD Hieff™ 2× | 2 ml | 5 ml |
| 11172-G | ARNasa libre H2Oh | 2 ml | 5 ml |
Almacenamiento
El tampón de lisis FCD y el tampón de lavado FCD se fundieron y almacenaron a 4 °C para evitar la contaminación. La solución de detención FCD, la DNasa I, la mezcla RT FCD 4× Hifair™ y la mezcla maestra SYBR qPCR FCD 2× Hieff™ deben almacenarse a entre -25 y -15 ℃.
Instrucciones
- Preparación de productos de escisión
1)1. Derretir el reactivo a temperatura ambiente, darle la vuelta y mezclarlo suavemente antes de usarlo, y utilizarlo después de una ligera centrifugación para evitar la formación de espuma*.
*La falta de mezcla de los reactivos, el uso de un oscilador para mezclar y la falta de configuración de los reactivos en hielo pueden provocar una disminución en el rendimiento de la reacción.
2) Dependiendo del tipo de célula**, transfiera las células a un tubo de centrífuga y centrifugue a 5000 rpm durante 2 minutos para recolectar las células y aspirar bien el medio. Si las células se cultivan en placas de 96 pocillos, el medio se puede aspirar directamente.
3) Agregue 150 μL de tampón de lavado FCD a cada pocillo, lave las células soplando, centrifugue a 5000 rpm durante 2 minutos y aspire el tampón de lavado FCD***.
4) Agregue 48 μL de solución tampón de lisis FCD y 2 μL de solución de DNasa Ⅰ a cada pocillo, sople y mezcle a temperatura ambiente, luego deje reposar durante 5 minutos y luego agregue 2.5 μL de solución de detención FCD después de la incubación****, y luego soplar y mezclar aproximadamente 5 veces para obtener el producto de escisión*****.
** El requisito básico para el número de celdas es 1 × 104 células por pocillo, y este kit se puede utilizar en el rango de 1 × 103 - 1 × 106 células. Si el número de células es mayor, la cantidad de solución tampón de lisis FCD y solución de DNasaⅠ se puede aumentar proporcionalmente según corresponda.
*** Las condiciones de centrifugación varían de una célula a otra, por lo que debe centrifugarse a una velocidad adecuada para las células que se utilicen.
**** Agregue 2,5 μL de solución de parada FCD a 50 μL de lisado y aumente la cantidad de solución de parada FCD según sea necesario.
***** Para el almacenamiento a largo plazo de soluciones de productos de lisis celular, colóquelas a -20 °C.
5) Derretir 4 x Hifair™ FCD RT Mix a temperatura ambiente y mezclar con una ligera inversión, colocar en hielo y configurar el sistema de reacción de acuerdo con la siguiente tabla:
| Componentes | Volumen (μL) | Concentración final |
| Mezcla de 4× Hifair™ FCD RT | 5 | 1× |
| producto de escote****** | incógnita | incógnita |
| ARNasa libre H2Oh | Arriba a 20 | - |
*******El nivel de uso recomendado es de 2 a 5 μL, trate de no exceder el 45%.
- Transcripción inversa
Pipetee y mezcle suavemente la solución de reacción preparada anteriormente y realice la reacción de transcripción inversa de acuerdo con el procedimiento de la siguiente tabla:
| Temperatura | Tiempo (minuto) |
| 55℃* | 15 mín. |
| 85℃ | 5 mín. |
* La temperatura recomendada para la transcripción inversa es de 55 °C. Para plantillas con un alto contenido de GC o plantillas complejas, la temperatura de transcripción inversa se puede aumentar a 60 °C. El producto de transcripción inversa se puede utilizar directamente para la detección posterior mediante RT-qPCR. Para evitar la inhibición de la reacción de qPCR por parte del sistema de transcripción inversa y obtener el valor de Ct adecuado (10-35), el producto se puede diluir entre 10 y 1000 veces y luego se puede utilizar. Si no se realizan experimentos posteriores durante un corto período de tiempo, Se puede colocar a -20 ℃ para su almacenamiento.
- PCR cuantitativa de fluorescencia
1) Reacción sistema configuración
Se recomiendan las siguientes proporciones para la preparación de la solución de reacción (preparación en hielo).
| Componentes | Volumen (μL) | Concentración final |
| Mezcla maestra SYBR qPCR FCD Hieff™ 2× | 10 | 1× |
| Cebador directo (10 μmol/L) | 0,4 | 0,2 micromol/L |
| Cebador inverso (10 μmol/L) | 0,4 | 0.2 micromol/L |
| Producto de transcripción inversa* | incógnita | - |
| ARNasa libre H2Oh | Arriba a 20 | - |
*No agregue más de 1/10 del volumen de RT-qPCR del producto de transcripción inversa. Una alta concentración de plantilla puede provocar fácilmente una amplificación no específica. Se recomienda una dilución de 5 a 50 veces. La cantidad recomendada de plantilla es de 4 μL, trate de no exceder los 6 μL. Cuando el rendimiento de la reacción es deficiente, la concentración del cebador se puede ajustar en el rango de 0,2 a 1,0 μmol/L.
2) Procedimiento de amplificación cuantitativa por PCR de fluorescencia (método de dos pasos)
| Ciclo paso | Temperatura. | Tiempo | Ciclos |
| Desnaturalización inicial | 95℃ | 30 segundo | 1 |
| Desnaturalización | 95℃ | 10 segundo | 35-40 |
| Recocido/Extensión* | 60℃ | 30 segundo | |
| Etapa de curva de fusión | Valores predeterminados del instrumento | 1 | |
3) Procedimiento de amplificación rápida para PCR cuantitativa fluorescente (método de dos pasos)
| Ciclo paso | Temperatura. | Tiempo | Ciclos |
| Desnaturalización inicial | 95℃ | 10 segundo | 1 |
| Desnaturalización | 95℃ | 5 segundo | 40 |
| Recocido/Extensión* | 60℃ | 10 segundo | |
| Etapa de curva de fusión | Valores predeterminados del instrumento | 1 | |
* La temperatura de hibridación/extensión y el tiempo de extensión final se pueden ajustar adecuadamente según los requisitos experimentales. El programa rápido es adecuado para la mayoría de los genes, y el programa estándar se puede probar para genes individuales de estructura secundaria compleja.
Notas
- Este producto es solo para uso de investigación.
- Por favor, trabaje con bata de laboratorio y guantes desechables, para su seguridad.
Versión EN20230908
Documentos:
11172-Hieff™ Celda rápida directa. EN20230908.pdf
Pago y seguridad
Su información de pago se procesa de forma segura. No almacenamos detalles de la tarjeta de crédito ni tenemos acceso a la información de su tarjeta de crédito.
Consulta
También te puede gustar
Preguntas frecuentes
El producto es solo para fines de investigación y no está destinado a uso terapéutico o diagnóstico en humanos o animales. Los productos y el contenido están protegidos por patentes, marcas comerciales y derechos de autor propiedad de Yeasen Biotechnology. Los símbolos de marca comercial indican el país de origen, no necesariamente el registro en todas las regiones.
Algunas aplicaciones pueden requerir derechos de propiedad intelectual adicionales de terceros.
Yeasen se dedica a la ciencia ética y cree que nuestra investigación debe abordar cuestiones críticas al tiempo que garantiza la seguridad y los estándares éticos.