Descripción
Alto NGSMT. OnePot Pro DNA Library Prep Kit V3 es un kit de preparación de biblioteca basado en fragmentación enzimática de nueva generación Especialmente desarrollado y diseñado para Ilumina Plataforma de secuenciación &MGI. En comparación con los métodos tradicionales de construcción de bibliotecas, este producto emplea enzimas de fragmentación de alta calidad, lo que elimina el engorroso proceso ultrasónico. Simplifica la operación al combinar los módulos de fragmentación y reparación de extremos en uno solo. Además, las enzimas y el tampón para el módulo de ligadura están premezclados, lo que reduce significativamente el tiempo y el costo de la construcción de bibliotecas. Esto lo hace más adecuado para la construcción automatizada de bibliotecas. Este kit de preparación de biblioteca tiene una excelente tasa de conversión de biblioteca y es aplicable a muestras de todos los animales, plantas, microorganismos, etc. comunes, y también a las muestras FFPE. Sobre la base de la generación anterior del kit de construcción de bibliotecas, este producto exhibe una mayor eficiencia en la fragmentación, Reparación de extremos, colas de dA y ligadura de adaptadores que las versiones anteriores. La enzima de alta fidelidad mejora significativamente la uniformidad y fidelidad de la amplificación.
Presupuesto
| Cat.No. | 12194ES08 / 12194ES24 / 12194ES96 |
| Tamaño | 8 T/24 T/ 96 yo |
Componentes
| Componentes No. | Nombre | 12194ES08 | 12194ES24 | 12194ES96 |
| 12194-A | UntarMT. Buffer 3.0 | 80 μl | 240 μl | 960 μL |
| 12194-B | UntarMT. Enzima 3.0 | 80 μl | 240 μl | 960 μL |
| 12194-C | Mezcla preparada para ligadura | 200 μL | 600 μL | 3×800 μL |
| 12194-D | Mezcla de amplificación HF 2× Ultima | 200 μL | 600 μL | 3×800 μL |
[Nota]: Los componentes del kit son compatibles con ambos Ilumina &MGI plataforma de secuenciación, Si el adaptador completo fue utilizado, Alto NGSMT. Se necesita mezcla de imprimación (Yeasen Cat #12190 o Cat #12191).
Almacenamiento
Este producto debe almacenarse a -25~-15℃ durante 1 año.
Notas
1. Sobre la operación
1. Por favor, trabaje con bata de laboratorio y guantes desechables.,Para su seguridad.
2. Descongele los componentes a temperatura ambiente. Después de descongelar, mezcle bien mediante vórtex, gire el tubo brevemente y colóquelo en hielo para usarlo más tarde.
3. Al preparar la solución de reacción de cada paso, se recomienda utilizar una pipeta para mezclar bien o agitar suavemente. Una agitación vigorosa puede provocar una disminución en la producción de la biblioteca.
4. Se recomienda encarecidamente utilizar puntas de pipeta con filtro para evitar la contaminación cruzada. Asegúrese de cambiar las puntas de pipeta cuando procese distintas muestras.
5. Es muy probable que las operaciones inadecuadas provoquen contaminaciones por aerosoles, lo que afectará la precisión de los resultados. Se recomienda el aislamiento físico obligatorio de las regiones de mezcla de la reacción de PCR y las regiones de ensayo de purificación del producto de PCR. Equipado con equipos como pipetas especializadas para la construcción de bibliotecas. Realice una limpieza de rutina para cada área limpiando las superficies con hipoclorito de sodio al 0,5 % o lejía al 10 %
6. Este producto es solo para uso de investigación.
2. Fragmentación del ADN
1. El kit es compatible con 100 pg - 1000 ng de ADN de entrada. Se recomienda encarecidamente utilizar ADN de entrada de alta calidad con A260/A280 = 1,8-2,0.
2. Los experimentos siguientes podrían verse afectados si se introdujeran altas concentraciones de sales como el agente quelante de metales con el ADN de entrada. Recomendamos eluir la muestra de ADN en DdH2Oh para la fragmentación.
3. Consulte la tabla 6 para el tiempo de fragmentación de muestras de ADN estándar. El kit tiene un bajo sesgo de fragmentación y proporciona una cobertura de GC uniforme para muestras de ADN con una amplia gama de composiciones de GC. Ajuste el tiempo de fragmentación según sus requisitos experimentales.
4. Para una fragmentación precisa, prepare la reacción en hielo.
3. Ligadura del adaptador
1. Los kits de adaptador largo (adaptador con código de barras) Illumina o MGI y los kits de adaptador corto están disponibles para que los clientes elijan según sus requisitos experimentales.
2. Se recomienda seleccionar adaptadores comerciales de alta calidad. Si se seleccionan adaptadores de fabricación propia, confíe en una empresa con experiencia en la síntesis de cebadores NGS y remarque la necesidad de un control estricto de la contaminación. Además, se recomienda preparar la solución de hibridación de ADN en una mesa de trabajo limpia y utilizar solo un tipo de adaptador cada vez para evitar la contaminación cruzada.
3. Descongele los adaptadores en hielo o a 4 °C; cuando se opera a temperatura ambiente, la temperatura del laboratorio no debe superar los 25 °C para evitar que los adaptadores se desnaturalicen.
4. La calidad y la concentración de los adaptadores afectarán directamente la eficiencia de la ligadura y el rendimiento de la biblioteca. Una concentración demasiado alta de adaptadores favorece la formación de dímeros de adaptadores, mientras que una concentración demasiado baja de adaptadores reduce la tasa de ligadura y el rendimiento de la biblioteca. Diluciones correspondientes con tampón TE según la cantidad de ADN de entrada cuando se utiliza el adaptador. Tabla 1-2 enumera los métodos de dilución recomendados para adaptadores convencionales y UMI para diferentes cantidades de ADN de entrada utilizando este kit para las plataformas de secuenciación Illumina o MGI.
Mesa 1 El Illumina recomendado Cantidad de adaptadores para diferentes entradas ADN
| Aporte ADN | doRelación de dilución del adaptador convencional | Concentración | Relación de dilución del adaptador UMI | Concentración |
| <1 ng | 7,5 veces | 2 micrometros | 15 veces | 1 micrometros |
| 1 en ~ 10 ng | 3 veces | 5 micrometros | 3 veces | 5 μM |
| 10 en ~ 200 en | 1,5 veces | 10 micrometros | Doble pliegue | 7,5 μM |
| >200 ng | 0 pliegues | 15 μM | 0 pliegues | 15 μM |
Mesa 2 El recomendado MGI Cantidad de adaptadores para diferentes entradas ADN
| Aporte ADN | doRelación de dilución del adaptador convencional | Concentración | Relación de dilución del adaptador UMI | Concentración |
| <1 ng | 5 veces | 2 micrometros | 10 veces | 1 micrometros |
| 1 en ~ 10 ng | Doble pliegue | 5 micrometros | Doble pliegue | 5 μM |
| 10 en ~ 200 en | 0 pliegues | 10 micrometros | 1.25 pliegues | 8 micrometros |
| >200 ng | 0 pliegues | 10 micrometros | 0 pliegues | 10 micrometros |
4. Limpieza y selección de tamaño de ADN con microesferas
1. La selección del tamaño del ADN se puede realizar antes de la reparación final/cola de dA, después de la ligadura del adaptador o después de la amplificación.
2. Se recomienda realizar la selección de tamaño inmediatamente después de la ligadura del adaptador si la cantidad de ADN de entrada es superior a 50 ng; De lo contrario, realice la selección de tamaño después de la amplificación.
3. El potenciador de ligadura contiene una alta concentración de PEG, lo que puede causar un impacto significativo en la selección precisa del tamaño. Por lo tanto, si la selección del tamaño se va a realizar inmediatamente después de la ligadura del adaptador, se recomienda encarecidamente agregar un paso de limpieza de perlas antes de la selección del tamaño. El paso de selección del tamaño se puede realizar directamente si se realiza antes de la reparación final/cola de dA o después La amplificación de la biblioteca.
4. Las perlas magnéticas deben equilibrarse a temperatura ambiente antes de su uso, de lo contrario, el rendimiento disminuirá y el efecto de selección de tamaño se verá afectado.
5. Las perlas magnéticas deben mezclarse bien mediante vórtex o pipeteo antes de su uso.
6. No aspire las perlas al transferir el sobrenadante; incluso trazas de perlas pueden afectar las siguientes reacciones.
7. El etanol al 80% debe prepararse recién, de lo contrario afectará la eficiencia de recuperación.
8. Para una selección precisa del tamaño, se recomienda comenzar con un volumen de más de 100 μL. Si es menor, se recomienda aumentar el volumen hasta 100 μL con agua ultrapura.
9. Las perlas magnéticas deben secarse a temperatura ambiente antes de eluir el producto. Una sequedad insuficiente puede provocar que los residuos de etanol afecten fácilmente las reacciones posteriores; una sequedad excesiva puede provocar que las perlas magnéticas se agrieten y reduzcan el rendimiento de la purificación. Normalmente, un secado a temperatura ambiente durante 3 a 5 minutos es suficiente para permitir que las perlas se sequen por completo.
10. Si es necesario, las muestras de ADN purificadas o de tamaño seleccionado se eluyen en 0,1× El tampón TE se puede almacenar a 4 °C durante 1-2 semanas o a -20 °C durante un mes.
5. Amplificación de la biblioteca
1. La realización o no de la amplificación de la biblioteca depende de la cantidad de ADN de entrada, los tipos de adaptadores, las aplicaciones de los datos de secuenciación, etc. El paso de amplificación es necesario si se utilizan adaptadores parciales. Cuando se utilizan adaptadores de longitud completa, si el ADN de entrada es <200 ng, se recomienda realizar la amplificación; de lo contrario, no es necesaria.
2. La cantidad de ciclos de amplificación debe controlarse estrictamente. Una amplificación insuficiente puede generar un bajo rendimiento de la biblioteca; la sobreamplificación puede generar mayor sesgo, errores, lecturas duplicadas y productos quiméricos. Tabla 3 enumera los números de ciclos recomendados que apuntan al rendimiento de la biblioteca de 1 μg.
Mesa 3 El número recomendado de ciclos para generar 1.000 ng de rendimiento de la biblioteca
| Entrada de ADN | Número de ciclos necesarios para generar 1 μg de rendimiento de biblioteca |
| 1000-2000 en | 2 - 4 |
| 500 ng | 2 - 4 |
| 250 ng | 4 - 6 |
| 100 ng | 5 - 7 |
| 50 ng | 7 - 9 |
| 10 ng | 9 - 11 |
| 5 ng | 10 - 12 |
| 1 ng | 12 - 15 |
| 100 páginas | 16 - 18 |
Nota
1.Tabla 3 muestra la cantidad de parámetros de bucle utilizando pruebas de ADN de entrada de alta calidad de alrededor de 200 pb. La calidad del ADN FFPE varía mucho y, cuando la calidad del ADN es deficiente o la longitud de la biblioteca es larga, es necesario aumentar adecuadamente la cantidad de ciclos para obtener bibliotecas suficientes.
2. Si se requiere la selección de tamaño durante el proceso de construcción de la biblioteca, se recomienda un número de ciclo más alto para la amplificación de la biblioteca; de lo contrario, se recomienda un número de ciclo más bajo.
3. Si se utilizan adaptadores incompletos, se deben amplificar al menos 2 ciclos para formar un adaptador completo.
6. Análisis de calidad de la biblioteca
1. La calidad de las bibliotecas construidas generalmente se analiza midiendo las concentraciones y distribuciones de tamaño.
2. Las concentraciones de las bibliotecas se pueden medir mediante métodos basados en fluorescencia como Qubit y PicoGreen o qPCR.
3. NO se recomienda utilizar métodos de cuantificación basados en absorbancia como NanoDrop.
4. Se recomienda utilizar el método qPCR para cuantificar las bibliotecas: los métodos basados en fluorescencia como Qubit y PicoGreen no pueden diferenciar las estructuras de dsADN incompletas (insertos sin adaptador o con solo uno de los extremos ligado con adaptador) de las bibliotecas completas. El método qPCR solo amplificará y medirá las bibliotecas completas con ambos extremos ligados con adaptadores (las bibliotecas secuenciables), lo que proporciona una medición más precisa para la carga.
5. La distribución del tamaño de las bibliotecas se puede analizar utilizando Agilent Bioanalyzer u otros dispositivos basados en los principios de electroforesis capilar o microfluídica.
7. Otros materiales
1. Perlas magnéticas para purificación de ADN: Hieff NGSMT. Perlas de selección de ADN (Yeasen Cat. n.° 12601) o AMPure® XP Beads (A63880) u otros productos equivalentes.
2.Adaptadores: Adaptador completo para Illumina: Yeasen Cat#13519-13520; Cebadores CDI dobles 384: Yeasen Gato n.° 12412 ~ Gato n.° 12413; 384 cebadores de índice dual único (UDI): Yeasen Gato n.° 12312 ~ Gato n.° 12315; Adaptadores UMI UDI: Yeasen Cat#13370~Cat#13371; Adaptador completo para MGI: Yeasen Cat#13360-13362. Mezcla de iniciadores de ADN:Gato n.° 12190 o Gato#12191.
3. Análisis de calidad de la biblioteca: Agilent 2100 Bioanalyzer DNA 1000 Chip/High Sensitivity Chip u otros productos equivalentes; reactivos cuantitativos de biblioteca.
4. Otros materiales: etanol absoluto, agua ultrapura estéril, puntas de pipeta de baja retención, tubo de PCR, soportes magnéticos, termociclador, etc.
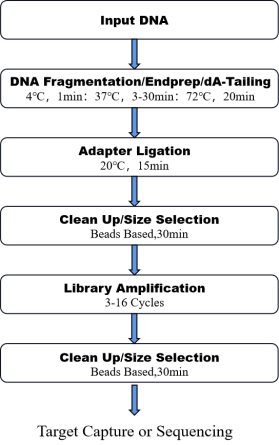
8. Flujo de trabajo
Figura 1. El flujo de trabajo de Una olla Pro ADN Kit de preparación para la biblioteca
Cifras
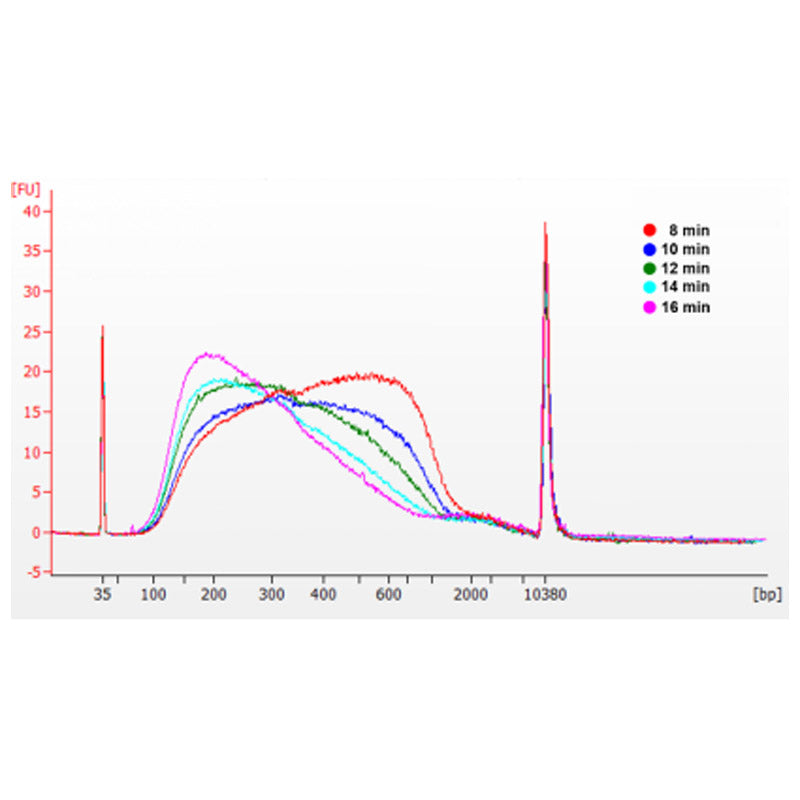
Los tamaños de los fragmentos de inserción obtenidos en diferentes condiciones de fragmentación
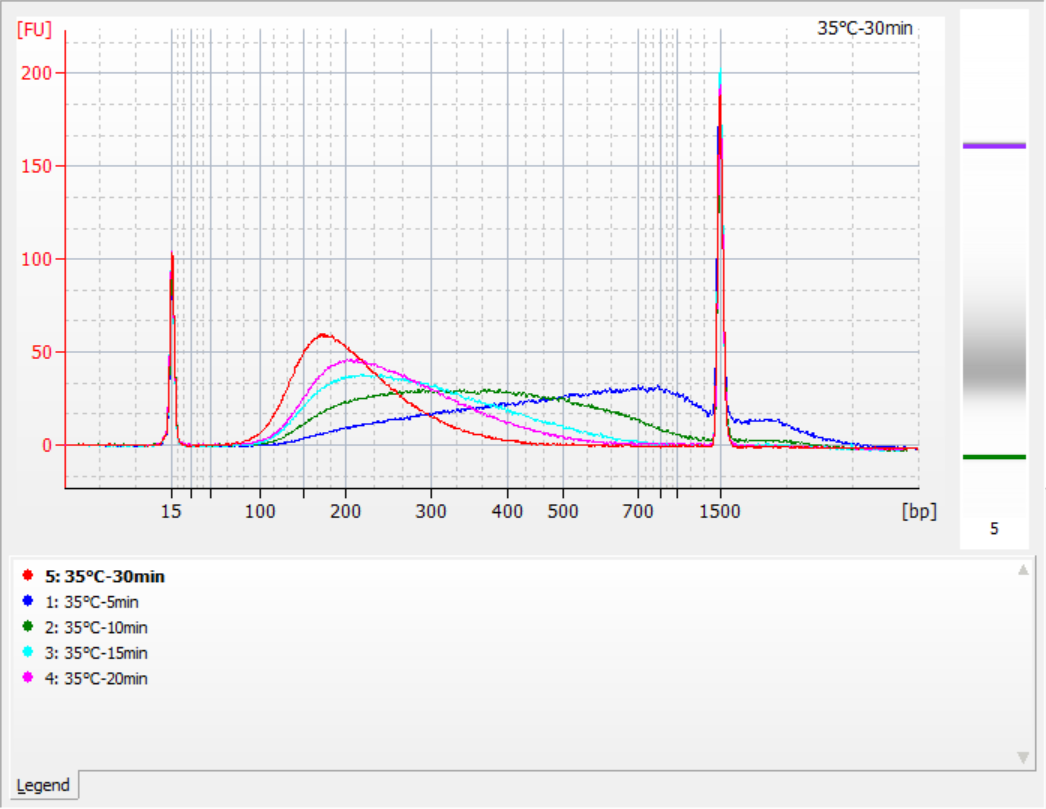
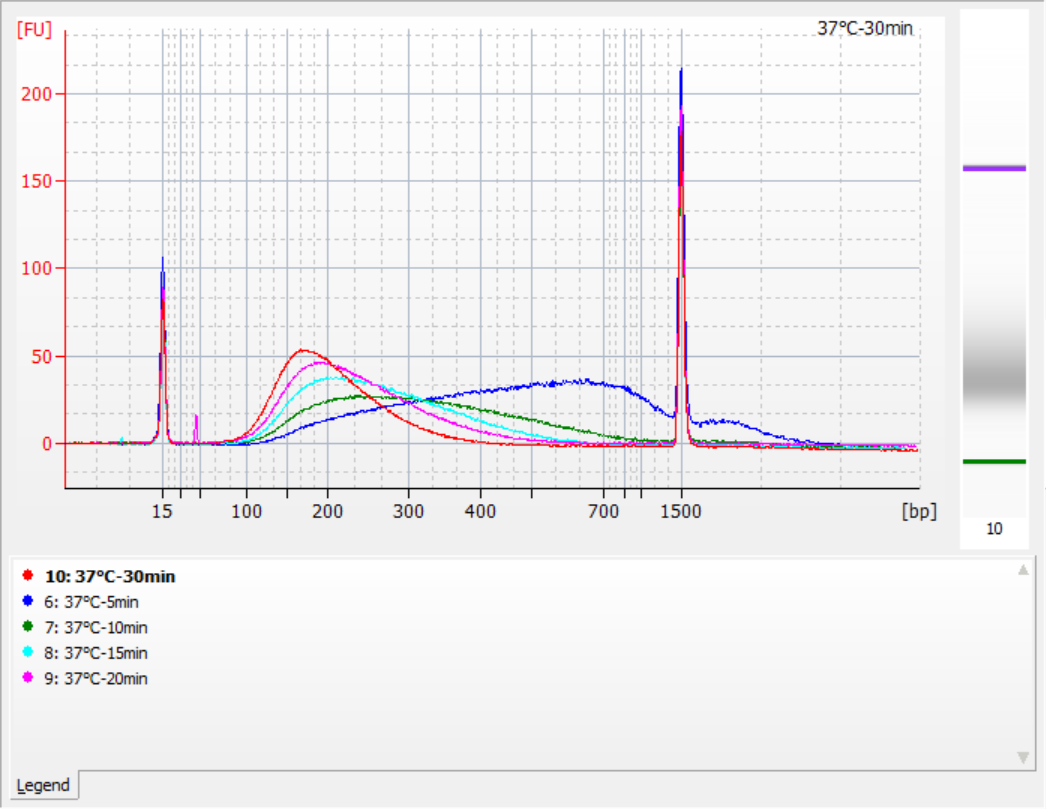
Utilizando 500 ng de ADN genómico estándar como plantilla, se construyeron bibliotecas con este kit. Las condiciones de fragmentación fueron digestión enzimática a 32 °C, 35 °C y 37 °C durante 5, 10, 15, 20 y 30 minutos respectivamente. Los productos fragmentados se purificaron con perlas magnéticas 1.2x y se eluyeron con 21 μL de ddH.2O. La concentración se midió utilizando Qubit y la distribución de los fragmentos de inserción recuperados se muestra en la siguiente figura.
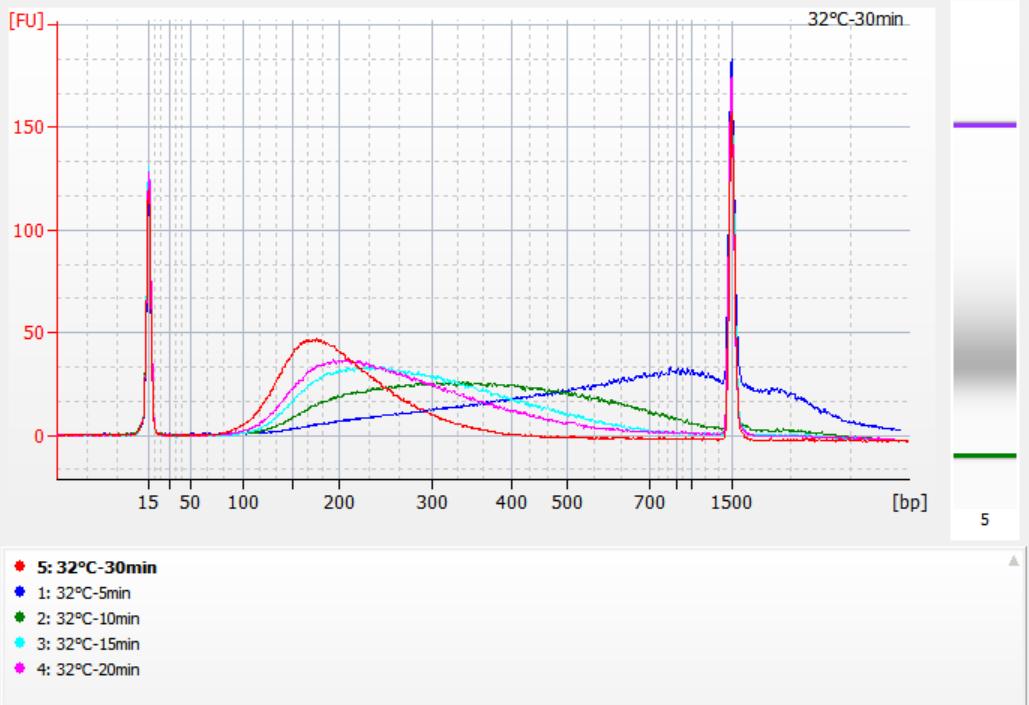 |
Figura 2. Perfiles de la biblioteca a 32 °C para diferentes tiempos de digestión enzimática
 |
 |
Pago y seguridad
Su información de pago se procesa de forma segura. No almacenamos detalles de la tarjeta de crédito ni tenemos acceso a la información de su tarjeta de crédito.
Consulta
También te puede gustar


Preguntas frecuentes
El producto es solo para fines de investigación y no está destinado a uso terapéutico o diagnóstico en humanos o animales. Los productos y el contenido están protegidos por patentes, marcas comerciales y derechos de autor propiedad de Yeasen Biotechnology. Los símbolos de marca comercial indican el país de origen, no necesariamente el registro en todas las regiones.
Algunas aplicaciones pueden requerir derechos de propiedad intelectual adicionales de terceros.
Yeasen se dedica a la ciencia ética y cree que nuestra investigación debe abordar cuestiones críticas al tiempo que garantiza la seguridad y los estándares éticos.