Descripción
Descripción del Producto
El kit de preparación de biblioteca de ARN de modo dual Hieff NGS™ Ultima es un kit de construcción de biblioteca de secuenciación de ARN total para la plataforma de secuenciación Illumina® y MGI®, que incluye reactivos de fragmentación de ARN, reactivos de transcripción inversa, reactivos de síntesis de ADNc de doble cadena convencionales y específicos de la cadena y reactivos de amplificación de biblioteca. La biblioteca de secuenciación se puede construir seguida del kit de purificación de ARNm o del kit de eliminación de ARNr. El módulo de síntesis de dos cadenas está equipado con dos tampones para satisfacer la necesidad de una biblioteca convencional o una biblioteca específica de la cadena. Entre ellos, el dTTP se reemplaza con dUTP en el tampón de síntesis de dos cadenas específico de la cadena, por lo que el dUTP se puede agregar a la segunda cadena de ADNc. La ADN polimerasa de alta fidelidad utilizada en este kit no puede amplificar la plantilla de ADN que contiene uracilo, logrando la especificidad de la cadena. Todos los reactivos proporcionados han sido sometidos a un estricto control de calidad y verificación funcional, lo que garantiza la estabilidad y reproducibilidad de la construcción de la biblioteca en la mayor medida posible.
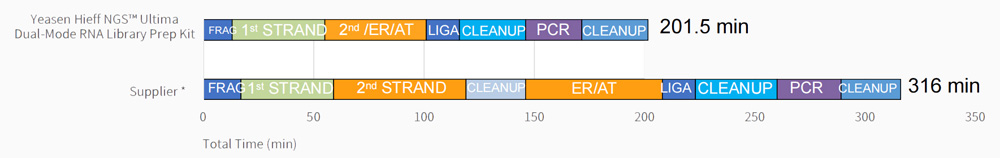
Flujo de trabajo
Componentes del producto
| Componentes | 12308ES24 | 12308ES96 | |||
| 12308-A | 2× Tampón de fragmentación/cebado | 250 μL | 930 μL | ||
| 12308-B | Mezcla de enzimas de primera hebra | 48 μl | 192 μL | ||
| 12308-C | Reactivo de especificidad de cadena | 150 μl | 580 μl | ||
| 12308-D | Tampón de segunda cadena (dNTP) | 720 μL | 2×1440 μL | ||
| 12308-E | Tampón de segunda cadena (dUTP) | 720 μL | 2×1440 μL | ||
| 12308-F | Mezcla maestra de enzimas de segunda hebra | 120 μL | 480 μl | ||
| 12308-G | Potenciador de ligadura | 720 μL | 2×1440 μL | ||
| 12308-H | Nueva ligasa de ADN T4 | 120 μL | 480 μl | ||
| 12308-I | 2×Super Canace® II Mezcla de alta fidelidad | 600 μL | 2×1200 μL | ||
| 12308-K | H2O libre de nucleasas | 300 μL | 1000 μL | ||
Nota: Este kit es compatible con las plataformas Illumina y MGI, pero Illumina adicional o MGI Mezcla de imprimación (n.° de cat. 13335 Mezcla de imprimación para Illumina) y Cat# 13334 Mezcla de imprimación para MGI ) es requerido.
Envío y almacenamiento
Los componentes del kit de preparación de biblioteca de ARNm de modo dual Hieff NGS™ Ultima en la Caja I se envían con bolsas de hielo y se pueden almacenar a 2-8 °C durante un año.
Los componentes del kit de preparación de biblioteca de ARNm de modo dual Hieff NGS™ Ultima en la Caja II se envían con hielo seco y se pueden almacenar a -20 °C durante un año.
Precauciones
1 Operación
1.1 Por su seguridad y salud, utilice equipos de protección personal (EPP), como batas de laboratorio y guantes desechables, cuando trabaje con este producto. ¡Este producto es SOLO para uso en investigación!
1.2 Descongele los componentes a temperatura ambiente. Mezcle bien invirtiéndolos varias veces, centrifugue brevemente y colóquelos en hielo para su uso.
1.3 Se recomienda realizar cada paso de la reacción en un termociclador con tapa calefactada. El termociclador debe precalentarse a la temperatura establecida antes de su uso.
1.4 Es necesario contar con suministros libres de contaminación por ARNasa y limpiar periódicamente el área experimental.
1.5 Las operaciones inadecuadas pueden muy probablemente causar contaminaciones por aerosoles, lo que afecta la precisión del resultado. Se recomienda el aislamiento físico obligatorio de las regiones de mezcla de la reacción de PCR y las regiones de ensayo de purificación del producto de PCR. Equipado con equipos como pipetas especializadas para la construcción de bibliotecas.
2 Ligadura del adaptador
2.1 Los kits de adaptador largo (adaptador con código de barras) Illumina o MGI y los kits de adaptador corto están disponibles para que los clientes elijan según sus requisitos experimentales.
2.2 Se recomienda seleccionar adaptadores comerciales de alta calidad. Si se seleccionan adaptadores de fabricación propia, confíe en una empresa con experiencia en la síntesis de cebadores NGS y remarque la necesidad de un control estricto de la contaminación. Además, se recomienda preparar la solución de hibridación de ADN en una mesa de trabajo limpia y utilizar solo un tipo de adaptador cada vez para evitar la contaminación cruzada.
2.3 Descongele los adaptadores en hielo o a 4 °C; cuando se opera a temperatura ambiente, la temperatura del laboratorio no debe superar los 25 °C para evitar que los adaptadores se desnaturalicen.
2.4 La concentración del adaptador afecta directamente la eficiencia de la ligación y el rendimiento de la biblioteca. El volumen de adaptador añadido al kit se fija en 5 μl. Se recomienda diluir los adaptadores con tampón TE 0,1× y los adaptadores diluidos se pueden almacenar a 4 °C durante 48 horas. En la Tabla 1 se enumera la cantidad de adaptador recomendada para diferentes cantidades de ARN de entrada.
Tabla 1-1 Cantidad recomendada de adaptador Illumina para diferentes ARN de entrada
| Entrada de ARN total | Ilumina Concentración de stock de adaptadores |
| 10 ng | 1 μM |
| 100 ng | 1,5 μM |
| 500 ng | 3 μM |
| ≥1 μg | 5 μM |
Tabla 1-2 Cantidad recomendada de adaptador MGI® para diferentes ARN de entrada
| Entrada de ARN total | MGI Concentración de stock de adaptadores |
| 100-499 en | 2 micrometros |
| 500-4000 ng | 5 μM |
*El uso del adaptador se puede ajustar según los diferentes tipos de muestras de ARN total y la cantidad de entrada.
3 Amplificación de la biblioteca
3.1 Sobre la base de la ADN polimerasa de primera generación, la ADN polimerasa de alta fidelidad del kit ha mejorado enormemente su uniformidad de amplificación y no presenta sesgo de amplificación.
3.2 Si el adaptador indexado (también conocido como adaptador largo o adaptador Y grande) se liga al ADN objetivo, la mezcla de cebadores proporcionada en este kit se puede utilizar para la amplificación; si se utiliza un "adaptador corto" o un "adaptador Y pequeño" para la ligadura del ADN, se necesitan cebadores de índice para la amplificación.
3.3 La cantidad de ciclos de amplificación debe controlarse estrictamente. Una amplificación insuficiente puede dar lugar a un bajo rendimiento de la biblioteca; la sobreamplificación puede generar mayor sesgo, errores, lecturas duplicadas, productos quiméricos y acumulación de mutaciones de expansión. La Tabla 2 enumera la cantidad de ciclos recomendados para la amplificación por PCR.
Tabla 2 Número recomendado de ciclos para generar una biblioteca de ARN*
| Entrada de ARN total | Número de ciclos | |
| No varado | Varado | |
| 10 ng | 15 | 15 |
| 100 ng | 14 | 14 |
| 500 ng | 12 | 13 |
| 1 μg | 11 | 12 |
Nota: *El rendimiento de la biblioteca no solo está relacionado con la cantidad de entrada y el número de ciclos de amplificación. pero también se ve afectada por la calidad de las muestras, las condiciones de fragmentación y las condiciones de clasificación. En el proceso de construcción de la biblioteca, elija las condiciones más adecuadas según la situación real.
4. Limpieza y selección del tamaño del ADN con perlas
4.1 Existen varios pasos en el proceso de construcción de la biblioteca que requieren perlas magnéticas para la purificación del ADN. Recomendamos perlas de selección de ADN Hieff NGS™ (Yeasen Cat#12601) o perlas magnéticas AMPure® XP (Beckman Cat#A63880) para la purificación del ADN y la selección del tamaño.
4.2 Las perlas magnéticas deben equilibrarse a temperatura ambiente antes de su uso, de lo contrario el rendimiento disminuirá y el efecto de selección de tamaño se verá afectado.
4.3 Las perlas magnéticas deben mezclarse bien mediante vórtex o pipeteo antes de su uso.
4.4 No aspire las perlas al transferir el sobrenadante; incluso trazas de perlas pueden afectar las siguientes reacciones.
4.5 El etanol al 80% debe prepararse recién, de lo contrario afectará la eficiencia de recuperación.
4.6 Las perlas magnéticas deben secarse a temperatura ambiente antes de eluir el producto. Una sequedad insuficiente puede provocar que los residuos de etanol afecten fácilmente las reacciones posteriores; una sequedad excesiva puede provocar que las perlas magnéticas se agrieten y reduzcan el rendimiento de la purificación. Normalmente, un secado a temperatura ambiente durante 3 a 5 minutos es suficiente para permitir que las perlas se sequen por completo.
4.7 Si es necesario, las muestras de ADN purificadas o de tamaño seleccionado eluidas en tampón TE se pueden almacenar a 4 °C durante 1-2 semanas o a -20 °C durante un mes.
5 Análisis de la calidad de la biblioteca
5.1 Normalmente, la calidad de la biblioteca construida se puede evaluar mediante la distribución de longitud y la detección de concentración.
5.2 Detección de la concentración de la biblioteca: métodos basados en colorantes fluorescentes de ADN de doble cadena, como Qubit®, PicoGreen®, etc.; cuantificación absoluta basada en qPCR.
5.3 Los métodos basados en detección espectral, como NanoDrop®, etc., no son aplicables a la detección de concentraciones de bibliotecas.
5.4 Se recomienda la qPCR para la detección de la concentración de la biblioteca: mediante Qubit®, PicoGreen® y otros métodos basados en colorantes fluorescentes de ADN de doble cadena, no se puede distinguir de forma eficaz entre productos ligados a adaptadores en un extremo, productos no ligados a adaptadores en ambos extremos y otros productos de doble cadena incompletos. La cuantificación absoluta de la qPCR se basa en el principio de la amplificación por PCR, que solo cuantifica la biblioteca completa del adaptador en ambos extremos de la muestra (la biblioteca que se puede secuenciar), excluyendo la interferencia de bibliotecas no secuenciables que no están ligadas al adaptador en los extremos de un solo extremo o de doble extremo.
5.5 La detección de la distribución de la longitud de la biblioteca se puede realizar mediante Agilent Bioanalyzer 2100 y otros equipos basados en el principio de electroforesis capilar o microfluídica.
Documentos:
Citas y referencias:
[1] Descifrando el microbioma intestinal de la carpa herbívora mediante un enfoque multiómico
M Li, H Liang, H Yang, Q Ding, R Xia, J Chen, W Zhou… - Microbioma, 2024 IF:19.4
[2] La detección agrupada de CRISPR identifica a los P-Bodies como represores de la transición epitelial-mesenquimal del cáncer
L Fang, L Zhang, M Wang, Y He, J Yang, Z Huang… - Investigación sobre el cáncer, 2024 IF:12.7
[3] El péptido 1 derivado de Klotho inhibe la senescencia celular en el riñón fibrótico al restaurar la expresión de Klotho a través de la regulación postranscripcional.
X Zhang, L Li, H Tan, X Hong, Q Yuan, FF Hou… - Teranósticos, 2024 IF:12.4
[4] Células madre parcialmente recubiertas de exoesqueleto para la restauración del miocardio infartado
H He, Y Yuan, Y Wu, J Lu, X Yang, K Lu… - Materiales Avanzados, 2023 IF: 29,4
[5] Innovación genómica y reconfiguración regulatoria durante la evolución del género de algodón Gossypium
M Wang, J Li, Z Qi, Y Long, L Pei, X Huang… - Genética de la naturaleza, 2022 IF:41.379
[6] Los alelos de riesgo MTMR3 mejoran la inmunidad IgA inducida por el receptor tipo Toll 9 en la nefropatía por IgA
Y Wang, T Gan, S Qu, L Xu, Y Hu, L Liu, S Shi, J Lv… - Kidney International, 2023 IF:19.6
[7] Complementación dividida de editores base para minimizar ediciones fuera de objetivo
X Xiong, K Liu, Z Li, FN Xia, XM Ruan, X He, JF Li - Plantas naturales, 2023 IF:18.6
[8] Las vesículas de membrana externa bacterianas diseñadas que encapsulan adenovirus oncolíticos mejoran la eficacia de la viroterapia contra el cáncer al aumentar la autofagia de las células tumorales.
W Ban, M Sun, H Huang, W Huang, S Pan… - Comunicaciones de la naturaleza, 2023 IF:16.6
[9] La resistencia de las células cancerosas al IFNγ puede ocurrir a través de una mayor actividad de la vía de reparación de roturas de doble cadena
T Han, X Wang, S Shi, W Zhang, J Wang, Q Wu… - Investigación en inmunología del cáncer, 2023 IF: 12.0
[10] Terapia combinada basada en un sistema de nanoadministración biomimética de doble objetivo para superar la resistencia al cisplatino en el carcinoma hepatocelular
Y Huang, Q Kou, Y Su, L Lu, X Li, H Jiang… - Journal of Nanobiotechnology, 2023 IF: 10.9
[11] PIAS3 promueve la ferroptosis regulando TXNIP a través de la vía de señalización de TGF-β en el carcinoma hepatocelular.
W Bao, J Wang, K Fan, Y Gao, J Chen - Investigación farmacológica, 2023 IF:9.3
[12] Identificación de dianas proteicas de unión al ARN con HyperTRIBE en Saccharomyces cerevisiae
W Piao, C Li, P Sun, M Yang, Y Ding, W Song… - Revista Internacional de Ciencias Moleculares, 2023 IF:5.6
[13] La microbiota regula la transición del ciclo de vida y la dinámica de los nematocitos en las medusas
S Peng, L Ye, Y Li, F Wang, T Sun, L Wang, W Hao… - Iscience, 2023 IF: 5.08
[14] Los perfiles del transcriptoma y de los miRNA revelan la red reguladora y los reguladores clave de la formación del xilema secundario en el álamo “84K”
H Wang, P Zhao, Y He, Y Su, X Zhou… - Revista internacional de ciencia molecular, 2023 IF:5.6
Pago y seguridad
Su información de pago se procesa de forma segura. No almacenamos detalles de la tarjeta de crédito ni tenemos acceso a la información de su tarjeta de crédito.
Consulta
También te puede gustar
Preguntas frecuentes
El producto es solo para fines de investigación y no está destinado a uso terapéutico o diagnóstico en humanos o animales. Los productos y el contenido están protegidos por patentes, marcas comerciales y derechos de autor propiedad de Yeasen Biotechnology. Los símbolos de marca comercial indican el país de origen, no necesariamente el registro en todas las regiones.
Algunas aplicaciones pueden requerir derechos de propiedad intelectual adicionales de terceros.
Yeasen se dedica a la ciencia ética y cree que nuestra investigación debe abordar cuestiones críticas al tiempo que garantiza la seguridad y los estándares éticos.

