Descripción
Las perlas de selección de ADN Hieff NGS™ se preparan según el principio SPRI (inmovilización inversa en fase sólida) y se pueden utilizar para la purificación y selección de tamaño de ADN durante la preparación de bibliotecas de secuenciación de próxima generación (NGS). Las perlas de selección de ADN Hieff NGS™ son compatibles con varios kits de preparación de bibliotecas de ADN y ARN y son una buena alternativa a las Perlas AMPure.
Componentes
| Componentes No. | Nombre | 12601ES08 | 12601ES56 | 12601ES75 |
| 12601 | Perlas de selección de ADN Hieff NGS™ | 5 ml | 60 ml | 450 ml |
Presupuesto
| Línea de productos | Perlas de limpieza y selección de ADN |
| Material de partida | ADN |
| Compatibilidad | ADN |
| Tecnología de aislamiento | Perla magnética |
| Tipo de producto final | ADN |
| Para usar con (aplicación) | Limpieza del ADN, selección del tamaño del ADN |
Envío y almacenamiento
Las perlas se envían con bolsas de hielo y se pueden almacenar a una temperatura entre 2 °C y 8 °C durante un año.
Instrucciones
- 1. Preparación
Equilibrar las perlas de selección a temperatura ambiente durante al menos 30 minutos antes de su uso.
- 2. Selección del tamaño del ADN
El flujo de operación de selección de tamaño se muestra en la Figura 1 y el protocolo es el siguiente.
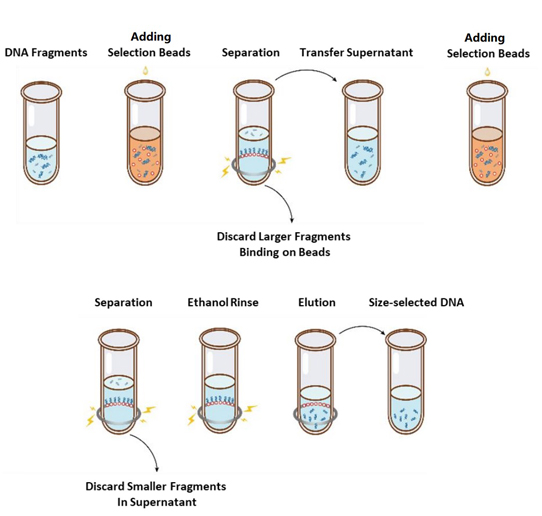
Figura 1. Diagrama de flujo de la selección del tamaño del ADN
2.1 Mezcle bien las perlas mediante agitación en vórtex o pipeteando hacia arriba y hacia abajo cada vez antes de usarlas.
2.2 Agregue la primera ronda de perlas de selección a la muestra (consulte la Tabla 1). Mezcle bien agitando con vórtex o pipeteando hacia arriba y hacia abajo al menos 10 veces.
2.3 Incubar a temperatura ambiente durante 5 min.
2.4 Centrifugue brevemente el tubo y colóquelo sobre un soporte magnético. Cuando la solución esté transparente (aproximadamente 5 minutos), transfiera el sobrenadante a un nuevo tubo de PCR.
2.5 Agregue la segunda ronda de perlas de selección a la muestra del paso 2.4 de acuerdo con la Tabla 1. Mezcle bien agitando o pipeteando hacia arriba y hacia abajo al menos 10 veces.
2.6 Incubar a temperatura ambiente durante 5 min.
2.7 Centrifugue brevemente el tubo y colóquelo sobre un soporte magnético. Cuando la solución esté transparente (aproximadamente 5 minutos), aspire el sobrenadante y deséchelo.
2.8 Mantenga el tubo en el soporte magnético y agregue 200 μL de etanol al 80 % recién preparado sin alterar las perlas. Incube a temperatura ambiente durante 30 segundos. Aspire el etanol y deséchelo.
2.9 Repita el paso 2.8 una vez para un total de dos lavados.
2.10 Retire el etanol residual con puntas de pipeta de 10 µL. Mantenga el tubo en el soporte magnético, seque al aire las perlas de selección con la tapa abierta hasta que aparezcan grietas (aproximadamente 5 minutos).
Nota: No seque demasiado las perlas de selección. Esto puede dar como resultado un objetivo de ADN de recuperación más bajo.
2.11 Retire el tubo del soporte magnético. Agregue una cantidad adecuada de ddH2O (≥20 µL) y mezcle bien agitando con vórtex o pipeteando hacia arriba y hacia abajo al menos 10 veces.
2.12 Incubar a temperatura ambiente durante 5 min.
Centrifugue brevemente el tubo y colóquelo sobre el soporte magnético. Cuando la solución esté transparente (aproximadamente 5 minutos), transfiera 20 μL del sobrenadante a un tubo nuevo.
- 3. Condiciones recomendadas para la selección del tamaño del ADN
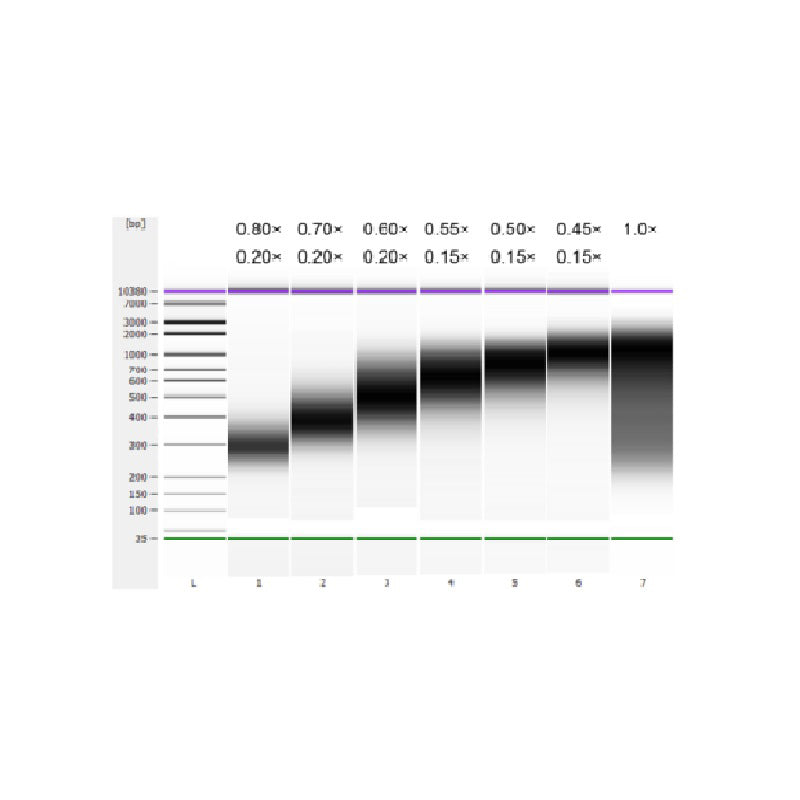
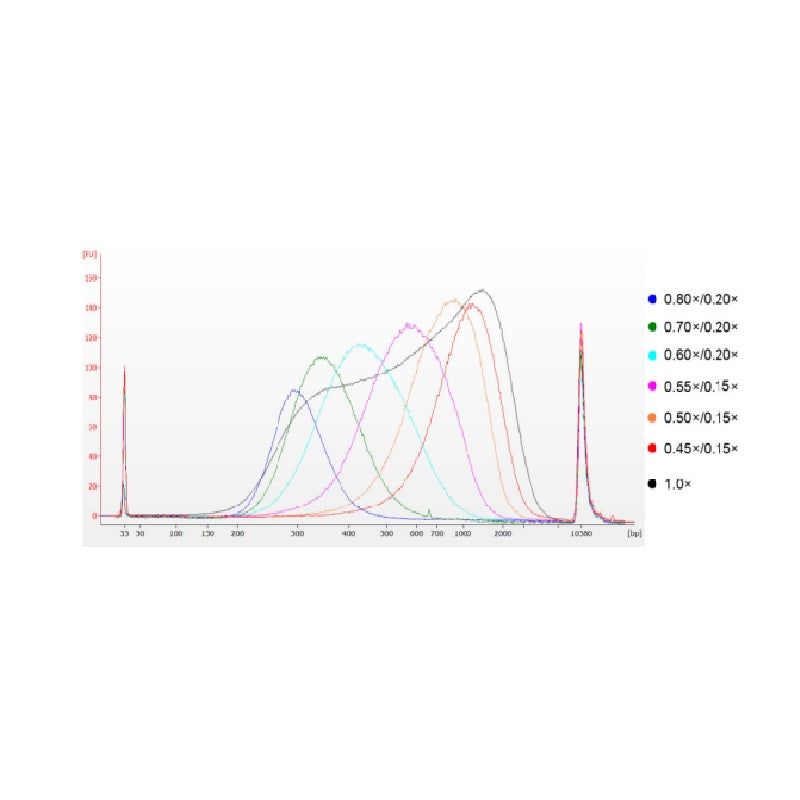
El ADN del timo de ternera se fragmentó mediante sonicación para preparar un fragmento de 100-1.000 pb, y se realizaron dos rondas de selección de tamaño de acuerdo con la Tabla 1. Los resultados se analizaron utilizando el Bioanalizador Agilent 2100 (Figura 2).
Tabla 1. Condiciones recomendadas para la selección del tamaño del ADN
| Longitud del fragmento de ADN | 250-350 pb | 320-420 pb | 450-550 pb | 550-700 pb | 700-900 pb | 800-1.000 pb |
| Relación de cuentas: ADN para la 1ª ronda | 0,80× | 0,70× | 0,60× | 0,55× | 0,50× | 0,45× |
| Relación de cuentas: ADN para el 2do Redondo | 0,20× | 0,20× | 0,20× | 0,15× | 0,15× | 0,15× |
Nota: "×" en la tabla indica el volumen de ADN de muestra. Por ejemplo, si la longitud del inserto de la biblioteca es de 250 pb y el volumen de ADN de muestra es de 100 μL, el volumen de perlas magnéticas utilizado en la primera ronda de clasificación es de 0,80×100 μL=80 μL; el volumen de perlas magnéticas utilizado en la segunda ronda de clasificación es de 0,20×100 μL=20 μL.
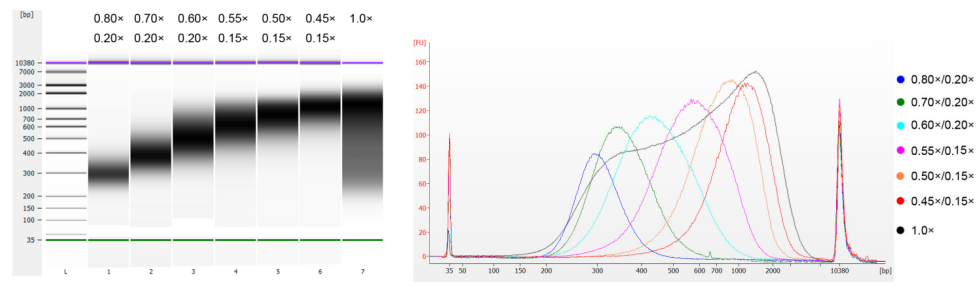
Figura 2. Electroferograma del chip de ADN de alta sensibilidad Agilent 2100
Notas:
1. Para su seguridad y salud, utilice bata de laboratorio y guantes desechables durante la operación.

Citado de "Integración específica de secuencia por la casposasa de la familia 1 de Candidatus Nitrosopumilus koreensis" AR1. Res. de ácidos nucleicos 2021;49(17):9938-9952. doi:10.1093/nar/gkab725"

Citado de "La infección reciente por Wolbachia altera las comunidades microbianas en poblaciones silvestres de Laodelphax striatellus. Microbiome. 2020;8(1):104. Publicado el 2 de julio de 2020. doi:10.1186/s40168-020-00878-x"
[1] Wang X, Yuan Q, Zhang W, et al. Integración específica de secuencia por la casposasa de la familia 1 de Candidatus Nitrosopumilus koreensis AR1. Res. de ácidos nucleicos 2021;49(17):9938-9952. doi:10.1093/nar/gkab725(IF:16.971)
[2] Duan XZ, Sun JT, Wang LT, et al. La infección reciente por Wolbachia altera las comunidades microbianas en poblaciones silvestres de Laodelphax striatellus. Microbiome. 2020;8(1):104. Publicado el 2 de julio de 2020. doi:10.1186/s40168-020-00878-x(IF:11.607)
[3] Canción B, Almatrafi E, Sang F, et al.Manejo de sedimentos tratados con Fenton con biocarbón y compost de estiércol de oveja: efectos sobre las características evolutivas de la comunidad bacteriana. J Environ Manage. 2022;316:115218. doi:10.1016/j.jenvman.2022.115218(IF:6.789)
[4] Huang C, Mei Q, Lou L, et al. Colitis ulcerosa en respuesta al trasplante de microbiota fecal mediante la modulación de la microbiota intestinal y el equilibrio de células Th17/Treg. Cells. 2022;11(11):1851. Publicado el 5 de junio de 2022. doi:10.3390/cells11111851(IF:6.600)
[5] Ghosh S, Yang X, Wang L, Zhang C, Zhao L. La alimentación prebiótica en fase activa altera la microbiota intestinal e induce un alivio independiente del peso de la esteatosis hepática y el colesterol sérico en ratones alimentados con una dieta rica en grasas. Comput Struct Biotechnol J. 2020;19:448-458. Publicado el 24 de diciembre de 2020. doi:10.1016/j.csbj.2020.12.011(IF:6.018)
[6] Gao X, Yu B, Yu J, et al. Perfiles de desarrollo de la digestión de carbohidratos en la dieta en lechones. Front Microbiol. 2022;13:896660. Publicado el 29 de abril de 2022. doi:10.3389/fmicb.2022.896660(IF:5.640)
[7] Li P, Zhang Y, Yan F, Zhou X. Características de un bacteriófago, vB_Kox_ZX8, aislado de Klebsiella oxytoca clínica y su efecto terapéutico en la bacteriemia en ratones. Front Microbiol. 2021;12:763136. Publicado el 3 de diciembre de 2021. doi:10.3389/fmicb.2021.763136(IF:5.640)
[8] Lin Z, Luo P, Huang D, Wu Y, Li F, Liu H. Estrategia basada en multiómica para el análisis de toxicidad de la acrilamida en el modelo Saccharomyces cerevisiae. Chem Biol Interact. 2021;349:109682. doi:10.1016/j.cbi.2021.109682(IF:5.194)
[9] Sun X, Lv W, Wang Y, et al. El gen Mrgprb2 desempeña un papel en las reacciones anafilactoides inducidas por la inyección de Houttuynia cordata. J Etnofarmacol. 2022;289:115053. doi:10.1016/j.jep.2022.115053(IF:4.360)
[10] Ma H, Lai B, Zan C, Di X, Zhu X, Wang K. GLO1 contribuye a la resistencia farmacológica de Escherichia coli mediante la inducción de β-lactamasas de espectro extendido de tipo PER. Infect Drug Resist. 2022;15:1573-1586. Publicado el 5 de abril de 2022. doi:10.2147/IDR.S358578(IF:4.003)
[11] Zhong Y, Zhao W, Tang Z, et al. Análisis transcriptómico comparativo de las diferentes etapas de desarrollo del ovario en el cangrejo de río rojo Procambarus clarkii. BMC Genomics. 2021;22(1):199. Publicado el 21 de marzo de 2021. doi:10.1186/s12864-021-07537-x(IF:3.969)
[12] Lian C, Yang H, Lan J, et al. El análisis comparativo de los genomas de los cloroplastos revela relaciones filogenéticas y variación intraespecífica en la planta medicinal Isodon rubescens. PLoS One. 2022;17(4):e0266546. Publicado el 6 de abril de 2022. doi:10.1371/journal.pone.0266546(IF:3.240)
[13] Diao G, Huang J, Zheng X, et al. Prostaglandina La E2 cumple una doble función en la regulación de la migración de las células dendríticas. Int J Mol Med. 2021;47(1):207-218. doi:10.3892/ijmm.2020.4801(IF:3.098)
[14] Bing XL, Zhao DS, Peng CW, Huang HJ, Hong XY. Similitudes y variaciones espaciales de las comunidades bacterianas y fúngicas en poblaciones de insectos del arroz (Hemiptera: Delphacidae). Insect Sci. 2020;27(5):947-963. doi:10.1111/1744-7917.12782(IF:2.791)
[15] Li X, Zhou S, Zhang J, Zhou Z, Xiong Q. Cambios direccionales en la comunidad bacteriana intestinal en larvas de la mosca soldado negra (Hermetia illucens). Animals (Basel). 2021;11(12):3475. Publicado el 6 de diciembre de 2021. doi:10.3390/ani11123475(IF:2.752)
[16] Yang J, Peng Y, Kong W. Identificación y secuenciación completa del genoma del virus críptico de la morera 1. Arch Virol. 2022;167(2):687-690. doi:10.1007/s00705-021-05350-1(IF:2.574)
[17] Chang Y, Xia X, Sui L, et al. La colonización endofítica de hongos entomopatógenos aumenta la resistencia de las plantas a las enfermedades al cambiar la comunidad bacteriana endofítica. J Basic Microbiol. 2021;61(12):1098-1112. doi:10.1002/jobm.202100494(SI:2.281)
[18] Ding CY, Ma YM, Li B, et al. Identificación y análisis funcional de genes expresados diferencialmente en Myzus persicae (Hemiptera: Aphididae) en respuesta a trans-anetol. J Insect Sci. 2022;22(1):3. doi:10.1093/jisesa/ieab094(IF:1.857)
Pago y seguridad
Su información de pago se procesa de forma segura. No almacenamos detalles de la tarjeta de crédito ni tenemos acceso a la información de su tarjeta de crédito.
Consulta
También te puede gustar
Preguntas frecuentes
El producto es solo para fines de investigación y no está destinado a uso terapéutico o diagnóstico en humanos o animales. Los productos y el contenido están protegidos por patentes, marcas comerciales y derechos de autor propiedad de Yeasen Biotechnology. Los símbolos de marca comercial indican el país de origen, no necesariamente el registro en todas las regiones.
Algunas aplicaciones pueden requerir derechos de propiedad intelectual adicionales de terceros.
Yeasen se dedica a la ciencia ética y cree que nuestra investigación debe abordar cuestiones críticas al tiempo que garantiza la seguridad y los estándares éticos.