Descripción
Hieff NGSMarca registrada El kit de preparación de biblioteca de ADN es un kit de construcción de bibliotecas de nueva generación especialmente desarrollado y diseñado para Illumina® y MGI® plataforma de secuenciaciónBasado en la generación anterior del kit de construcción de bibliotecas, este producto presenta una mayor eficiencia en la reparación de extremos, la unión de dA y la ligadura de adaptadores que las versiones anteriores. La enzima de alta fidelidad mejora significativamente la uniformidad y la fidelidad de la amplificación. El kit es compatible con la mayoría de los tipos de muestras de ADN, incluyendo ADN genómico estándar de animales, plantas y microorganismos, muestras FFPE, ADNcf y ADN ChIP.
Presupuesto
| N.º de cat. | 12927ES08 / 12927ES24 / 12927ES96 |
| Tamaño | 8 rxn / 24 rxn / 96 rxn |
Componentes
| Componentes No. | Nombre | 12927ES08 | 12927ES24 | 12927ES96 |
| 12927-A | 56 μL | 168 μL | 672 μL | |
| 12927-B | Enzima Endprep | 24 μL | 72 μL | 288 μL |
| 12927-DO | Potenciador de la ligadura | 240 μL | 720 μL | 3×960 μL |
| 12927-D | Rápido Ligasa de ADN T4 | 80 μL | 240 μL | 2×480 μL |
| 12927-MI | CanaceMarca registrada Mezcla de amplificación profesional | 200 μL | 600 μL | 3×800 μL |
Almacenamiento
Este producto debe almacenarse a -25 ~ -15 ℃ durante 1 año.
Notas
1. Acerca de la operación
1. Por favor, trabaje con bata de laboratorio y guantes desechables.,Para su seguridad.
2. Descongele los componentes a temperatura ambiente. Después de la descongelación, mezclar bien mediante vórtex, girar el tubo brevemente y colocarlo en hielo para su uso posterior.
3. Al preparar la solución de reacción de cada paso, se recomienda usar una pipeta para mezclar bien o agitar suavemente. Una agitación vigorosa puede reducir la producción de la biblioteca.
4. Se recomienda encarecidamente utilizar puntas de pipeta con filtro para evitar la contaminación cruzada. Asegúrese de cambiar las puntas de pipeta al procesar diferentes muestras.
5. Las operaciones incorrectas pueden causar contaminación por aerosoles, lo que afecta la precisión del resultado. Se recomienda el aislamiento físico obligatorio de las regiones de mezcla de la reacción de PCR y de las regiones de ensayo de purificación del producto de PCR. Se deben utilizar equipos como pipetas especializadas para la construcción de bibliotecas.Realice una limpieza de rutina para cada área limpiando las superficies con hipoclorito de sodio al 0,5 % o blanqueador al 10 %.
6. Este producto es solo para uso en investigación.
2. Fragmentación del ADN
1. Este kit es compatible con ADN fragmentado mecánicamente o enzimáticamente.
2. El kit es compatible con 100 pg - 1000 ng de ADN de entrada. Se recomienda encarecidamente utilizar ADN de entrada de alta calidad con una A260/A280 de 1,8 a 2,0. La Tabla 1 muestra la cantidad recomendada de ADN de entrada.
Tabla 1 Cantidad recomendada de ADN de entrada
| Solicitud | Tipos de muestra | ADN de entrada |
| WGS | Genoma complejo | 50 ng-1000 ng |
| Secuenciación de captura dirigida | Genoma complejo | 10 ng-1000 ng |
| WGS, secuenciación dirigida | ADN FFPE | 50 ng-1000 ng |
| Secuenciación dirigida | ADNcf/ADNct | ≥500 pg |
| WGS | genomas microbianos | ≥1 ng |
| WGS (sin PCR) | ADN de entrada de alta calidad | ≥50 ng |
Nota:Cuando el ADN de entrada es de mala calidad o se requiere una selección del tamaño del ADN, la cantidad de ADN de entrada se debe aumentar en consecuencia.
3. “ADN de entrada” se refiere específicamente a las muestras de ADN listas para reparación final/cola de dA.
4. Se recomienda un paso de purificación/selección de tamaño de las microesferas después de la fragmentación si la muestra de ADN de entrada contiene altas concentraciones de sales, como el agente quelante de metales. Las sales podrían afectar la eficiencia de las reacciones posteriores, como la reparación de extremos y la formación de colas de dA.Si utiliza el método de fragmentación mecánica, eluya las muestras de ADN en tampón TE en lugar de agua ultrapura esterilizada para la fragmentación. Si utiliza el método de fragmentación enzimática sin realizar la limpieza de las microesferas ni la selección de tamaño antes de preparar la biblioteca, asegúrese de que el tampón de parada utilizado no contenga una cantidad excesiva de agente quelante de metales. De lo contrario, limpie o seleccione las muestras fragmentadas y elúyalas en tampón TE o agua ultrapura esterilizada (≤50 μL) antes de preparar la biblioteca.
3. Ligadura del adaptador
1. Los kits de adaptador largo (adaptador con código de barras) Illumina o MGI y los kits de adaptador corto están disponibles para que los clientes elijan según sus requisitos experimentales.
2. Se recomendó seleccionar adaptadores comerciales de alta calidad. Si se opta por adaptadores de fabricación propia, se recomienda confiar la fabricación a una empresa con experiencia en la síntesis de cebadores NGS y destacar la necesidad de un estricto control de la contaminación. Además, se recomienda preparar la solución de hibridación de ADN en una mesa de trabajo limpia y utilizar solo un tipo de adaptador a la vez para evitar la contaminación cruzada.
3. Descongele los adaptadores en hielo o a 4 °C; cuando se opera a temperatura ambiente, la temperatura del laboratorio no debe superar los 25 °C para evitar que los adaptadores se desnaturalicen.
4. La calidad y concentración de los adaptadores afectarán directamente la eficiencia de la ligadura y el rendimiento de la biblioteca. Una concentración demasiado alta de adaptadores favorece la formación de dímeros, mientras que una concentración insuficiente reduce la tasa de ligadura y el rendimiento de la biblioteca. Diluciones correspondientes con tampón TE según la cantidad de ADN de entrada al usar el adaptador. Tabla 2 -5 enumera los métodos de dilución del adaptador recomendados para diferentes cantidades de ADN de entrada utilizando este kit.
Tabla 2 El Illumina™ recomendado Cantidad de adaptador para diferentes entradas de ADN
| ADN de entrada | Dilución del adaptador (volumen del adaptador: volumen total) | Concentración |
| 0,1 ng ~ 1 ngramo | 150 veces (1: 150) | 0,1 μM |
| 1 ng ~ 10 ng | 75 veces (1:75) | 0,2 μM |
| 10 ng ~ 25 ng | 15 veces (1:15) | 1 μM |
| 25 ng ~ 100 ng | 7,5 veces (1 : 7.5) | 2 μM |
| 100 ng ~ 1000 ng | Triple (1:3) | 5 μM |
Tabla 3 El MGI™ recomendado Cantidad de adaptador para diferentes entradas de ADN
| ADN de entrada | Dilución del adaptador (volumen del adaptador: volumen total) | Concentración |
| 0,1 ng ~ 1 ngramo | 100 pliegues (1:1)00) | 0,1 μM |
| 1 ng ~ 10 ng | 50-Pliegue (1 : 50) | 0,2 μM |
| 10 ng ~ 25 ng | 10-Pliegue (1:1)0) | 1 μM |
| 25 ng ~ 100 ng | 5 veces (1:5) | 2 μM |
| 100 ng ~ 1000 ng | 2-Pliegue (1 : 2) | 5 μM |
Tabla 4 El Illumina™ recomendado Cantidad de adaptador UMI para diferentes entradas de ADN
| ADN de entrada | Dilución del adaptador (volumen del adaptador: volumen total) | Concentración |
| 0,1 ng ~ 1 ngramo | 150 veces (1: 150) | 0,1 μM |
| 1 ng ~ 10 ng | 75 veces (1:75) | 0.2 μM |
| 10 ng ~ 25 ng | 15 veces (1:15) | 1 μM |
| 25 ng ~ 100 ng | 7,5 veces (1: 7,5) | 2 μM |
| 100 ng ~ 1000 ng | Triple (1:3) | 5 μM |
Tabla 5 El MGI™ recomendado Cantidad de adaptador UMI para diferentes entradas de ADN
| ADN de entrada | Dilución del adaptador (volumen del adaptador: volumen total) | Concentración |
| 5 ng ~ 25 nortegramo | 50 pliegues (1 : 50) | 0.2 μM |
| 25 ng ~ 100 ng | 10-Pliegue (1 : 10) | 1 μM |
| 100 ng ~ 1000 ng | 4-Pliegue (1 : 4) | 2,5 μM |
4. Limpieza de ADN y selección de tamaño con perlas
1. La selección del tamaño del ADN se puede realizar antes de la reparación final/cola de dA, después de la ligadura del adaptador o después de la amplificación.
2. Se recomienda realizar la selección de tamaño inmediatamente después de la ligadura del adaptador si la cantidad de ADN de entrada es superior a 50 ng.; De lo contrario, realice la selección de tamaño después de la amplificación.
3. El potenciador de ligadura contiene una alta concentración de PEG, lo que puede afectar significativamente la precisión de la selección de tamaño. Por lo tanto, si la selección de tamaño se realiza inmediatamente después de la ligadura del adaptador, se recomienda encarecidamente añadir un paso de limpieza de microesferas antes de la selección de tamaño. La selección de tamaño puede realizarse directamente si se realiza antes de la reparación final/cola de dA o después. La amplificación de la biblioteca.
4. Las perlas magnéticas deben equilibrarse a temperatura ambiente antes de su uso, de lo contrario, el rendimiento disminuirá y el efecto de selección de tamaño se verá afectado.
5. Las perlas magnéticas deben mezclarse bien mediante vórtex o pipeteo antes de su uso.
6. No aspire las perlas al transferir el sobrenadante; incluso trazas de perlas pueden afectar las siguientes reacciones.
7. El etanol al 80% debe prepararse recién, de lo contrario afectará la eficiencia de recuperación.
8. Para una selección precisa del tamaño, se recomienda comenzar con un volumen superior a 100 μL. Si es menor, se recomienda aumentarlo hasta 100 μL con agua ultrapura.
9. Las perlas magnéticas deben secarse a temperatura ambiente antes de eluir el producto. Una sequedad insuficiente puede provocar que los residuos de etanol afecten fácilmente las reacciones posteriores; una sequedad excesiva puede agrietar las perlas magnéticas y reducir el rendimiento de la purificación. Normalmente, un secado a temperatura ambiente de 3 a 5 minutos es suficiente para que las perlas se sequen por completo.
10. Si es necesario, las muestras de ADN purificadas o de tamaño seleccionado se eluyen en 0,1× El tampón TE se puede almacenar a 4 °C durante 1 o 2 semanas o a -20 °C durante un mes.
5. Amplificación de la biblioteca
1. La realización o no de la amplificación de la biblioteca depende de la cantidad de ADN de entrada, los tipos de adaptadores, las aplicaciones de los datos de secuenciación, etc. La amplificación es necesaria si se utilizan adaptadores parciales. Al utilizar adaptadores de longitud completa, si el ADN de entrada es <200 ng, se recomienda realizar la amplificación; de lo contrario, no es necesaria.
2. El número de ciclos de amplificación debe controlarse estrictamente. Una amplificación insuficiente puede resultar en un bajo rendimiento de la biblioteca; la sobreamplificación puede generar mayor sesgo, errores, lecturas duplicadas y productos quiméricos. Tabla 6 enumera los números de ciclos recomendados que apuntan al rendimiento de la biblioteca de 1 μg.
Tabla 6 Número recomendado de ciclos para generar 1.000 ng de rendimiento de la biblioteca
| ADN de entrada | Número de ciclos necesarios para generar 1 μg de rendimiento de biblioteca |
| 1000 ng | 2 - 4 |
| 500 ng | 2 - 4 |
| 250 ng | 4 - 6 |
| 100 ng | 5 - 7 |
| 50 ng | 7 - 9 |
| 10 ng | 9 - 11 |
| 5 ng | 10 - 12 |
| 1 ng | 12 - 15 |
| 100 páginas | 16 - 18 |
Nota:
1.Tabla 6 muestra la cantidad de parámetros de bucle utilizando pruebas de ADN de entrada de alta calidad de alrededor de 200 pb. La calidad del ADN FFPE varía mucho y, cuando la calidad del ADN es deficiente o la longitud de la biblioteca es larga, es necesario aumentar adecuadamente la cantidad de ciclos para obtener bibliotecas suficientes.
2.ISi se requiere la selección del tamaño durante el proceso de construcción de la biblioteca, se recomienda un número de ciclo más alto para la amplificación de la biblioteca; de lo contrario, se recomienda un número de ciclo más bajo.
3.ISi se utilizan adaptadores incompletos, se deben amplificar al menos 2 ciclos para formar un adaptador completo.
6. Análisis de la calidad de la biblioteca
1. La calidad de las bibliotecas construidas generalmente se analiza midiendo las concentraciones y distribuciones de tamaño.
2. Las concentraciones de las bibliotecas se pueden medir mediante métodos basados en fluorescencia como Qubit y PicoGreen o qPCR.
3. NO se recomienda utilizar métodos de cuantificación basados en absorbancia como NanoDrop.
4. Se recomienda utilizar el método qPCR para la cuantificación de bibliotecas: los métodos basados en fluorescencia, como Qubit y PicoGreen, no pueden diferenciar las estructuras de dsADN incompletas (insertos sin adaptador o con solo uno de los extremos ligado con un adaptador) de las bibliotecas completas. El método qPCR solo amplifica y mide las bibliotecas completas con ambos extremos ligados con adaptadores (las bibliotecas secuenciables), lo que proporciona una medición más precisa de la carga.
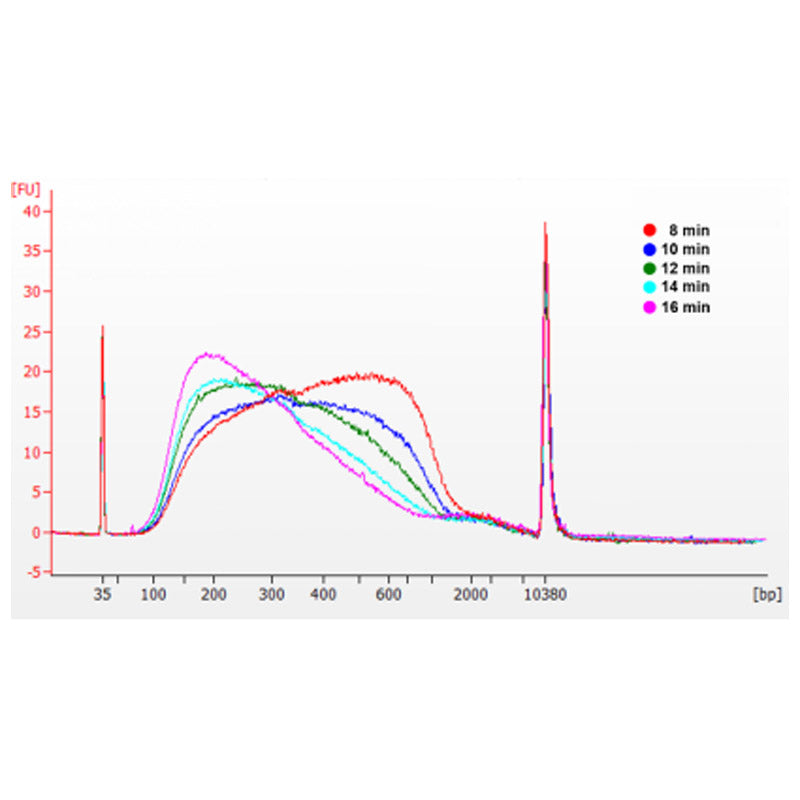
5.La distribución del tamaño de las bibliotecas se puede analizar utilizando Agilent Bioanalyzer u otros dispositivos basados en los principios de electroforesis capilar o microfluídica.
7. Otros materiales
1. Perlas magnéticas para purificación de ADN: Hieff NGSMarca registrada Perlas de selección de ADN (
2. Adaptadores: Adaptador completo para Illumina:
3. Análisis de calidad de biblioteca: Agilent 2100 Bioanalyzer DNA 1000 Chip/High Sensitivity Chip u otros productos equivalentes; reactivos cuantitativos de biblioteca.
4. Otros materiales: etanol absoluto, agua ultrapura estéril, puntas de pipeta de baja retención, tubo de PCR, soportes magnéticos, termociclador, etc.
Instrucciones
Paso 1. Reparación final/dA-Taling
1. Deshielo los reactivos mencionados en la Tabla 7 . Invierta para mezclar completamente los reactivos y colóquelos en hielo para su uso posterior.
2. Ensamble los reactivos según la Tabla 7 con hielo.
Mesa 7 Sistema de reacción de reparación de extremos/dA-Tailing
| Componentes | Volumen (μL) |
| ADN fragmentado | incógnita |
| Búfer de preparación final | 7 |
| Enzima Endprep | 3 |
| ddH2Oh | Hasta 60 |
3. Mezcle suavemente pipeteando o agitando. Centrifugue brevemente para obtener la solución.
4. Coloque el tubo en un termociclador y configure el programa de acuerdo con la tabla 8.
Tabla 8 Reparación de extremos/dA-Tailing programa de reacción
| Temperatura | Tiempo |
| Calentar la tapa a 105 °C | En |
| 30 °C | 30 minutos |
| 72 °C | 30 minutos |
| 4 °C | Sostener |
Paso 2. Ligadura del adaptador
1. Diluya el adaptador a la concentración adecuada según la Tabla 2-5.
2. Deshielo los reactivos mencionados en la Tabla 9 . Invierta para mezclar completamente los reactivos y colóquelos en hielo para su uso posterior.
3. Ensamble los reactivos según la Tabla 9 con hielo.
Tabla 9 Ligadura del adaptador sistema de reacción
| Componentes | Volumen (μL) |
| ADN con cola dA(Producto del paso 1) | 60 |
| Potenciador de la ligadura | 30* |
| Adaptador de ADN | 5** |
| Rápido Ligasa de ADN T4 | 10 |
| ddH2O | 5 |
| Total | 110 |
Nota*El potenciador de ligadura es viscoso. Mezcle bien invirtiendo o volteando. centrifugar brevemente antes de usar.
**La concentración original de IlluminaMarca registrada El adaptador de YEASE es de 15 μM. Diluya el adaptador según la cantidad de entrada. y fijar el volumen del adaptador en 5 μL.
**La concentración original del MGIMarca registrada El adaptador de YEASE es 10 μM. Diluya el adaptador según la cantidad de entrada. y fijar el volumen del adaptador en 5 μL.
4. Mezcle bien pipeteando suavemente hacia arriba y hacia abajo y centrifugando brevemente para recoger todo el líquido de los lados del tubo.
5. Incubar la muestra en un termociclador precalentado como se muestra en la Tabla 10 y realizar la reacción de conexión del adaptador.
Tabla 10 Programa de reacción de ligadura del adaptador
| Temperatura | Tiempo |
| Calentar la tapa a 105°C | Apagado |
| 20°C | 15 minutos |
| 4°C | Sostener |
Paso 3. Limpieza o selección de tamaño después de la ligadura del adaptador
Este paso es para purificar o seleccionar el tamaño del producto del paso 2 Con perlas magnéticas. La purificación permite eliminar residuos de adaptadores, dímeros de adaptadores u otros productos inutilizables.
Limpiarnorte compuesto de ADN ligado al adaptador
1. Preparación: tomar la prueba Hieff NGSMarca registrada Saque las perlas de selección de ADN del refrigerador y déjelas a temperatura ambiente durante al menos 30 minutos. Prepare etanol al 80 %.
2. Mezcle bien las perlas invirtiéndolas o colocándolas en el vértice.
3. Añadir 88 μL Hieff NGSMarca registrada Perlas de selección de ADN (0,8×, Perlas: ADN = 0,8:1) en el tubo que contiene el producto ligado al adaptador, agite y mezcle bien e incube a temperatura ambiente durante 5 minutos.
4. Centrifugue brevemente para diluir la solución y coloque el tubo de centrífuga en la gradilla magnética. Una vez que las perlas magnéticas se hayan absorbido por completo (aproximadamente 5 minutos), retire el líquido con cuidado.
5. Mantenga el tubo en el soporte magnético, Añada directamente al tubo 200 μL de etanol al 80 % recién preparado. Incube a temperatura ambiente durante 30 segundos y retire el líquido con cuidado.
6. Repetir Paso 5 nuevamente.
7. Mantenga el tubo en el soporte magnético, abra la tapa y seque las cuentas hasta que estén apenas agrietadas (no más de 5 minutos).
8. Retire el tubo del soporte magnético para la elución. y eluir el ADN
1). Si no es necesario seleccionar el tamaño del producto, agregue 21 μL de ddH2O directamente. Mezcle bien agitando con vórtex o pipeteando 10 veces. Incube a temperatura ambiente durante 5 minutos. Gire el tubo brevemente y colóquelo en un soporte magnético. Cuando la solución esté transparente (aproximadamente 5 minutos), transfiera 20 μL de sobrenadante a un nuevo tubo de PCR con cuidado, sin tocar las microesferas magnéticas.
2). Si es necesario seleccionar el tamaño del producto, agregue 102 μL de ddH2O directamente. Mezcle bien agitando con vórtex o pipeteando 10 veces. Incube a temperatura ambiente durante 5 minutos. Gire el tubo brevemente y colóquelo en un soporte magnético. Cuando la solución esté transparente (aproximadamente 5 minutos), transfiera 100 μL de sobrenadante a un nuevo tubo de PCR con cuidado, sin tocar las perlas magnéticas.
Nota:Si es necesario almacenar el producto purificado, se puede eluir con tampón TE.
Selección del tamaño del ADN ligado al adaptador
1.Preparación: tomar la prueba Hieff NGSMarca registrada Saque las perlas de selección de ADN del refrigerador y déjelas a temperatura ambiente durante al menos 30 minutos. Prepare etanol al 80 %.
2. Mezcle bien las perlas invirtiéndolas o colocándolas en el vértice.
3. En función de los tamaños deseados, agregue la primera ronda de perlas a las plantillas de ADN purificadas de 100 μL de acuerdo con la Tabla 11Mezcle bien agitando en vórtex o pipeteando 10 veces.
Mesa 11 Proporciones recomendadas de perlas:ADN para la selección del tamaño según las perlas
| Tamaño de la biblioteca de ADN insertada | 150 - 250 pb | 200-300 pb | 300-400 pb | 400-500 pb |
| Tamaño final de la biblioteca de ADN | 250-350 pb | 350-450 pb | 450-550 pb | 550-650 pb |
| Relación de volumen en el 1 calle redondo (Cuentas:ADN) | 0,80× | 0,70× | 0,60× | 0.55× |
| Relación de volumen en el 2 Dakota del Norte redondo (Cuentas:ADN) | 0,20× | 0,20× | 0,20× | 0,15× |
Nota: "×" en la tabla indica el volumen de la muestra de ADN. Por ejemplo, si la longitud del inserto de la biblioteca es de 250 pb y el volumen de ADN de la muestra es de 100 μL, el volumen de microesferas magnéticas utilizado en la primera ronda de clasificación es de 0,7 × 100 μL = 70 μL; el volumen de microesferas magnéticas utilizado en la segunda ronda de clasificación es de 0,20 × 100 μL = 20 μL. El volumen de microesferas recomendado en la tabla corresponde al ADN ligado al adaptador. Si el procedimiento de selección de tamaño se realiza antes de la ligadura, consulte los protocolos de fabricación de Hieff NGS.Marca registrada Perlas de selección de ADN (Cat#12601).
4. Incubar a temperatura ambiente durante 5 minutos.
5. Gire el tubo brevemente y colóquelo sobre un soporte magnético. Cuando la solución esté transparente (aproximadamente 5 minutos), transfiera el sobrenadante a un nuevo tubo de PCR.
6. Añade la segunda ronda de cuentas de selección a la muestra del paso 5 según la Tabla 11Mezcle bien agitando en un vórtex o pipeteando hacia arriba y hacia abajo al menos 10 veces.
7. Incubar a temperatura ambiente durante 5 minutos.
8. Centrifugue brevemente para diluir la solución y coloque el tubo de centrífuga en la gradilla magnética. Una vez que las perlas magnéticas se hayan adsorbido por completo (aproximadamente 5 minutos), retire el líquido con cuidado.
9. Mantenga el tubo en el soporte magnético, Añada directamente al tubo 200 μL de etanol al 80 % recién preparado. Incube a temperatura ambiente durante 30 segundos y retire el líquido con cuidado.
10. Repetir paso 9 de nuevo.
11. Mantenga el tubo en el soporte magnético, abra la tapa y seque las cuentas hasta que estén apenas agrietadas (no más de 5 minutos).
12. Retire el tubo del soporte magnético para la elución y añadir directamente 21 μL de ddH2O. Mezcle bien mediante vórtex o pipeteo vertical e incube a temperatura ambiente durante 5 minutos. (Nota: Si necesita conservar el producto purificado, eluya con tampón TE). Centrifugue brevemente el tubo y colóquelo en un soporte magnético hasta que el líquido se aclare (aproximadamente 5 minutos). Transfiera con cuidado 20 μL de sobrenadante a un nuevo tubo de PCR sin tocar las microesferas.
Paso 4 Amplificación de la biblioteca
Este paso puede enriquecer los productos purificados o de tamaño seleccionado mediante amplificación por PCR.
1. Descongele los reactivos enumerados en la tabla 12, inviértalos y mézclelos bien y colóquelos en hielo para su uso posterior.
2. Ensamble la siguiente reacción en un tubo de PCR esterilizado.
Tabla 12 reacción de PCR de ADN ligado al adaptador sistema
| Componentes | Volumen (μL) |
| ADN ligado por adaptador | 20 |
| CanaceMarca registrada Mezcla de amplificación profesional | 25 |
| Mezcla de imprimación* | 5 |
| Total | 50 |
[Nota]: * si se utilizó el adaptador completo, Hieff NGSMarca registrada Mezcla de imprimación (
3. Mezcle suavemente pipeteando o agitando y centrifugue brevemente para obtener la solución.
4. Coloque el tubo en un termociclador y configure el programa de acuerdo con la tabla 13 para iniciar la amplificación.
Tabla 13 Programa de reacción de amplificación por PCR
| Temperatura | Tiempo | Ciclo |
| Calentar la tapa a 105°C | En | - |
| 98°C | 45 segundo | 1 |
| 98°C | 15 segundo | Consulte la tabla 6 |
| 60°C | 30 segundos | |
| 72°C | 30 segundos | |
| 72°C | 1 minuto | 1 |
| 4°C | Sostener | - |
Paso 5 Limpieza posterior a la amplificación/selección de tamaño
La limpieza Los pasos hacia arriba se refieren a paso 3. Hieff NGSMarca registrada Las perlas de selección de ADN (0,9×, perlas:ADN = 0,9:1) se utilizan para purificar el producto de PCR. Si necesita seleccionar el tamaño, consulte paso 3.
Paso 6 Control de Calidad de las Bibliotecas Finales
La calidad de la biblioteca construida generalmente se evalúa midiendo la concentración y la distribución del tamaño. Para más detalles, consulte la Nota 6.
Pago y seguridad
Su información de pago se procesa de forma segura. No almacenamos detalles de la tarjeta de crédito ni tenemos acceso a la información de su tarjeta de crédito.
Consulta
También te puede gustar




Preguntas frecuentes
El producto es solo para fines de investigación y no está destinado a uso terapéutico o diagnóstico en humanos o animales. Los productos y el contenido están protegidos por patentes, marcas comerciales y derechos de autor propiedad de Yeasen Biotechnology. Los símbolos de marca comercial indican el país de origen, no necesariamente el registro en todas las regiones.
Algunas aplicaciones pueden requerir derechos de propiedad intelectual adicionales de terceros.
Yeasen se dedica a la ciencia ética y cree que nuestra investigación debe abordar cuestiones críticas al tiempo que garantiza la seguridad y los estándares éticos.