Descripción
Alto NGSMT. Universidad de California en Berkeley.A MÍ Las perlas de selección de ADN se preparan según el principio SPRI (inmovilización inversa en fase sólida) y es aplicable para la purificación de ADN y la selección de tamaño durante la preparación de bibliotecas de secuenciación de próxima generación (NGS). Alto NGSMT. Universidad de California en Berkeley.A MÍ Las perlas de selección de ADN son compatibles con varios kits de preparación de bibliotecas de ADN y ARN. Este producto se fabrica en una instalación ultra limpia con un alto nivel de limpieza y puede usarse para el paso de purificación de ADN durante los procesos de detección de patógenos.
Presupuesto
| Gato.norteo. | 17266ES03 /17266ES75 |
| Tamaño | 1 ml/450 ml |
Componentes
| Componentes No. | Nombre | 17266ES03 | 17266ES75 |
| 17266 | Alto NGS UCF.A MÍ Perlas de selección de ADN | 1 ml | 450 ml |
Almacenamiento
Este producto debe almacenarse en 2~8℃ durante 18 meses.
Envío y almacenamiento
Las perlas se envían con bolsas de hielo y se pueden almacenar a una temperatura entre 2 °C y 8 °C durante un año.
Instrucciones
- 1. Preparación
Equilibrar las perlas de selección a temperatura ambiente durante al menos 30 minutos antes de su uso.
- 2. Selección del tamaño del ADN
El flujo de operación de selección de tamaño se muestra en la Figura 1 y el protocolo es el siguiente.
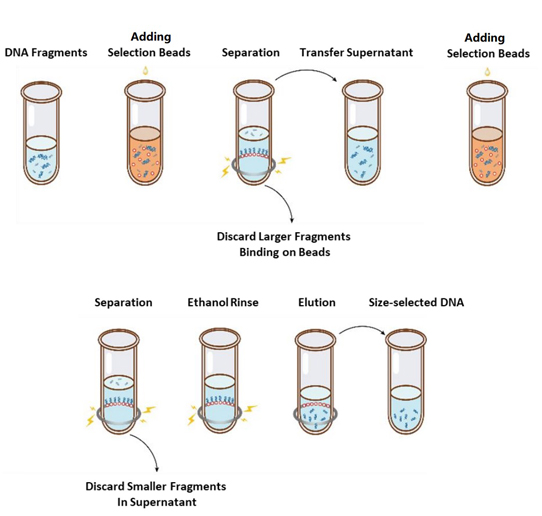
Figura 1. Diagrama de flujo de la selección del tamaño del ADN
2.1 Mezcle las cuentas a fondo vorando o pipetizando hacia arriba y hacia abajo cada vez antes de usar.
2.2 Agregue la primera ronda de perlas de selección a la muestra (consulte la Tabla 1). Mezcle bien agitando o pipeando hacia arriba y hacia abajo al menos 10 veces.
2.3 incubar a temperatura ambiente durante 5 min.
2.4 Gire por el tubo brevemente y colóquelo en un soporte magnético. Cuando la solución esté clara (aproximadamente 5 minutos), transfiera el sobrenadante a un nuevo tubo de PCR.
2.5 Agregue la segunda ronda de perlas de selección a la muestra del paso 2.4 Según la Tabla 1. Mezcle bien agitando o pipeando hacia arriba y hacia abajo al menos 10 veces.
2.6 Incubar a temperatura ambiente durante 5 min.
2.7 Gire por el tubo brevemente y colóquelo en un soporte magnético. Cuando la solución es clara (aproximadamente 5 minutos), aspira al sobrenadante y descarte.
2.8 Mantenga el tubo en el soporte magnético y agregue 200 μl de etanol al 80% recién preparado sin alterar las perlas, incubar a temperatura ambiente durante 30 segundos. Aspira al etanol y descarte.
2.9 Repita el paso 2.8 una vez para un total de dos lavados.
2.10 Retire el etanol residual con 10 µl de puntas de pipeta. Mantenga el tubo en el soporte magnético, seca el aire las perlas de selección con la tapa abierta hasta que aparezcan grietas (aproximadamente 5 minutos).
Nota: No seque demasiado las cuentas seleccionadas.Esto MayresultinLowerRecoveryDnatarget.
2.11 Retire el tubo del soporte magnético. Agregue una cantidad apropiada de DDH2O (≥20 µl) y mezcle bien vortando o pipetizando hacia arriba y hacia abajo al menos 10 veces.
2.12 incubar a temperatura ambiente durante 5 min.
Gire el tubo brevemente y colóquelo sobre el soporte magnético. Cuando la solución esté clara (aproximadamente 5 minutos), transfiera 20 μl del sobrenadante a un nuevo tubo.
- 3.Condiciones recomendadas para la selección del tamaño del ADN
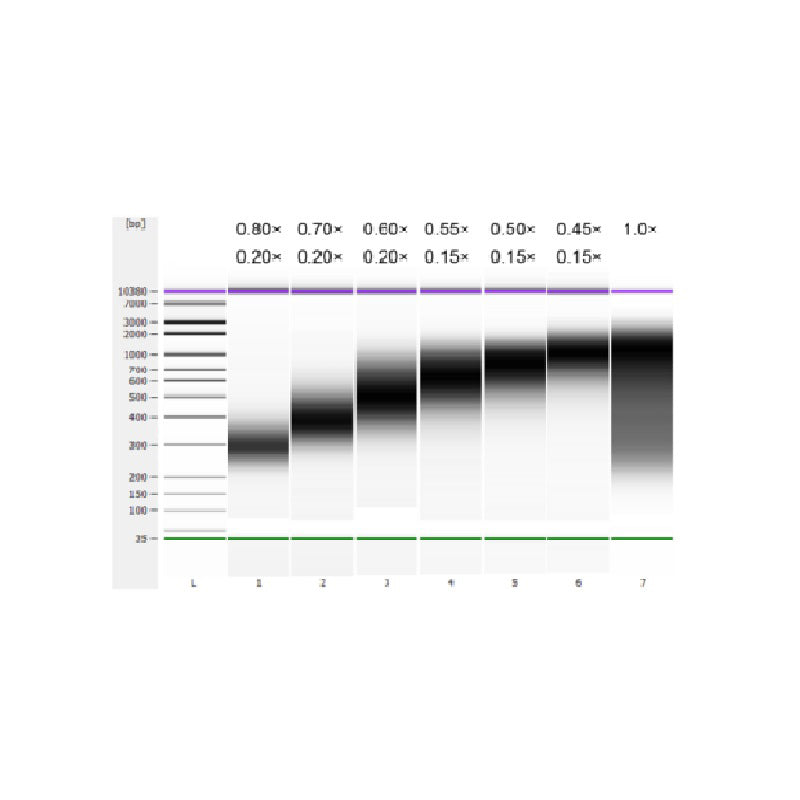
El ADN del timo de ternera se fragmentó mediante sonicación para preparar un fragmento de 100-1.000 pb y se realizaron dos rondas de selección de tamaño de acuerdo con la Tabla 1. Los resultados se analizaron utilizando el bioanalizador Agilent 2100 (Figura 2).
Tabla 1. Condición recomendada para la selección del tamaño del ADN
| Longitud del fragmento de ADN | 250-350 pb | 320-420 pb | 450-550 pb | 550-700 pb | 700-900 pb | 800-1.000 pb |
| Relación de cuentas: ADN para la 1ª ronda | 0.80× | 0.70× | 0.60× | 0.55× | 0.50× | 0.45× |
| Relación de cuentas: ADN para el 2do Redondo | 0.20× | 0.20× | 0.20× | 0.15× | 0.15× | 0.15× |
Nota: "×" en la tabla indica el volumen de muestra de ADN. Por ejemplo, si la longitud de inserción de la biblioteca es de 250 pb y el volumen de ADN de la muestra es de 100 μl, el volumen de perlas magnéticas utilizadas en la primera ronda de clasificación es 0.80 × 100 μl = 80 μl; El volumen de cuentas magnéticas utilizadas en la segunda ronda de clasificación es 0.20 × 100 μl = 20 μl.
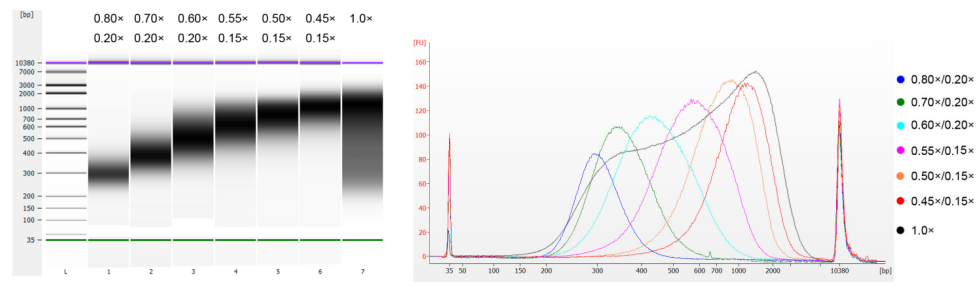
Figura 2. Electroferograma con chip de ADN de alta sensibilidad Agilent 2100
Notas:1. Para su seguridad y salud, use abrigos de laboratorio y guantes desechables para su funcionamiento.
Pago y seguridad
Su información de pago se procesa de forma segura. No almacenamos detalles de la tarjeta de crédito ni tenemos acceso a la información de su tarjeta de crédito.
Consulta
También te puede gustar
Preguntas frecuentes
El producto es solo para fines de investigación y no está destinado a uso terapéutico o diagnóstico en humanos o animales. Los productos y el contenido están protegidos por patentes, marcas comerciales y derechos de autor propiedad de Yeasen Biotechnology. Los símbolos de marca comercial indican el país de origen, no necesariamente el registro en todas las regiones.
Algunas aplicaciones pueden requerir derechos de propiedad intelectual adicionales de terceros.
Yeasen se dedica a la ciencia ética y cree que nuestra investigación debe abordar cuestiones críticas al tiempo que garantiza la seguridad y los estándares éticos.