Descripción
El kit de detección de micoplasma GMyc-PCR utiliza principalmente el método PCR para detectar la infección por micoplasma en diversos materiales biológicos (como cultivos celulares, secreciones de animales de experimentación, suero animal, etc.). Combina varias ventajas: es sensible, específico, rápido y se puede detectar directamente con sobrenadantes de cultivos celulares. Este producto detecta micoplasma en materiales biológicos como células cultivadas mediante el método PCR. Los cebadores utilizados están diseñados de acuerdo con la región conservada de la secuencia de ARNr 16S-23S del micoplasma y solo amplifican específicamente el ADN del micoplasma, con alta sensibilidad y especificidad de detección. La amplificación por PCR y el análisis por electroforesis solo toman unas pocas horas y la operación es conveniente y simple.
El cultivo celular es un experimento común en la investigación de las ciencias biológicas. A diferencia de otros métodos experimentales de uso común, el cultivo celular es un proceso continuo y dinámico, y las células a menudo responden a errores de manipulación o contaminantes que a menudo presentan estados celulares o apariencia anormales del medio. Si está contaminado por micoplasma, la morfología celular no presenta cambios obvios y es fácil pasarlo por alto. A menudo no se detecta hasta que la contaminación es muy grave. Puede haber cientos de micoplasmas en la membrana celular contaminada, estos micoplasmas compiten por los nutrientes y liberan metabolitos tóxicos, lo que afecta gravemente los resultados experimentales.
Los estudios han demostrado que al menos 20 tipos de micoplasma pueden contaminar las células, entre los que los más comunes son: Mycoplasma oral (M. orale), Mycoplasma arginina (M. arginini), Mycoplasma hyorhinis (M. hyorhinis), Micoplasma fermentum (M. fermentans), Mycoplasma hominis (M. hominis), Mycoplasma salivarius (M. salivarium), Mycoplasma pulmonar (M. pulmonis) y Mycoplasma pera (M. pirum). La tasa de contaminación por micoplasma de las células cultivadas varía entre el 4% y el 92%. Las fuentes de contaminación incluyen el entorno de trabajo, el propio operador (algunos micoplasmas son flora normal del cuerpo humano), el medio de cultivo, el suero, la contaminación cruzada de células, el equipo experimental y la contaminación del tejido u órgano original a partir del cual se prepararon las células.
Identificar la causa subyacente de los problemas durante el cultivo celular es una tarea difícil y que requiere mucho tiempo, en la que se deben sospechar los cambios repentinos y es necesario aplicar buenas prácticas de análisis y realizar análisis periódicos para detectar la contaminación por micoplasma. Existen muchos métodos para la detección de micoplasma, como el cultivo directo, la tinción de fluorescencia de ADN, la ELISA y los métodos de PCR.
Característica
- Los cebadores utilizados fueron diseñados de acuerdo con la región conservada de la secuencia del ARNr 16S-23S de Mycoplasma.
- Sólo el ADN de micoplasma amplificado específicamente
- Excelente sensibilidad y especificidad.
- Se pudieron detectar más de 20 especies de micoplasma
Solicitud
- Detección de micoplasma
Componentes
| Componentes No. | Nombre | 40601ES10 (10 ensayos) | 40601ES20 (20 ensayos) |
| 40601-A | Mezcla de PCR GMyc-1st | 250 µL | 2×250 µL |
| 40601-B | Mezcla de PCR GMyc-2nd | 250 µL | 2×250 µL |
| 40601-C | Plantilla de control positivo N | 20 µL | 20 µL |
[Notas] 1. Cuando no se utiliza durante un tiempo prolongado, se puede almacenar congelado a una temperatura de -85 a -65 ℃.
2. La reacción de PCR es extremadamente sensible.Para evitar falsos positivos, al agregar muestras se añade un control positivo al final.
Almacenamiento
Este producto puede almacenarse a una temperatura de entre -25 y -15 ℃ durante 18 meses. Si no se utiliza durante un tiempo prolongado, manténgalo alejado de la luz.
Cifras
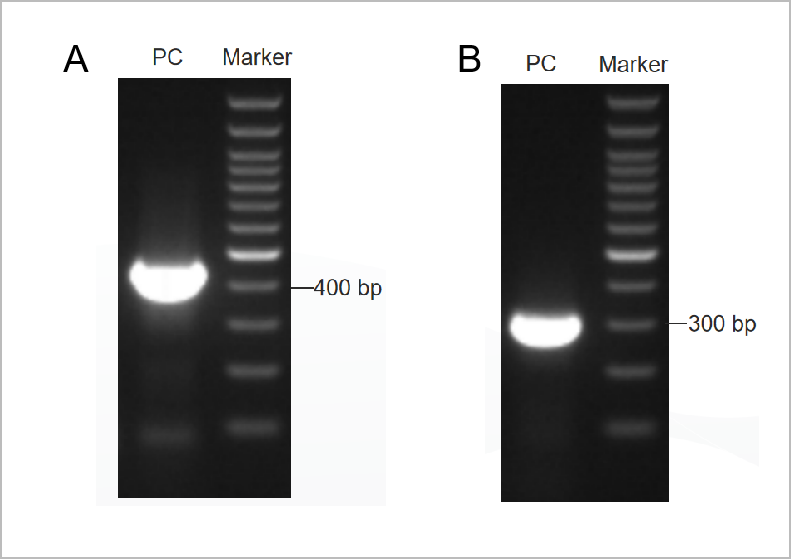
Figura 1. Resultado del electroforetograma del kit de prueba PCR Mycoplama.
El electroforetograma de la primera ronda se muestra en la Figura 1A y el electroforetograma de la segunda ronda se muestra en la Figura 1B. (M: marcador de 1 kb, 1-2: controles positivos, 3: control negativo)
[1] Rao XS, Cong XX, Gao XK, et al. La fosforilación mediada por AMPK mejora la autoinhibición de TBC1D17 para promover la captación de glucosa dependiente de Rab5. Cell Death Differ. 2021;28(12):3214-3234. doi:10.1038/s41418-021-00809-9(IF:15.828)
[2] Guo F, Li L, Li J, et al. Secuenciación multiómica de células individuales de embriones tempranos y células madre embrionarias de ratón. Cell Res. 2017;27(8):967-988. doi:10.1038/cr.2017.82(IF:15.606)
[3] Hao Y, He B, Wu L, et al. La translocación nuclear de p85β promueve la tumorogénesis del cáncer con mutante del dominio helicoidal de PIK3CA. Nat Commun. 2022;13(1):1974. Publicado el 13 de abril de 2022. doi:10.1038/s41467-022-29585-x(IF:14.919)
[4] Shu X, Liu M, Lu Z, et al. El mapeo de todo el genoma revela que la desoxiuridina está enriquecida en el ADN centromérico humano. Nat Chem Biol. 2018;14(7):680-687. doi:10.1038/s41589-018-0065-9(IF:13.843)
[5] Li X, Xiong X, Wang K, et al. El mapeo de todo el transcriptoma revela un metiloma de N(1)-metiladenosina reversible y dinámico. Nat Chem Biol. 2016;12(5):311-316. doi:10.1038/nchembio.2040(IF:12.709)
[6] Sun L, Yang X, Huang X, et al. La 2-hidroxilación de ácidos grasos reprime la tumorigénesis y la metástasis colorrectal a través del eje transcripcional YAP. Cancer Res. 2021;81(2):289-302. doi:10.1158/0008-5472.CAN-20-1517(IF:12.701)
[7] Sun Z, Zhang Z, Wang QQ, Liu JL. La inactivación combinada de CTPS1 y ATR es letal sintéticamente para las células cancerosas que sobreexpresan MYC. Cancer Res. 2022;82(6):1013-1024. doi:10.1158/0008-5472.CAN-21-1707(IF:12.701)
[8] Song J, Zhuang Y, Zhu C, et al. Funciones diferenciales de la PUS10 humana en el procesamiento de miRNA y la pseudouridilación de ARNt. Nat Chem Biol. 2020;16(2):160-169. doi:10.1038/s41589-019-0420-5(IF:12.154)
[9] He B, Pan H, Zheng F, et al. El ARN largo no codificante LINC00930 promueve la glucólisis tumoral y la proliferación celular mediadas por PFKFB3 en el carcinoma nasofaríngeo. J Exp Clin Cancer Res. 2022;41(1):77. Publicado el 24 de febrero de 2022. doi:10.1186/s13046-022-02282-9(IF:11.161)
[10] Tang B, Liu BH, Liu ZY, Luo MY, Shi XH, Pang DW. Puntos cuánticos con un recubrimiento zwitteriónico anfifílico compacto. ACS Appl Mater Interfaces. 2022;14(24):28097-28104. doi:10.1021/acsami.2c04438(IF:9.229)
[11] Huang C, Zhang Z, Chen L, et al.La acetilación en los dominios N y C-terminales de Src regula distintas funciones de la tumorigénesis mediada por STAT3. Cancer Res. 2018;78(11):2825-2838. doi:10.1158/0008-5472.CAN-17-2314(IF:9.130)
[12] Wu X, Yu M, Zhang Z, et al. DDB2 regula la replicación del ADN a través de la degradación independiente de PCNA de CDT2. Cell Biosci. 2021;11(1):34. Publicado el 8 de febrero de 2021. doi:10.1186/s13578-021-00540-5(IF:7.133)
[13] Wang J, Zhang Y, Liu X, Liu H. Optimización de la terapia adaptativa en función de la accesibilidad a la subpoblación resistente a los tumores. Cancers (Basel). 2021;13(21):5262. Publicado el 20 de octubre de 2021. doi:10.3390/cancers13215262(IF:6.639)
[14] Feng W, Liu R, Xie X, et al. La sumoilación de la α-tubulina es una nueva modificación que regula la dinámica de los microtúbulos. J Mol Cell Biol. 2021;13(2):91-103. doi:10.1093/jmcb/mjaa076(IF:6.216)
[15] Yu M, Hu X, Yan J, Wang Y, Lu F, Chang J. El inhibidor de RIOK2 NSC139021 ejerce efectos antitumorales en el glioblastoma mediante la inducción de la detención del ciclo celular y la apoptosis mediadas por Skp2. Biomedicines. 2021;9(9):1244. Publicado el 17 de septiembre de 2021. doi:10.3390/biomedicines9091244(IF:6.081)
[16] Ren S, Cai Y, Hu S, et al. La berberina ejerce una actividad antitumoral en el linfoma difuso de células B grandes mediante la modulación del eje c-myc/CD47. Biochem Pharmacol. 2021;188:114576. doi:10.1016/j.bcp.2021.114576(IF:5.858)
[17] Wen F, Sun X, Sun C, et al. La degradación proteosomal mediada por TRAF6 regula negativamente la expresión de TAGLN en células de cáncer de próstata. Mol Cancer Res. 2021;19(7):1113-1122. doi:10.1158/1541-7786.MCR-20-0513(IF:5.852)
[18] Tang B, Sun EZ, Zhang ZL, et al. El dominio de colesterol secuestrado por esfingomielina recluta la proteína de unión a formina 17 para la constricción de fosas recubiertas de clatrina en la entrada del virus de la influenza. J Virol. 2022;96(5):e0181321. doi:10.1128/JVI.01813-21(IF:5.103)
[19] Hu J, Ren W, Qiu W, et al. Generación de una línea de células madre pluripotentes inducidas (XDCMHi001-A) a partir de un paciente con espondilitis anquilosante con mutación JAK2. Stem Cell Res. 2020;45:101788. doi:10.1016/j.scr.2020.101788(IF:4.495)
[20] Xiao S, Yao X, Ye J, Tian X, Yin Z, Zhou L. La modificación epigenética facilita la expresión aberrante de la prolina sintasa PYCR1 en el cáncer gástrico [publicado en línea antes de la impresión, 30 de mayo de 2022]. Biochim Biophys Acta Gene Regul Mech. 2022;1865(6):194829. doi:10.1016/j.bbagrm.2022.194829(IF:4.490)
[21] Wang J, Zhang Y, Liu X, Liu H. ¿Es eficaz el tratamiento periódico fijo para el sistema tumoral sin información completa? Cancer Manag Res. 2021;13:8915-8928. Publicado el 30 de noviembre de 2021. doi:10.2147/CMAR.S339787(IF:3.989)
[22] Yang X, Ren S, Rehman ZU, et al. Caracterización molecular, expresión e identificación funcional de la quinasa de unión a TANK 1 (TBK1) de la vaca (Bos taurus) y la cabra (Capra hircus). Dev Comp Immunol. 2022;133:104444. doi:10.1016/j.dci.2022.104444(IF:3.636)
[23] Zheng D, Chang X, Liu Y, et al. El 2-metoxi-5((3,4,5-trimetoxifenil)seleninil)fenol revierte la migración e invasión celular inducida por EGF a través de la regulación negativa de MDM2 en líneas celulares de cáncer de mama. Cancer Biol Ther. 2019;20(4):513-523. doi:10.1080/15384047.2018.1537578(IF:3.373)
[24] Xu F, Zhang S, Liu Z, et al. TEX9 y eIF3b actúan en sinergia funcional para promover la progresión del carcinoma de células escamosas esofágico. BMC Cancer. 2019;19(1):875. Publicado el 3 de septiembre de 2019. doi:10.1186/s12885-019-6071-9(IF:2.933)
[25] Pan H, Sun L, Wang W, et al. ARN largo no codificante sérico LOC553103 como biomarcador diagnóstico y pronóstico no específico para tipos comunes de cáncer humano. Clin Chim Acta. 2020;508:69-76. doi:10.1016/j.cca.2020.05.017(IF:2.615)
[26] Li N, Lin SM, Li Y, Sun J, Zhang L, Chen M.Una línea de células madre pluripotentes inducidas (GZHMCi004-A) derivada de un feto con mutación heterocigótica G380R en el gen FGFR3 que causa acondroplasia. Stem Cell Res. 2021;53:102322. doi:10.1016/j.scr.2021.102322(IF:2.020)
[27] Luo Q, Wei C, Long Y, et al. Generación de una línea de células madre embrionarias humanas con ELTD1 inactivado mediante el sistema iCRISPR/Cas9. Stem Cell Res. 2021;53:102350. doi:10.1016/j.scr.2021.102350(IF:2.020)
[28] Liu YQ, Ling TW, Wang HY, Yang YH, Song WJ, Wang TC. Generación de una línea de células madre pluripotentes inducidas sin integración (LZUSHI001-A) a partir de un paciente epiléptico con mutación DGKG. Stem Cell Res. 2022;61:102768. doi:10.1016/j.scr.2022.102768(IF:2.020)
[29] Chen M, Lin SM, Li N, Li Y, Li Y, Zhang L. Una línea de células madre pluripotentes inducidas (GZHMCi003-A) derivada de un feto con deleción heterocigótica del exón 3 en el gen RUNX2 que causa displasia cleidocraneal. Stem Cell Res. 2021;51:102166. doi:10.1016/j.scr.2021.102166(IF:2.020)
[30] Xu Y, Wang X, Qiu T, et al. Generación de una línea de células madre pluripotentes inducidas (FDCHI007-A) derivada de un paciente con encefalopatía epiléptica y del desarrollo tipo 31 portador del gen c.545C heterocigoto. > Una mutación en el gen DNM1. Stem Cell Res. 2022;60:102709. doi:10.1016/j.scr.2022.102709(IF:2.020)
[31] Fan T, He J, Wang Y, Yu J, Sun W. Generación de una línea de células madre pluripotentes inducidas (FDCHi006-A) a partir de una niña de 7 años con pubertad precoz central. Stem Cell Res. 2021;56:102542. doi:10.1016/j.scr.2021.102542(IF:2.020)
[32] Gong X, Zheng Z, Yang T, Zheng H, Xiao X, Jia N. Generación de una línea de células madre pluripotentes inducidas (iPSC) corregida genéticamente isogénicamente (OGHFUi001-A-1) a partir de un paciente con encefalopatía epiléptica infantil temprana tipo 1 (EIEE1) con una mutación hemicigótica R330L en el gen ARX. Stem Cell Res. 2022;60:102693. doi:10.1016/j.scr.2022.102693(IF:2.020)
[33] Jia N, Gong X, Chen J, et al. Generación de una línea de células madre pluripotentes inducidas (OGHFUi001-A) a partir de una encefalopatía epiléptica infantil temprana tipo 1 con mutación ARX. Stem Cell Res. 2021;53:102367. doi:10.1016/j.scr.2021.102367(IF:2.020)
[34] Zhu W, Zhou Y, Wang Q, et al. Generación de una línea de células madre pluripotentes inducidas (iPSC) humanas a partir de fibroblastos de la piel de un paciente portador de una mutación E363Q en el gen PSEN1. Stem Cell Res. 2022;61:102769. doi:10.1016/j.scr.2022.102769(IF:2.020)
[35] Luo F, Long K, Li X, et al. La deficiencia de LRRC8A atenúa la necrosis inducida por hipoxia en células 3T3-L1. Biosci Biotechnol Biochem. 2020;84(6):1139-1145. doi:10.1080/09168451.2020.1730689(IF:1.516)
Pago y seguridad
Su información de pago se procesa de forma segura. No almacenamos detalles de la tarjeta de crédito ni tenemos acceso a la información de su tarjeta de crédito.
Consulta
También te puede gustar
Preguntas frecuentes
El producto es solo para fines de investigación y no está destinado a uso terapéutico o diagnóstico en humanos o animales. Los productos y el contenido están protegidos por patentes, marcas comerciales y derechos de autor propiedad de Yeasen Biotechnology. Los símbolos de marca comercial indican el país de origen, no necesariamente el registro en todas las regiones.
Algunas aplicaciones pueden requerir derechos de propiedad intelectual adicionales de terceros.
Yeasen se dedica a la ciencia ética y cree que nuestra investigación debe abordar cuestiones críticas al tiempo que garantiza la seguridad y los estándares éticos.

