Descripción
E. coli El kit de detección de residuos de ADN de células huésped se utiliza para el análisis cuantitativo de E. coli El ADN de la célula huésped se reduce en muestras intermedias, productos semiacabados y terminados de diversos productos biológicos.
Este kit adopta la sonda fluorescente Taqman y el método de reacción en cadena de la polimerasa (PCR), que tiene un límite de detección mínimo de nivel fg y puede detectar de manera específica y rápida la E. coli residual. ADN celular. El kit debe utilizarse junto con el kit de preparación de muestras de ADN residual (n.° de cat. 18461ES).
Presupuesto
| Cat.No. | 41308ES50-ES / 41308ES60-ES |
| Tamaño | 50 DIEZ / 100 T-ES |
Componentes
| Componentes No. | Nombre | 41308ES50-ES | 41308ES60-ES |
| 41308-A | E. coli Mezcla de qPCR | 0,75 ml | 1.5 ml |
| 41308-B | E. coli Mezcla de cebadores y sondas | 250 μL | 500 μL |
| 41308-C | Tampón de dilución de ADN | 2×1,8 ml | 4×1,8 ml |
| 41308-D | E. coli Control de ADN (30 ng/μL) | 25 μL | 50 μl |
Almacenamiento
Este producto debe almacenarse a una temperatura entre -25 y -15 °C durante 2 años.
Tanto el 41308-A como el 41308-B deben almacenarse protegidos de la luz.
Modelos de instrumentos aplicables
Incluyen, entre otros:
Bio-Rad: Módulo óptico CFX96.
Thermo Scientific: ABI 7500; ABI Quant Studio 5.
Instrucciones
- E. coli. Dilución estándar de ADN y preparación de curva estándar
La bacteria E. coli El control de ADN se diluyó en gradiente utilizando el tampón de dilución de ADN proporcionado en el kit.*, y la dilución
La concentración es 300 pg/μL, 30 pg/μL, 3 pg/μL, 300 fg/μL, 30 fg/μL.
Vea las instrucciones detalladas a continuación:
- Descongele el control de coliDNA y el tampón de dilución de ADN en hielo. Una vez que se hayan descongelado por completo, agite suavemente para mezclar y centrifugue a baja velocidad durante 10 segundos.
- Saque seis tubos limpios de 1,5 ml, marcados con Std0, Std1, Std2, Std3, Std4, Std5.
- Agregue 90 μL de tampón de dilución de ADN y 10 μL de control coliDNA al tubo de microcentrífuga de 1,5 mL etiquetado como Std0, es decir Diluir a 3 ng/μL. Mezclar y luego centrifugar durante 10 segundos. Envasar el estándar de ADN diluido en subpaquetes y almacenarlo a corto plazo (no más de 3 meses) a -25~-15℃.**Evite la congelación y descongelación repetidas.
- Agregue 90 μL de tampón de dilución de ADN a otros tubos***, luego siga el procedimiento a continuación para las diluciones seriadas****.
| Tubo | Relación de dilución | Concentración estándar |
| Norma 1 | 10 μL de Std0 + 90 μL de tampón de dilución de ADN | 300 pg/μL |
| Estándar 2 | 10 μL de Std1 + 90 μL de tampón de dilución de ADN | 30 pg/μL |
| Estándar 3 | 10 μL de Std2 + 90 μL de tampón de dilución de ADN | 3 pg/μL |
| Estándar 4 | 10 μL de Std3 + 90 μL de tampón de dilución de ADN | 300 fg/μL |
| Estándar 5 | 10 μL de Std4 + 90 μL de tampón de dilución de ADN | 30 fg/μL |
Tabla 1 Dilución en gradiente estándar
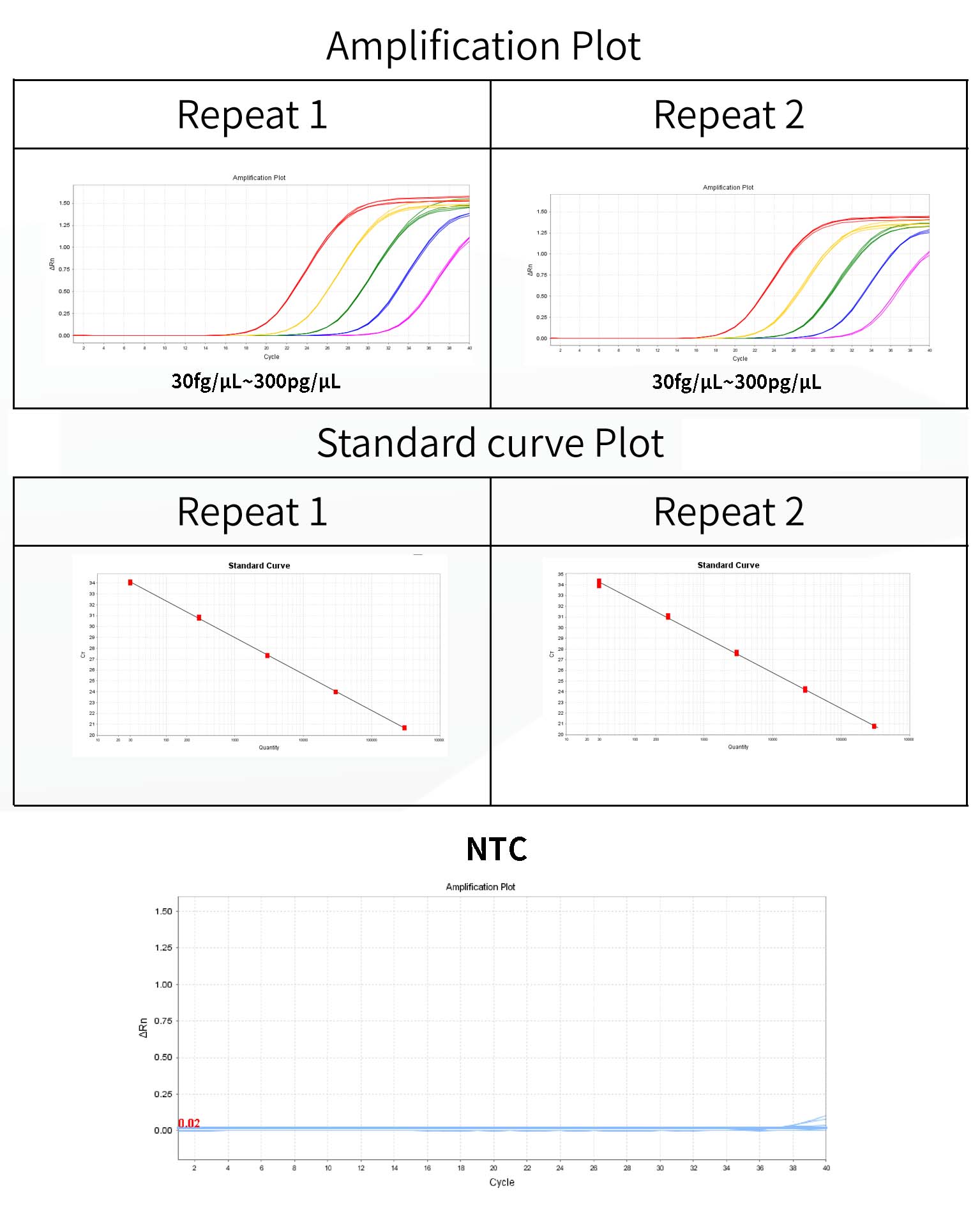
*Se requieren tres pocillos replicados para cada concentración. El rango de detección es 30 fg/μL~300pg/μL y este rango se puede ampliar.
**Para reducir la cantidad de congelaciones y descongelaciones repetidas y evitar la contaminación, se recomienda almacenar el control de ADN en alícuotas a -25~-15℃ por primera vez.
***Una vez descongelado, el tampón de dilución de ADN se puede almacenar a 2-8 °C durante 7 días. Si no se utiliza durante un tiempo prolongado, guárdelo a -25 ~ -15 ℃.
****Asegúrese de que la plantilla esté completamente mezclada, agite suavemente la mezcla durante 15 segundos a 1 minuto por cada dilución de gradiente.
- Preparación del control de recuperación de extracción (ERC)
Establezca la concentración de ADN de E. coli en ERC según sea necesario (la muestra de ERC se preparó con 30 pg de ADN de E. coli como ejemplo), de la siguiente manera:
- Agregue 100 μL de muestra de prueba en un tubo limpio de 1,5 mL, luego agregue 10 μL de estándar de ADN de E. coli 3pg/μL (Std3) y mezcle bien, marcado como ERC.
- Realice la extracción de ADN de la muestra de ERC junto con las muestras de prueba para preparar la muestra de ERC purificada.
- Preparación de la solución de control negativo (NCS)
Establezca el control negativo en el experimento, los pasos de operación específicos son los siguientes:
1) Agregue 100 μL de matriz de muestra (o tampón de dilución de ADN) en un tubo limpio de 1,5 mL, luego marcado como NCS.
2) Realice la extracción de ADN de la muestra de NCS junto con las muestras de prueba para preparar la muestra de NCS purificada.
- Preparación sin control de plantilla (NTC)
Establezca el control sin plantilla en el experimento, los pasos de operación específicos son los siguientes:
1) NTC no requiere pretratamiento de la muestra y se puede configurar en la etapa de detección de ADN residual mediante qPCR.
2) La muestra de NTC en cada tubo o pocillo es de 20 μL de mezcla (es decir, 15 μL de mezcla de qPCR de E. coli + 5 μL de mezcla de cebador y sonda de E. coli) + 10 μL de tampón de dilución de ADN. Se recomienda configurar tres pocillos replicados.
- Sistema de reacción PCR
| Componente | Volumen (μL) |
| E. coli Mezcla de qPCR* | 15 |
| MI.coli Mezcla de cebadores y sondas | 5 |
| Plantilla de ADN | 10 |
| Volumen total** | 30 |
Tabla 2 Sistema de reacción
*Calcule el volumen total de la reacción de PCR mediante el número de reacciones: qPCR Mix = (el número de reacciones + 2) × (15 + 5) μL (incluidas las pérdidas de dos pocillos de reacción). Se recomiendan más de tres réplicas para cada muestra en el experimento.
**Después de tapar el tubo o sellar la placa, centrifugue el tubo o la placa de reacción a baja velocidad durante 10 segundos. Después de agitar y mezclar lo suficiente durante 5 segundos, repita la centrifugación para recoger el líquido de la tapa o la pared hasta el fondo. Evite que se formen burbujas durante la operación.
Consulte la siguiente tabla para ver la configuración de placa recomendada:
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | CNT |
| TS 1 | TS 1 | TS 1 |
| Norma 1 | Norma 1 | Norma 1 |
|
|
|
| B | CNT |
| TS 2 | TS 2 | TS 2 |
| Norma 2 | Norma 2 | Norma 2 |
|
|
|
| do | CNT |
| TS 3 | TS 3 | TS 3 |
| Norma 3 | Norma 3 | Norma 3 |
|
|
|
| D |
|
|
|
|
|
| Norma 4 | Norma 4 | Norma 4 |
|
|
|
| mi | CN |
| CERC 1 | CERC 1 | CERC 1 |
| Norma 5 | Norma 5 | Norma 5 |
|
|
|
| F | CN |
| CERC 2 | CERC 2 | CERC 2 |
|
|
|
|
|
|
|
| GRAMO | CN |
| CEI 3 | CEI 3 | CEI 3 |
|
|
|
|
|
|
|
| yo |
|
|
|
|
|
|
|
|
|
|
|
|
Tabla 3 Computadora en placa de referencia
El diseño de la placa incluye: 5 Std (la curva estándar de 5 concentraciones estándar), 1 NTC (control sin plantilla), 1 NCS (solución de control negativo), 3 TS (muestras de prueba), 3 ERC (control de recuperación de extracción).Tres pocillos replicados para cada muestra.
- Configuración Pautas para un instrumento PCR(Método de 2 pasos) (p. ej., instrumento de qPCR Thermo ABI 7500, software versión 2.0)
Las siguientes instrucciones se aplican únicamente al instrumento de qPCR Thermo ABI 7500 (versión de software 2.0). Si utiliza un instrumento diferente, consulte la guía del instrumento correspondiente para obtener las pautas de configuración.
1) Genere un nuevo experimento, elija la plantilla de cuantificación absoluta o definida por el usuario.
2) Cree una sonda de detección, denominada "E. coli-DNA", seleccione el fluoróforo indicador como "FAM" y el fluoróforo de extinción como "Ninguno". La fluorescencia de referencia es "ROX" (la fluorescencia de referencia puede basarse en el modelo del instrumento, etc., seleccione si necesita agregarla).
3) En el panel "Muestras", agregue toda la información de las muestras una por una. Luego, seleccione los pocillos, elija el objetivo y las muestras correspondientes. Configure la tarea de E. coli Estándar de ADN como estándar y asigne los valores 300000, 30000, 3000, 300, 30 (la unidad de concentración de ADN en cada pocillo es fg/μL) en la columna Cantidad y nombre los pocillos Std. 1, estándar 2, estándar 3, estándar 4, estándar 5. De acuerdo con esto, configure la tarea de NTC como NTC. Configure NCS, TS y ERC. como Desconocido y nómbrelos según el diseño de placa anterior. Luego haga clic en Siguiente.
4) Establezca el programa de amplificación: establezca el volumen de reacción en 30 μL.
| Paso del ciclo | Temperatura (℃) | Tiempo | Ciclos |
| Desnaturalización inicial | 95℃ | 10 minutos | 1 |
| Desnaturalización | 95℃ | 15 segundos | 40 |
| Recocido/extensión (recolección de fluorescencia) | 60℃ | 30 segundos |
Tabla 4 Procedimiento de amplificación
- Análisis de los resultados de qPCR
1) El sistema proporcionará automáticamente el umbral en el panel Gráfico de amplificación del análisis. El umbral proporcionado por el sistema a veces está demasiado cerca de la línea base, lo que genera una gran diferencia en Ct entre los pocillos replicados. Puede ajustar manualmente el umbral a una posición adecuada y hacer clic en Analizar. Luego, puede verificar inicialmente si la curva de amplificación es normal en el Gráfico de componentes múltiples.
2) En la pestaña Análisis de resultados, revise el gráfico de la curva estándar. Verifique los valores de R2, Eficiencia, Pendiente y la intersección con Y. Para una curva estándar normal, R²>0,99, 90%≤Eff%≤110%, -3,6≤Pendiente≤-3,1.
3) En el panel 'Ver tabla de pocillos' en Análisis, las concentraciones de cada muestra se muestran en Cantidad, la unidad es fg/μL, las unidades se pueden convertir en el informe del ensayo.
4) Los ajustes de los parámetros del análisis de resultados deben basarse en el modelo específico y la versión del software utilizados y, generalmente, el instrumento puede interpretarlos automáticamente.
5) Calcule la tasa de recuperación de picos basándose en los resultados de la prueba de la muestra TS que se va a medir y la recuperación de picos de la muestra ERC; la tasa de recuperación de picos debe estar entre el 50 % y el 150 %.Fórmula del medidor de tasa de recuperación con picos: Recuperación (%) = {Sample spiked assay (eg.pg/μL) - Sample assay (eg.pg/μL)} x Volumen de elución (μL) / Valor teórico de la cantidad de adición de ADN (por ejemplo, pg) x 100%.
6) El valor Ct del control negativo NCS debe ser mayor que la media de la concentración Ct más baja del estándar.
- El control NTC sin plantilla debe ser indeterminado o el valor Ct ≥3
Notas
- Este producto es solo para uso de investigación.
- Por favor trabaje con bata de laboratorio y guantes desechables, para su seguridad.
3. Lea atentamente este manual antes de utilizar este reactivo. El experimento debe estar estandarizado, incluida la manipulación de la muestra, la preparación del sistema de reacción y la adición de la muestra.
4. Asegúrese de que cada componente esté completamente agitado y centrifugado a baja velocidad antes de su uso.
Pago y seguridad
Su información de pago se procesa de forma segura. No almacenamos detalles de la tarjeta de crédito ni tenemos acceso a la información de su tarjeta de crédito.
Consulta
También te puede gustar
Preguntas frecuentes
El producto es solo para fines de investigación y no está destinado a uso terapéutico o diagnóstico en humanos o animales. Los productos y el contenido están protegidos por patentes, marcas comerciales y derechos de autor propiedad de Yeasen Biotechnology. Los símbolos de marca comercial indican el país de origen, no necesariamente el registro en todas las regiones.
Algunas aplicaciones pueden requerir derechos de propiedad intelectual adicionales de terceros.
Yeasen se dedica a la ciencia ética y cree que nuestra investigación debe abordar cuestiones críticas al tiempo que garantiza la seguridad y los estándares éticos.