Descripción
Se utiliza el kit de detección de lentivirus con capacidad de replicación (RCL) para detectar cuantitativamente lentivirus replicantes que Puede ocurrir en una Variedad de productos celulares asociados con lentivirus. Vectores de riesgos potenciales.
Este kit diseña primers específicos para el Secuencia del gen VSV-G de las proteínas de la envoltura lentiviral. Y adopta taqman sonda fluorescente y el método de reacción en cadena de la polimerasa (PCR), que tiene un límite de detección de nivel de 1 copias/μL y Puede detectar de forma específica y rápida El riesgo de lentivirus con capacidad de replicación. El kit necesita para usar junto con el kit de preparación de muestras de ADN residual (Cat. n.° 18461ES).
Componentes
| Componentes No. | Nombre | 41311ES50 | 41311ES60 |
| 41311-A | Mezcla de PCR cuantitativa de RCL | 0,75 ml | 1.5 ml |
| 41311-B | Mezcla de cebadores y sondas RCL | 200 microlitros | 400 microlitros |
| 41311-C | Dilución de ADN Buffer | 2 x 1,8 ml | 4 x 1,8 ml |
| 41311-D | Control de ADN RCL (5×10E8) copias/μL)) | 25 microlitros | 50 microlitros |
| 41311-E | CI* | 50 microlitros | 100 microlitros |
* CI: Interno control.
Almacenamiento
Este producto debe almacenarse a una temperatura entre -25 y -15 ℃. para 2 años.
Tanto el 41311-A como el 41311-B deben almacenarse protegidos de la luz.
Aplicable instrumento modelos
Incluyen, entre otros: Bio-Rad: Módulo óptico CFX96; Thermo Scientific: Peso bruto: 7500 Estudio cuantitativo ABI 5; Paso ABI OnePlus.
Instrucciones
- ADN RCL Estándar dilución y Estándar curva preparación
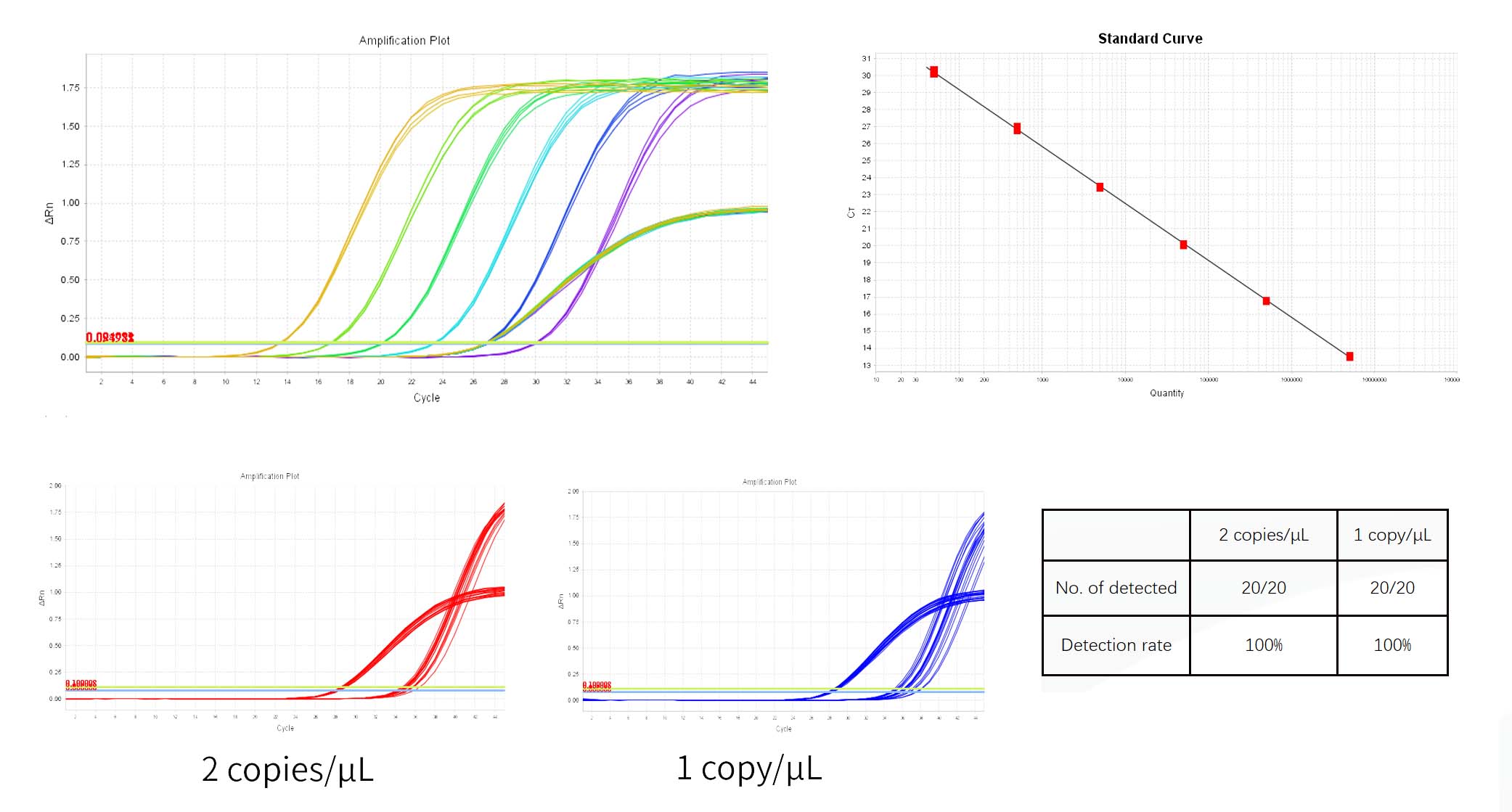
El control de ADN RCL se diluyó en gradiente utilizando el tampón de dilución de ADN proporcionado en el kit* , y la dilución La concentración es 5×10E7 copias/μL, 5×10E6 copias/μL, 5×10E5 copias/μL, 5×10E4 copias/μL, 5×10E3 copias/μL, 5×10E2 copias/μL, 5×10E1 copias/μL.
Vea las instrucciones detalladas a continuación:
1) Descongele el control de ADN RCL y el tampón de dilución de ADN en hielo. Después de descongelarse por completo, Agite suavemente para mezclar, y centrifugar a baja velocidad durante 10 segundos.
2) Saque siete tubos limpios de 1,5 ml, marcados con Std0, Std1, Std2, Std3, Std4, Std5, Std6.
3) Añadir 90 μL de tampón de dilución de ADN y 10 μL de control de ADN RCL al tubo de microcentrífuga de 1,5 mL etiquetado como Std0, a saber diluir a 5×10E7 copias/μL. Mezclar y Luego centrifugue durante 10 segundos. Empaquete el estándar de ADN diluido y podrá Almacenado a corto plazo (no más de 3 meses) a -25~-15℃** Evite la congelación y descongelación repetidas.
4) Añadir 90 μL de tampón de dilución de ADN en otros tubos*** , luego siga el procedimiento a continuación para Las diluciones seriadas**** .
| Tubo | Relación de dilución | Concentración estándar |
| Norma 1 | 10 μL Std0 + 90 μL de dilución de ADN Buffer | 5×10E6 copias/μL |
| Estándar 2 | 10 μL estándar 1 + 90 Dilución de ADN en μL Buffer | 5×10E5 copias/μL |
| Estándar 3 | 10 μL estándar 2 + 90 Dilución de ADN en μL Buffer | 5×10E4 copias/μL |
| Estándar 4 | 10 μL de Std3 + 90 μL de dilución de ADN Buffer | 5×10E3 copias/μL |
| Estándar 5 | 10 μL estándar 4 + 90 Dilución de ADN en μL Buffer | 5×10E2 copias/μL |
| Norma 6 | 10 μL estándar 5 + 90 μL de ADN Dilución Buffer | 5×10E1 copias/μL |
Tabla 1 Dilución en gradiente estándar
*Tres reproducir exactamente pozos son requerido para cada Concentración.La detección rango es 5×10E1 copias/μL~5×10E6 copias/μL y este rango poder ser Ampliado si es necesario.
** A reducir el número de repetir congelación-descongelación y evitar contaminación, es es recomendado a almacenar el ADN control en alícuotas en -25 ~ -15 ℃ para el primero tiempo.
*** Una vez descongelado, ADN dilución buffer podría ser almacenado en 2-8 °C para 7 días, si no usado para a largo tiempo por favor almacenar en -25 ~ -15 ℃ .
**** Hacer seguro el plantilla es completamente mezclado, suavemente agitar el mezcla para 15 segundos a 1 minuto para cada gradiente dilución.
- Recuperación de extracción Control (CRE) preparación
Establezca la concentración de ADN RCL en ERC según sea necesario (la muestra de ERC se preparó con 5×10E4 copia ADN RCL como un ejemplo), como sigue:
1) Añadir 100 Vierta μL de muestra de prueba en un tubo limpio de 1,5 mL y luego agregue 10 μL 5×10E3 copias/μL Estándar de ADN RCL (Std4) y mezclar bien, marcado como ERC.
2) Realice la extracción de ADN de la muestra de ERC junto con las muestras de prueba para preparar la muestra de ERC purificada.
- Control negativo Solución (NCS) preparación
Establezca el control negativo en el experimento, los pasos de operación específicos son los siguientes:
1) Añadir 100 μL de matriz de muestra (o tampón de dilución de ADN) en un tubo limpio de 1,5 mL, luego marcado como Estado Civil.
2) Realizar la extracción de ADN de NCS Muestra junto con las muestras de prueba para preparar el NCS purificado muestra.
- Sin plantilla Control (CNT) preparación
Establezca el control sin plantilla en el experimento, los pasos de operación específicos son los siguientes:
1) NTC no requiere pretratamiento de la muestra y se puede configurar en la etapa de detección de ADN residual mediante qPCR. contenido.
2) La muestra de NTC en cada tubo o pocillo es de 20 μL de mezcla (es decir, 15 Mezcla de qPCR de RCL μL + 4 microlitros LCR Cebador y sonda Mezcla + 1 μL de CI) + 10 μL de tampón de dilución de ADN. Se recomienda configurar tres pocillos de réplica.
- PCR reacción sistema
| Componente | Volumen (μL) |
| Mezcla de PCR cuantitativa de RCL* | 15 |
| Mezcla de cebadores y sondas RCL | 4 |
| CI | 1 |
| Plantilla de ADN | 10 |
| Total volumen** | 30 |
Tabla 2 Sistema de reacción
* Calcular el total PCR reacción volumen por el número de Reacciones: qPCR Mezcla =(el número de reacciones+2) × (15+4+1) μL (incluido el pérdidas de dos pocillos de reacción). Se recomiendan más de tres réplicas para cada muestra en el experimento.
** Después tapado el tubo o caza de focas el placa, centrífuga el reacción tubo o lámina en bajo velocidad para 10 segundos después suficiente sacudida y mezclando para 5 segundos, repetir centrífugo a recolectar el líquido de el tapa o muro a el Abajo. Evitar Burbujas durante operación.
Consulte la siguiente tabla para ver la configuración de placa recomendada:
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | CNT |
| TS 1 | TS 1 | TS 1 |
| estándar 1 | estándar 1 | estándar 1 |
|
|
|
| B | CNT |
| TS 2 | TS 2 | TS 2 |
| estándar 2 | estándar 2 | estándar 2 |
|
|
|
| do | CNT |
| TS 3 | TS 3 | TS 3 |
| estándar 3 | estándar 3 | estándar 3 |
|
|
|
| D |
|
|
|
|
|
| estándar 4 | estándar 4 | estándar 4 |
|
|
|
| mi | CN |
| CEI 1 | CEI 1 | CEI 1 |
| estándar 5 | estándar 5 | estándar 5 |
|
|
|
| F | CN |
| CERC 2 | CERC 2 | CERC 2 |
| estándar 6 | estándar 6 | estándar 6 |
|
|
|
| GRAMO | CN |
| CEI 3 | CEI 3 | CEI 3 |
|
|
|
|
|
|
|
| yo |
|
|
|
|
|
|
|
|
|
|
|
|
Tabla 3 Computadora encendida referencia junta
El diseño de la placa incluye: 6 Std (la curva estándar de 6 concentraciones estándar), 1 NTC (sin control de plantilla), 1 NCS (solución de control negativo), 3 TS (muestras de prueba), 3 ERC (control de recuperación de extracción).Tres pozos replicados para Cada muestra.
- Pautas de configuración para a PCR Instrumento
Las siguientes instrucciones se aplican únicamente a Termo Instrumento qPCR ABI 7500 (Software versión 2.0). Si tu usas un Para instrumentos diferentes, consulte la guía del instrumento correspondiente para obtener pautas de configuración.
1) Generar un nuevo experimento, elegir la plantilla de cuantificación absoluta o definido por el usuario.
2) Cree 1 sonda de detección, denominada "RCL-DNA", seleccione el fluoróforo reportero como "FAM" y apague el fluoróforo como "ninguno"; crea 1 sonda de detección más, nómbrala "IC" y selecciona el fluoróforo reportero como "CY5" y apaga el fluoróforo. fluoróforo como "ninguno". La fluorescencia de referencia es ROX" (la fluorescencia de referencia puede basarse en la modelo de instrumento, etc., seleccionar si Necesitas agregarlo).
3) En el Panel 'Muestras', agregue toda la información de las muestras por turno. Luego, seleccione los pocillos, elija el objetivo y las muestras correspondientemente. la tarea del estándar de ADN RCL como estándar, y asignar el valores 5000000, 500000, 50000, 5000, 500, 50 (la unidad de concentración de ADN en cada pocillo es copias/μL) en la columna Cantidad y nombre el pozos Std 1, Std 2, Std 3, Std 4, Std 5, Std 6, respectivamente. Tarea de NTC como NTC. Establezca NCS, TS y ERC como Desconocido, y los nombró de acuerdo con el diseño de la placa anterior correspondientemente. Luego haga clic en siguiente.
4) Configure el programa de amplificación: establezca el volumen de reacción en 30 μL.
| Paso del ciclo | Temperatura (℃) | Tiempo | Ciclos |
| Digestión contaminada | 37℃ | 5 mín. | 1 |
| Desnaturalización inicial | 95℃ | 5 mín. | 1 |
| Desnaturalización | 95℃ | 15 segundo |
45 |
| Recocido/extensión (recolección de fluorescencia) | 60℃ | 30 segundos |
Tabla 4 Procedimiento de amplificación
- Análisis de PCR cuantitativa resultados
1) El sistema dará automáticamente el Umbral en el Panel de gráfico de amplificación de Análisis. El umbral dado A veces, el sistema está demasiado cerca de la línea base, lo que genera una gran diferencia. en Ct entre pocillos replicados. Puedes ajustarlo manualmente el Umbral a una posición apropiada y haga clic Analizar. Luego puedes comprobar inicialmente si la curva de amplificación es normal en el gráfico multicomponente.
2) En el resultado Pestaña Análisis, revise el gráfico de la curva estándar. Verificar el valores para R2, Eficiencia, Pendiente y Intersección con el eje Y. Para una curva estándar normal, R²>0,99, 90%≤Eff%≤110%, -3,6≤Pendiente≤-3,1.
3) En el 'Vista panel de la mesa bien Análisis, las concentraciones de cada muestra se muestran en Cantidad, La unidad son copias/μL, las unidades se pueden convertir en el informe del ensayo.
4) La configuración de los parámetros de El análisis de resultados debe basarse en el modelo específico y el software. versión utilizado y, generalmente, puede ser interpretado automáticamente por el instrumento.
5) Calcule la tasa de recuperación de picos en función de los resultados de la prueba de La muestra TS a medir y la punta de muestra recuperación ERC, la tasa de recuperación de Se requieren picos Estar entre 50%~150%. Fórmula del medidor de tasa de recuperación pico:
Recuperación (%) = {Sample spiked assay (eg.copies/μL) - Sample assay (eg.copies/μL)} x Volumen de elución (μL) / Teorético Valor de la cantidad de ADN añadido (por ejemplo, copias) x100%。
6) El Ct valor de el control negativo NCS debe ser mayor que la media de la concentración más baja Ct de el estándar.
7) Plantilla de control libre NTC Debe ser Indeterminado o Ct valor ≥38.
Notas
- Este producto es solo para uso de investigación.
- Por favor, trabaje con bata de laboratorio y guantes desechables, para su seguridad.
3. Lea atentamente este manual antes de usarlo. Este reactivo y el experimento deben estandarizarse, incluyendo manipulación de muestras, preparación del sistema de reacción y Adición de muestra.
4. Asegúrese de que cada componente esté completamente agitado y centrifugado a baja velocidad antes de su uso.
Pago y seguridad
Su información de pago se procesa de forma segura. No almacenamos detalles de la tarjeta de crédito ni tenemos acceso a la información de su tarjeta de crédito.
Consulta
También te puede gustar
Preguntas frecuentes
El producto es solo para fines de investigación y no está destinado a uso terapéutico o diagnóstico en humanos o animales. Los productos y el contenido están protegidos por patentes, marcas comerciales y derechos de autor propiedad de Yeasen Biotechnology. Los símbolos de marca comercial indican el país de origen, no necesariamente el registro en todas las regiones.
Algunas aplicaciones pueden requerir derechos de propiedad intelectual adicionales de terceros.
Yeasen se dedica a la ciencia ética y cree que nuestra investigación debe abordar cuestiones críticas al tiempo que garantiza la seguridad y los estándares éticos.