Description
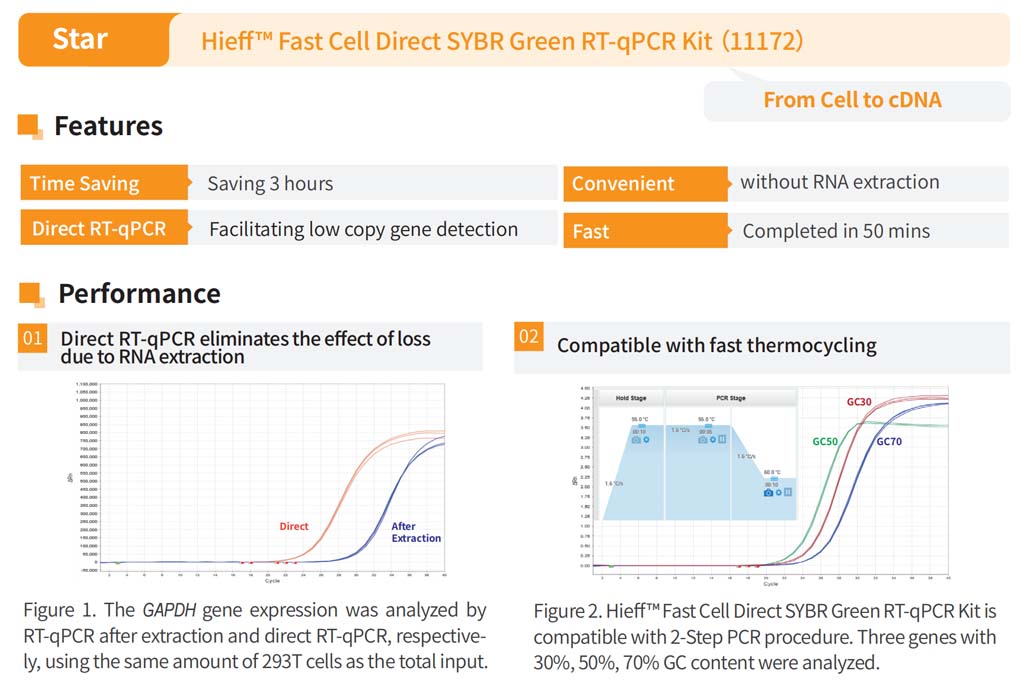
Chef™ Fast Cell Direct Le kit RT-qPCR basé sur le colorant vert SYBR convient à l'extraction d'ARN à partir de toutes sortes de cellules animales (telles que les cellules de paroi de lignée cellulaire et les cellules en suspension, les cellules de culture primaire, diverses cellules souches, les cellules iPS, etc.), sans avoir besoin d'extraire l'ARN, et peut être utilisé directement pour l'analyse de l'expression qPCR, qui est courte, facile à utiliser et présente un faible taux d'erreur. Il ne faut qu'une heure et demie pour réaliser les étapes allant de la préparation du modèle à la réaction de transcription inverse et à l'analyse de l'expression génétique.
Des réactifs de transcription inverse et de détection de fluorescence sont inclus dans le kit, qui peut être utilisé pour l'analyse de l'expression génétique sans qu'il soit nécessaire d'acheter des réactifs supplémentaires.
Caractéristiques
| N° de cat. | 11172ES40 / 11172ES60 |
| Taille | 40 T/100 T |
Composants
| Composants N° | Nom | 11172ES40 | 11172ES60 |
| 11172-A | Tampon de lyse FCD | 2 mL | 5 mL |
| 11172-B | Tampon de lavage FCD | 8 ml | 20 mL |
| 11172-C | Solution d'arrêt FCD | 100 μL | 250 μL |
| 11172-D | ADNase I | 80 μL | 200 μL |
| 11172-F | Mélange Hifair™ FCD RT 4× | 200 μL | 500 μL |
| 11172-F | Mélange maître SYBR qPCR FCD Hieff™ 2× | 2 ml | 5 ml |
| 11172-G | H sans RNase2O | 2 ml | 5 ml |
Stockage
Le tampon de lyse FCD et le tampon de lavage FCD ont été fondus et stockés à 4 °C pour éviter toute contamination. La solution d'arrêt FCD, la DNase I, le mélange 4× Hifair™ FCD RT, le mélange maître 2× Hieff™ FCD qPCR SYBR doivent être conservés entre -25 et -15 ℃.
Instructions
- Préparation des produits de clivage
1)1. Faire fondre le réactif à température ambiante, le retourner et le mélanger doucement avant utilisation, et l'utiliser après une légère centrifugation pour éviter la formation de mousse*.
*Le fait de ne pas mélanger les réactifs, d’utiliser un oscillateur pour le mélange et de ne pas configurer les réactifs sur la glace peut entraîner une diminution des performances de la réaction.
2) Selon le type de cellules**, transférer les cellules dans un tube à centrifuger et centrifuger à 5000 tr/min pendant 2 min pour recueillir les cellules et bien aspirer le milieu. Si les cellules sont cultivées dans des plaques à 96 puits, le milieu peut être aspiré directement.
3)Ajoutez 150 μL de tampon de lavage FCD à chaque puits, lavez les cellules en soufflant, centrifugez à 5 000 tr/min pendant 2 min et aspirez le tampon de lavage FCD***.
4)Ajoutez 48 μL de solution tampon de lyse FCD et 2 μL de solution de DNase Ⅰ dans chaque puits, soufflez et mélangez à température ambiante, puis laissez reposer pendant 5 minutes, puis ajoutez 2.5 μL de solution d'arrêt FCD après incubation ****, puis souffler et mélanger environ 5 fois pour obtenir le produit de clivage *****.
** L'exigence de base pour le nombre de cellules est de 1 × 104 cellules par puits, et ce kit peut être utilisé dans la gamme de 1 × 103 - 1 × 106 cellules. Si le nombre de cellules est plus grand, la quantité de solution tampon de lyse FCD et de solution de DNaseⅠ peut être augmentée proportionnellement selon le cas.
*** Les conditions de centrifugation varient d’une cellule à l’autre, veuillez donc centrifuger à une vitesse adaptée aux cellules utilisées.
**** Ajoutez 2,5 μL de solution d’arrêt FCD à 50 μL de lysat et augmentez la quantité de solution d’arrêt FCD selon les besoins.
***** Pour le stockage à long terme des solutions de produits de lyse cellulaire, placer à -20°C.
5) Faire fondre 4 x Hifair™ FCD RT Mix à température ambiante et mélanger avec une légère inversion, placer sur de la glace et configurer le système de réaction selon le tableau ci-dessous :
| Composants | Volume (μL) | Concentration finale |
| Mélange Hifair™ FCD RT 4× | 5 | 1× |
| produit de clivage****** | x | x |
| H sans RNase2O | En haut à 20 | - |
*******Le niveau d'utilisation recommandé est de 2 à 5 μL, essayez de ne pas dépasser 45 %.
- Transcription inverse
Pipeter et mélanger délicatement la solution de réaction préparée ci-dessus et effectuer la réaction de transcription inverse selon la procédure du tableau ci-dessous :
| Température | Temps (min) |
| 55℃* | 15 min |
| 85℃ | 5 min |
* La température de transcription inverse recommandée est de 55°C. Pour les modèles à teneur élevée en GC ou les modèles complexes, la température de transcription inverse peut être augmentée à 60°C. Le produit de transcription inverse peut être utilisé directement pour la détection RT-qPCR en aval. Afin d'éviter l'inhibition de la réaction qPCR par le système de transcription inverse et d'obtenir la valeur Ct appropriée (10-35), le produit peut être dilué 10 à 1000 fois puis utilisé. Si les expériences en aval ne sont pas effectuées pendant une courte période, il peut être placé à -20℃ pour le stockage.
- PCR quantitative par fluorescence
1) Réaction système configuration
Les ratios suivants sont recommandés pour la préparation de la solution réactionnelle (préparation sur glace).
| Composants | Volume (μL) | Concentration finale |
| Mélange maître SYBR qPCR FCD Hieff™ 2× | 10 | 1× |
| Amorce directe (10 μmol/L) | 0,4 | 0,2 μmol/L |
| Amorce inverse (10 μmol/L) | 0,4 | 0.2 μmol/L |
| Produit de transcription inverse* | x | - |
| H sans RNase2O | En haut à 20 | - |
* Ne pas ajouter plus de 1/10 du volume de RT-qPCR du produit de transcription inverse. Une concentration élevée de matrice entraînera facilement une amplification non spécifique, dilution appropriée de 5 à 50 fois. La quantité recommandée de matrice est de 4 μL, essayez de ne pas dépasser 6 μL. Lorsque les performances de la réaction sont médiocres, la concentration de l'amorce peut être ajustée dans la plage de 0,2 à 1,0 μmol/L.
2) Procédure d'amplification PCR quantitative par fluorescence (méthode en deux étapes)
| Faire du vélo étape | Temp. | Temps | Cycles |
| Dénaturation initiale | 95℃ | 30 seconde | 1 |
| Dénaturation | 95℃ | 10 seconde | 35-40 |
| Recuit/Extension* | 60℃ | 30 seconde | |
| Étape de la courbe de fusion | Valeurs par défaut de l'instrument | 1 | |
3) Procédure d'amplification rapide pour PCR quantitative fluorescente (méthode en deux étapes)
| Faire du vélo étape | Temp. | Temps | Cycles |
| Dénaturation initiale | 95℃ | 10 seconde | 1 |
| Dénaturation | 95℃ | 5 seconde | 40 |
| Recuit/Extension* | 60℃ | 10 seconde | |
| Étape de la courbe de fusion | Valeurs par défaut de l'instrument | 1 | |
* La température de recuit/d'extension et le temps d'extension final peuvent être ajustés de manière appropriée en fonction des exigences expérimentales. Le programme rapide convient à la plupart des gènes, et le programme standard peut être essayé pour des gènes individuels à structure secondaire complexe.
Remarques
- Ce produit est destiné à la recherche uniquement.
- Veuillez travailler avec des blouses de laboratoire et des gants jetables, pour votre sécurité.
Version EN20230908
Documents:
11172-Hieff™ Fast Cell Direct. EN20230908.pdf
Paiement et sécurité
Vos informations de paiement sont traitées en toute sécurité. Nous ne stockons pas les détails de la carte de crédit ni accès aux informations de votre carte de crédit.
Enquête
Vous pouvez aussi aimer
FAQ
Le produit est destiné à des fins de recherche uniquement et n'est pas destiné à un usage thérapeutique ou diagnostique chez l'homme ou l'animal. Les produits et le contenu sont protégés par des brevets, des marques déposées et des droits d'auteur appartenant à Yeasen Biotechnology. Les symboles de marque indiquent le pays d'origine, pas nécessairement l'enregistrement dans toutes les régions.
Certaines applications peuvent nécessiter des droits de propriété intellectuelle tiers supplémentaires.
Yeasen se consacre à la science éthique, estimant que nos recherches doivent répondre à des questions cruciales tout en garantissant la sécurité et les normes éthiques.