Description
Hieff NGSMT OnePot Pro DNA Library Prep Kit V3 est un kit de préparation de bibliothèque basé sur la fragmentation enzymatique de nouvelle génération spécialement développé et conçu pour Illumina Plateforme de séquençage &MGI. Par rapport aux méthodes traditionnelles de construction de bibliothèques, ce produit utilise des enzymes de fragmentation de haute qualité, éliminant ainsi le processus ultrasonique fastidieux. Il simplifie l'opération en combinant les modules de fragmentation et de réparation des extrémités en un seul. De plus, les enzymes et le tampon du module de ligature sont pré-mélangés, ce qui réduit considérablement le temps et le coût de construction de la bibliothèque. Cela le rend plus adapté à la construction automatisée de bibliothèques. Ce kit de préparation de bibliothèque a un excellent taux de conversion de bibliothèque et est applicable aux échantillons de tous les animaux, plantes, micro-organismes, etc. courants, ainsi qu'aux échantillons FFPE. Sur la base de la génération précédente de kit de construction de bibliothèque, ce produit présente une efficacité supérieure en matière de fragmentation, réparation des extrémités, queue dA et ligature de l'adaptateur par rapport aux versions précédentes. L'enzyme haute fidélité améliore considérablement l'uniformité et la fidélité de l'amplification.
Caractéristiques
| N° de cat. | 12194ES08 / 12194ES24 / 12194ES96 |
| Taille | 8 T / 24 T / 96 T |
Composants
| Composants N° | Nom | 12194ES08 | 12194ES24 | 12194ES96 |
| 12194-A | FrottisMT Tampon 3.0 | 80 μL | 240 μL | 960 μL |
| 12194-B | FrottisMT Enzyme 3.0 | 80 μL | 240 μL | 960 μL |
| 12194-C | Mélange prêt à l'emploi pour ligature | 200 μL | 600 μL | 3×800 μL |
| 12194-D | 2× Ultima HF Amplification Mix | 200 μL | 600 μL | 3×800 μL |
[Note]: Les composants du kit sont compatibles avec les deux Illumina &MGI plateforme de séquençage, si l'adaptateur complet a été utilisé, Hieff NGSMT Un mélange d'apprêt (Yeasen Cat#12190 ou Cat#12191) est nécessaire.
Stockage
Ce produit doit être conservé entre -25 et -15 ℃ pendant 1 année.
Remarques
1. À propos de l'opération
1. Veuillez travailler avec des blouses de laboratoire et des gants jetables,pour votre sécurité.
2. Décongeler les composants à température ambiante. Après décongélation, bien mélanger au vortex, faire tourner brièvement le tube et le placer sur de la glace pour une utilisation ultérieure.
3. Lors de la préparation de la solution de réaction de chaque étape, il est recommandé d'utiliser une pipette pour bien mélanger ou agiter doucement. Une agitation vigoureuse peut entraîner une diminution du rendement de la bibliothèque.
4. Il est fortement recommandé d'utiliser des embouts de pipette filtrés pour éviter toute contamination croisée. Veillez à changer les embouts de pipette lors du traitement de différents échantillons.
5. Des opérations incorrectes peuvent très probablement provoquer des contaminations par aérosol, affectant la précision des résultats. Il est recommandé d'isoler physiquement les zones de mélange de réaction PCR et les zones d'essai de purification des produits PCR. Equipé d'équipements tels que des pipettes spécialisées pour la construction de bibliothèques. Effectuez un nettoyage de routine pour chaque zone en essuyant les surfaces avec de l'hypochlorite de sodium à 0,5 % ou de l'eau de Javel à 10 %
6. Ce produit est destiné à la recherche uniquement.
2. Fragmentation de l'ADN
1. Le kit est compatible avec 100 pg - 1 000 ng d'ADN d'entrée. Il est fortement recommandé d'utiliser de l'ADN d'entrée de haute qualité avec A260/A280 = 1,8-2,0.
2. Les expériences suivantes pourraient être affectées si des concentrations élevées de sels comme l'agent chélateur des métaux étaient introduites avec l'ADN d'entrée. Nous recommandons d'éluer l'échantillon d'ADN dans ddH2O pour la fragmentation.
3. Veuillez vous référer au tableau 6 pour le temps de fragmentation des échantillons d'ADN standard. Le kit présente un faible biais de fragmentation et fournit une couverture GC uniforme pour les échantillons d'ADN avec une large gamme de compositions GC. Veuillez ajuster le temps de fragmentation en fonction de vos besoins expérimentaux.
4. Pour une fragmentation précise, veuillez préparer la réaction sur de la glace.
3. Ligature de l'adaptateur
1. Des kits d'adaptateurs longs (adaptateurs à code-barres) Illumina ou MGI et des kits d'adaptateurs courts sont disponibles pour que les clients puissent choisir en fonction de leurs besoins expérimentaux.
2. Il a été recommandé de sélectionner des adaptateurs commerciaux de haute qualité. Si vous choisissez des adaptateurs fabriqués par vos soins, veuillez faire appel à une entreprise expérimentée dans la synthèse d'amorces NGS et notez la nécessité d'un contrôle strict de la contamination. De plus, il est recommandé de préparer la solution de recuit d'ADN dans un banc propre et d'utiliser uniquement un type d'adaptateur à la fois pour éviter toute contamination croisée.
3. Veuillez décongeler les adaptateurs sur la glace ou à 4°C ; en fonctionnement à température ambiante, la température du laboratoire ne doit pas dépasser 25°C pour éviter la dénaturation des adaptateurs.
4. La qualité et la concentration des adaptateurs affecteront directement l'efficacité de la ligature et le rendement de la bibliothèque. Une concentration trop élevée d'adaptateurs favorise la formation de dimères d'adaptateurs tandis qu'une concentration trop faible d'adaptateurs réduit le taux de ligature et le rendement de la bibliothèque. Dilutions correspondantes avec le tampon TE en fonction de la quantité d'ADN d'entrée lors de l'utilisation de l'adaptateur. Tableau 1-2 répertorie les méthodes de dilution recommandées pour les adaptateurs conventionnels et UMI pour différentes quantités d'ADN d'entrée à l'aide de ce kit pour les plates-formes de séquençage Illumina ou MGI.
Tableau 1 L'Illumina recommandé quantité d'adaptateur pour différentes entrées ADN
| Saisir ADN | Crapport de dilution de l'adaptateur conventionnel | Concentration | Rapport de dilution de l'adaptateur UMI | Concentration |
| <1 ng | 7,5 fois | 2 µM | 15 fois | 1 µM |
| 1 n ~ 10 ng | 3 volets | 5 µM | 3 volets | 5 μM |
| 10 n ~ 200 n | 1,5 fois | 10 µM | 2 volets | 7,5 μM |
| >200 ng | 0-Fois | 15 μM | 0-Fois | 15 μM |
Tableau 2 Le recommandé MGI quantité d'adaptateur pour différentes entrées ADN
| Saisir ADN | Crapport de dilution de l'adaptateur conventionnel | Concentration | Rapport de dilution de l'adaptateur UMI | Concentration |
| <1 ng | 5 fois | 2 µM | 10 fois | 1 µM |
| 1 n ~ 10 ng | 2 volets | 5 µM | 2 volets | 5 μM |
| 10 n ~ 200 n | 0-Fois | 10 µM | 1.25 fois | 8 µM |
| >200 ng | 0-Fois | 10 µM | 0-Fois | 10 µM |
4. Nettoyage et sélection de la taille de l'ADN à partir de billes
1. La sélection de la taille de l'ADN peut être effectuée avant la réparation de l'extrémité/queue dA, après la ligature de l'adaptateur ou après l'amplification.
2. Il est recommandé d’effectuer une sélection de taille juste après la ligature de l’adaptateur si la quantité d’ADN d’entrée est supérieure à 50 ng ; sinon, veuillez effectuer la sélection de taille après amplification.
3. Le Ligature Enhancer contient une concentration élevée de PEG, ce qui peut avoir un impact significatif sur la sélection précise de la taille. Ainsi, si la sélection de la taille doit être effectuée juste après la ligature de l'adaptateur, il est fortement recommandé d'ajouter une étape de nettoyage des billes avant la sélection de la taille. L'étape de sélection de la taille peut être effectuée directement si elle est effectuée avant la réparation de l'extrémité/la queue dA ou après l'amplification de la bibliothèque.
4. Les billes magnétiques doivent être équilibrées à température ambiante avant utilisation, sinon le rendement diminuera et l'effet de sélection de taille sera affecté.
5. Les billes magnétiques doivent être bien mélangées par vortex ou pipetage avant utilisation.
6. N'aspirez pas les billes lors du transfert du surnageant, même des traces de billes peuvent avoir un impact sur les réactions suivantes.
7. L'éthanol à 80 % doit être fraîchement préparé, sinon cela affectera l'efficacité de la récupération.
8. Pour une sélection précise de la taille, il est recommandé de commencer avec un volume supérieur à 100 μL. Si le volume est inférieur, il est recommandé d'augmenter le volume jusqu'à 100 μL avec de l'eau ultra pure.
9. Les billes magnétiques doivent être séchées à température ambiante avant l'élution du produit. Une sécheresse insuffisante peut facilement entraîner un effet résiduel de l'éthanol sur les réactions ultérieures ; une sécheresse excessive peut provoquer la fissuration des billes magnétiques et réduire le rendement de purification. Normalement, un séchage à température ambiante pendant 3 à 5 minutes suffit pour permettre aux billes de sécher complètement.
10. Si nécessaire, les échantillons d'ADN purifiés ou sélectionnés en fonction de la taille sont élués dans 0,1× Le tampon TE peut être conservé à 4°C pendant 1 à 2 semaines ou à -20°C pendant un mois.
5. Amplification de la bibliothèque
1. La nécessité ou non d'effectuer une amplification de la bibliothèque dépend de la quantité d'ADN d'entrée, des types d'adaptateurs, des applications des données de séquençage, etc. L'étape d'amplification est nécessaire si des adaptateurs partiels sont utilisés. Lorsque des adaptateurs pleine longueur sont utilisés, si l'ADN d'entrée est < 200 ng, il est recommandé d'effectuer une amplification ; sinon, l'amplification n'est pas nécessaire.
2. Le nombre de cycles d'amplification doit être strictement contrôlé. Une amplification insuffisante peut entraîner un faible rendement de la bibliothèque. Une suramplification peut entraîner une augmentation des biais, des erreurs, des lectures dupliquées et des produits chimériques. Tableau 3 répertorie les numéros de cycles recommandés ciblant le rendement de la bibliothèque de 1 μg.
Tableau 3 Le nombre de cycles recommandé pour générer Rendement de la bibliothèque : 1 000 ng
| Entrée ADN | Nombre de cycles nécessaires pour générer 1 μg de rendement de bibliothèque |
| 1000-2000 n | 2 - 4 |
| 500 ng | 2 - 4 |
| 250 ng | 4 - 6 |
| 100 ng | 5 - 7 |
| 50 ng | 7 - 9 |
| 10 ng | 9 - 11 |
| 5 ng | 10 - 12 |
| 1 ng | 12 - 15 |
| 100 pages | 16 - 18 |
Note
1.Table 3 montre le nombre de paramètres de boucle en utilisant des tests ADN d'entrée de haute qualité d'environ 200 pb. La qualité de l'ADN FFPE varie considérablement et lorsque la qualité de l'ADN est médiocre ou que la longueur de la bibliothèque est longue, le nombre de cycles doit être augmenté de manière appropriée pour obtenir des bibliothèques suffisantes.
2. Si la sélection de la taille est requise pendant le processus de création de la bibliothèque, un nombre de cycles plus élevé pour l'amplification de la bibliothèque est recommandé ; sinon, un nombre de cycles inférieur est recommandé.
3. Si des adaptateurs incomplets sont utilisés, au moins 2 cycles doivent être amplifiés pour former un adaptateur complet.
6. Analyse de la qualité de la bibliothèque
1. La qualité des bibliothèques construites est généralement analysée en mesurant les concentrations et les distributions de taille.
2. Les concentrations des bibliothèques peuvent être mesurées par des méthodes basées sur la fluorescence telles que Qubit et PicoGreen ou qPCR.
3. Il n'est PAS recommandé d'utiliser des méthodes de quantification basées sur l'absorbance telles que NanoDrop.
4. Il est recommandé d'utiliser la méthode qPCR pour la quantification des bibliothèques : les méthodes basées sur la fluorescence telles que Qubit et PicoGreen ne peuvent pas différencier les structures d'ADN double brin incomplètes (inserts sans adaptateur ou avec une seule des extrémités ligaturée avec un adaptateur) des bibliothèques complètes. La méthode qPCR n'amplifiera et ne mesurera que les bibliothèques complètes avec les deux extrémités ligaturées avec des adaptateurs (les bibliothèques séquencables), offrant ainsi une mesure plus précise pour le chargement.
5. La distribution de taille des bibliothèques peut être analysée à l'aide d'Agilent Bioanalyzer ou d'autres appareils basés sur les principes de l'électrophorèse capillaire ou de la microfluidique.
7. Autres matériaux
1. Billes magnétiques pour la purification de l'ADN : Hieff NGSMT Billes de sélection d'ADN (Yeasen Cat#12601) ou AMPure® Perles XP (A63880) ou autres produits équivalents.
2.Adaptateurs : Adaptateur complet pour Illumina : Yeasen Cat#13519-13520 ; 384 amorces doubles CDI : Yeasen Chat#12412~Chat#12413; 384 amorces uniques à double index (UDI) : Yeasen Chat#12312~Chat#12315; Adaptateurs UMI UDI : Yeasen Cat#13370~Cat#13371 ; Adaptateur complet pour MGI : Yeasen Cat#13360-13362. Mélange d'amorces d'ADN:Cat#12190 ou Numéro de chat 12191.
3. Analyse de qualité de bibliothèque : Agilent 2100 Bioanalyzer DNA 1000 Chip/High Sensitivity Chip ou autres produits équivalents ; réactifs quantitatifs de bibliothèque.
4. Autres matériels : éthanol absolu, eau ultra pure stérile, pointes de pipettes à faible rétention, tube PCR, supports magnétiques, thermocycleur, etc.
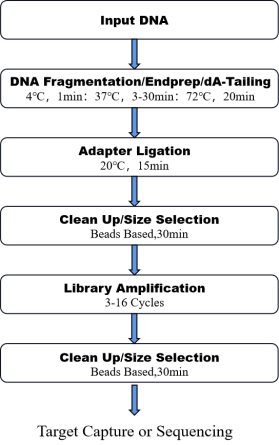
8. Flux de travail
Figure 1. Le flux de travail de Un pot Pro ADN Kit de préparation pour la bibliothèque
Chiffres
Les tailles des fragments d'insertion obtenus dans différentes conditions de fragmentation
En utilisant 500 ng d'ADNg standard comme modèle, des bibliothèques ont été construites avec ce kit. Les conditions de fragmentation étaient une digestion enzymatique à 32 °C, 35 °C et 37 °C pendant 5, 10, 15, 20 et 30 minutes respectivement. Les produits fragmentés ont été purifiés avec des billes magnétiques 1,2x et élués avec 21 μL de ddH2O. La concentration a été mesurée à l’aide de Qubit, et la distribution des fragments d’insertion récupérés est illustrée dans la figure suivante.
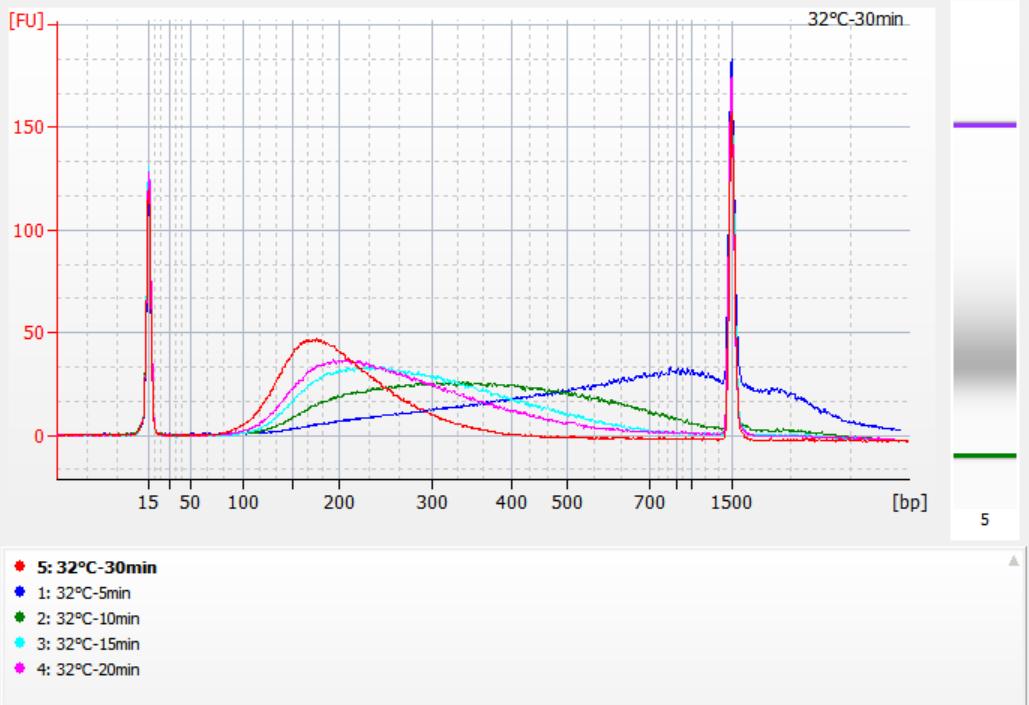 |
Figure 2. Profils de bibliothèque à 32°C pour différents temps de digestion enzymatique
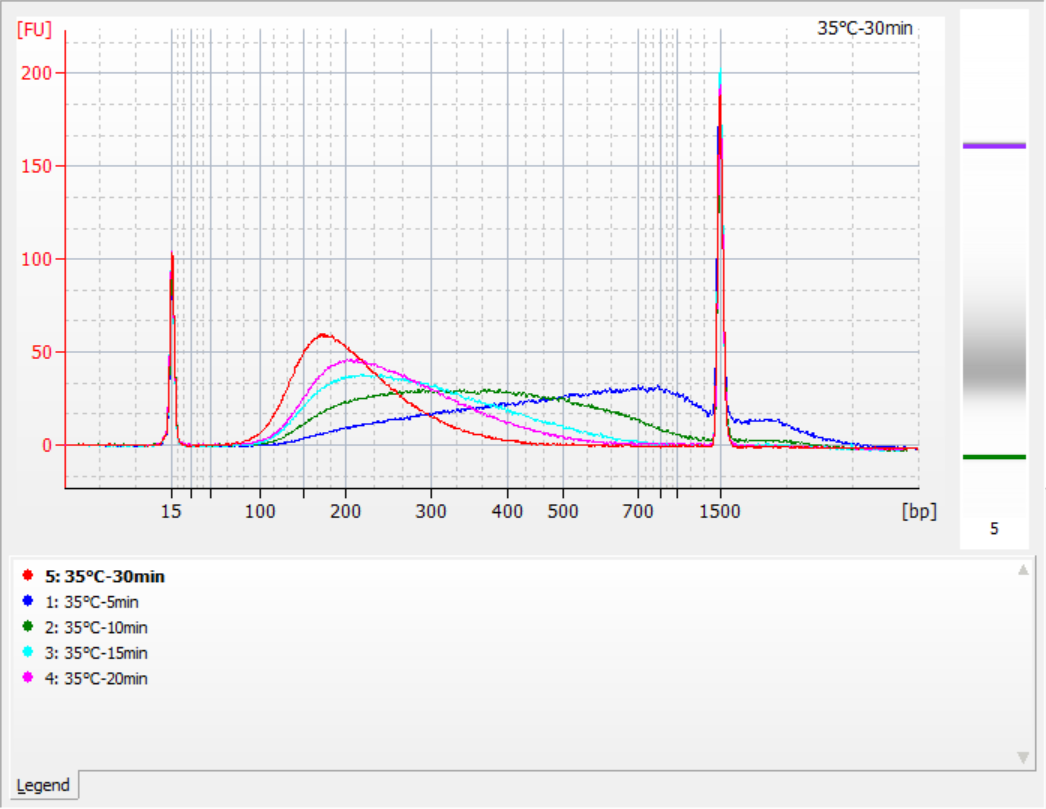 |
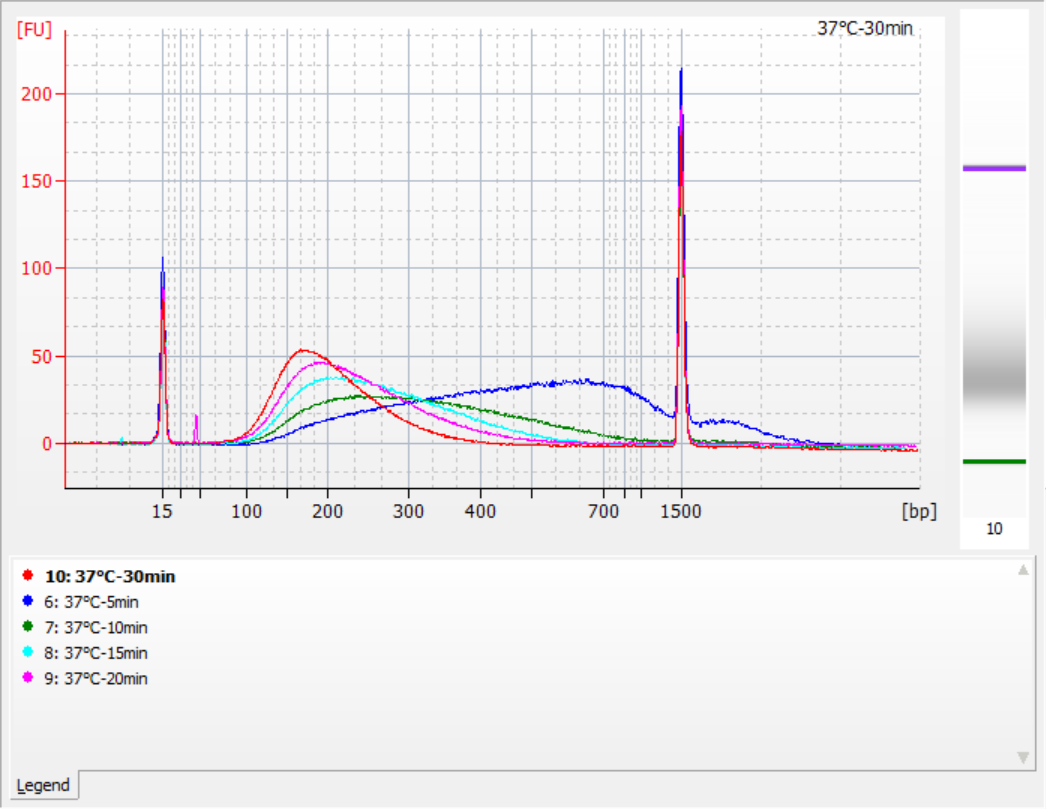 |
Paiement et sécurité
Vos informations de paiement sont traitées en toute sécurité. Nous ne stockons pas les détails de la carte de crédit ni accès aux informations de votre carte de crédit.
Enquête
Vous pouvez aussi aimer
FAQ
Le produit est destiné à des fins de recherche uniquement et n'est pas destiné à un usage thérapeutique ou diagnostique chez l'homme ou l'animal. Les produits et le contenu sont protégés par des brevets, des marques déposées et des droits d'auteur appartenant à Yeasen Biotechnology. Les symboles de marque indiquent le pays d'origine, pas nécessairement l'enregistrement dans toutes les régions.
Certaines applications peuvent nécessiter des droits de propriété intellectuelle tiers supplémentaires.
Yeasen se consacre à la science éthique, estimant que nos recherches doivent répondre à des questions cruciales tout en garantissant la sécurité et les normes éthiques.