Description
Les billes de sélection d'ADN Hieff NGS™ sont préparées selon le principe SPRI (Solid Phase Reverse Immobilization) et sont applicables à la purification de l'ADN et à la sélection de la taille lors de la préparation des bibliothèques de séquençage de nouvelle génération (NGS). Les billes de sélection d'ADN Hieff NGS™ sont compatibles avec divers kits de préparation de bibliothèques d'ADN et d'ARN et constituent une bonne alternative Billes AMPure.
Composants
| Composants N° | Nom | 12601ES08 | 12601ES56 | 12601ES75 |
| 12601 | Billes de sélection d'ADN Hieff NGS™ | 5 ml | 60 ml | 450 ml |
Caractéristiques
| Gamme de produits | Billes de nettoyage et de sélection d'ADN |
| Matériel de départ | ADN |
| Compatibilité | ADN |
| Technologie d'isolation | Bille magnétique |
| Type de produit final | ADN |
| À utiliser avec (application) | Nettoyage de l'ADN, sélection de la taille de l'ADN |
Expédition et stockage
Les perles sont expédiées avec des packs de glace et peuvent être conservées entre 2°C et 8°C pendant un an.
Instructions
- 1. Préparation
Équilibrer les billes de sélection à température ambiante pendant au moins 30 min avant utilisation.
- 2. Sélection de la taille de l'ADN
Le flux d’opération de sélection de taille est illustré dans la Figure 1 et le protocole est le suivant.
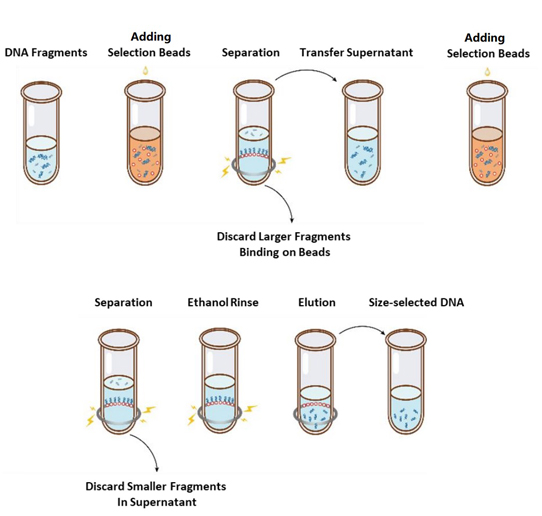
Figure 1. Organigramme de la sélection de la taille de l'ADN
2.1 Mélangez soigneusement les billes en les vortexant ou en les pipetant de haut en bas à chaque fois avant utilisation.
2.2 Ajoutez la première série de billes de sélection à l'échantillon (voir le tableau 1). Mélangez soigneusement en vortexant ou en pipetant de haut en bas au moins 10 fois.
2.3 Incuber à température ambiante pendant 5 min.
2.4 Centrifuger brièvement le tube et le placer sur un support magnétique. Lorsque la solution est claire (environ 5 min), transférer le surnageant dans un nouveau tube PCR.
2.5 Ajoutez la deuxième série de billes de sélection à l'échantillon de l'étape 2.4 conformément au tableau 1. Mélangez soigneusement en vortexant ou en pipetant de haut en bas au moins 10 fois.
2.6 Incuber à température ambiante pendant 5 min.
2.7 Centrifuger brièvement le tube et le placer sur un support magnétique. Lorsque la solution est claire (environ 5 min), aspirer le surnageant et le jeter.
2.8 Conservez le tube dans le support magnétique et ajoutez 200 μL d'éthanol à 80 % fraîchement préparé sans perturber les billes, puis laissez incuber à température ambiante pendant 30 secondes. Aspirez l'éthanol et jetez-le.
2.9 Répétez l’étape 2.8 une fois pour un total de deux lavages.
2.10 Éliminez l'éthanol résiduel avec des pointes de pipette de 10 µL. Gardez le tube dans le support magnétique, séchez les billes de sélection à l'air avec le couvercle ouvert jusqu'à ce que des fissures apparaissent (environ 5 min).
Remarque : ne séchez pas trop les billes de sélection. Cela peut entraîner une récupération plus faible de la cible ADN.
2.11 Retirez le tube du support magnétique. Ajoutez une quantité appropriée de ddH2O (≥ 20 µL) et mélangez soigneusement en vortexant ou en pipetant de haut en bas au moins 10 fois.
2.12 Incuber à température ambiante pendant 5 min.
Centrifuger brièvement le tube et le placer sur le support magnétique. Lorsque la solution est claire (environ 5 minutes), transférer 20 μL du surnageant dans un nouveau tube.
- 3. Conditions recommandées pour la sélection de la taille de l'ADN
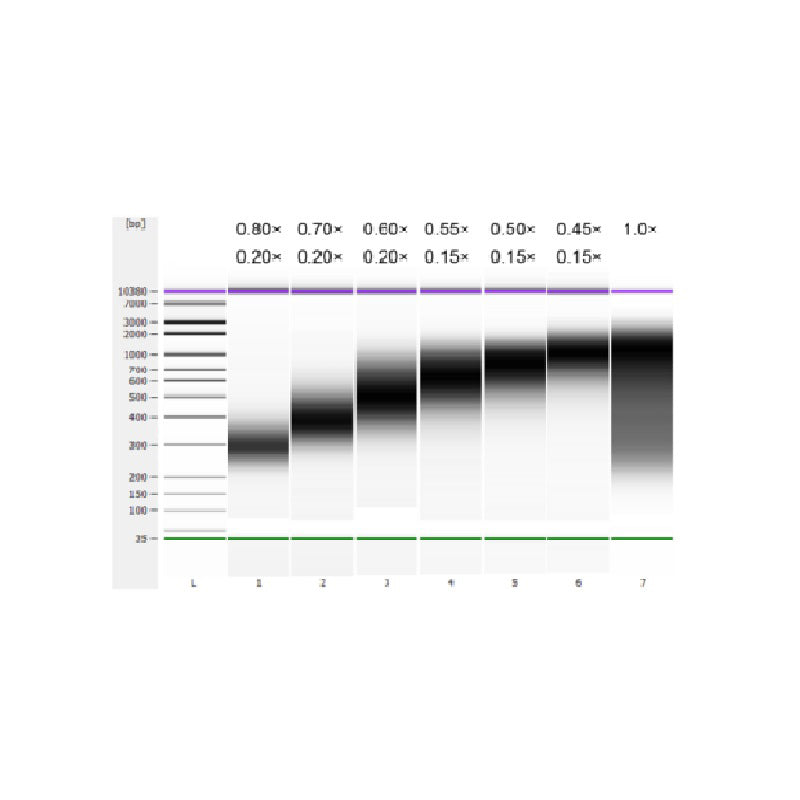
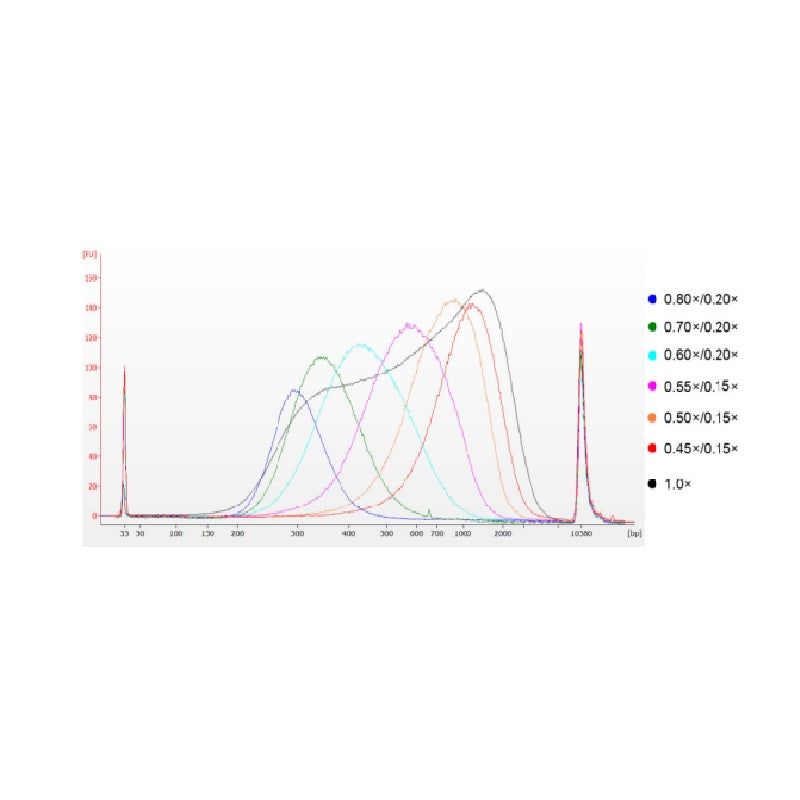
L'ADN du thymus de veau a été fragmenté par sonication pour préparer un fragment de 100 à 1 000 pb, et deux cycles de sélection de taille ont été effectués conformément au tableau 1. Les résultats ont été analysés à l'aide du bioanalyseur Agilent 2100 (figure 2).
Tableau 1. Conditions recommandées pour la sélection de la taille de l'ADN
| Longueur du fragment d'ADN | 250-350 pb | 320-420 pb | 450-550 pb | 550-700 pb | 700-900 pb | 800-1 000 pb |
| Proportion de billes : ADN pour le 1er tour | 0,80× | 0,70× | 0,60× | 0,55× | 0,50× | 0,45× |
| Proportion de billes : ADN pour le 2ème Rond | 0,20× | 0,20× | 0,20× | 0,15× | 0,15× | 0,15× |
Remarque : « × » dans le tableau indique le volume de l'échantillon d'ADN. Par exemple, si la longueur d'insertion de la bibliothèque est de 250 pb et que le volume de l'échantillon d'ADN est de 100 μL, le volume de billes magnétiques utilisées lors du premier cycle de tri est de 0,80 × 100 μL = 80 μL ; le volume de billes magnétiques utilisées lors du deuxième cycle de tri est de 0,20 × 100 μL = 20 μL.
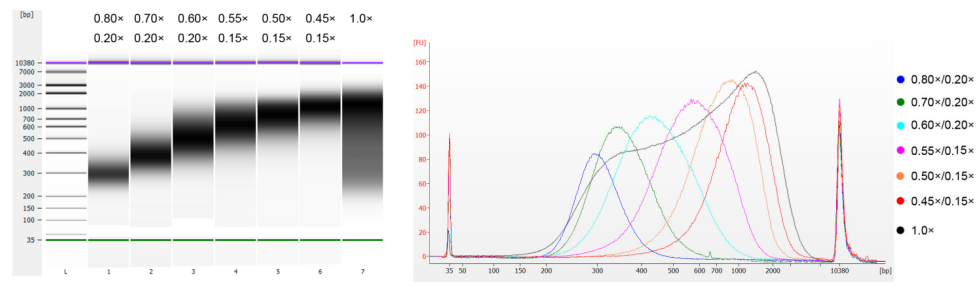
Figure 2. Électrophorégramme de la puce à ADN haute sensibilité Agilent 2100
Remarques :
1. Pour votre sécurité et votre santé, veuillez porter des blouses de laboratoire et des gants jetables pour l'opération.

Cité à partir de « Intégration spécifique de séquence par la casposase de la famille 1 de Candidatus Nitrosopumilus koreensis AR1. Nucleic Acids Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725"

Cité dans « Une infection récente par Wolbachia modifie les communautés microbiennes dans les populations sauvages de Laodelphax striatellus. Microbiome. 2020 ; 8(1) : 104. Publié le 2 juillet 2020. doi : 10.1186/s40168-020-00878-x »
[1] Wang X, Yuan Q, Zhang W, et al. Intégration spécifique de séquence par la casposase de la famille 1 de Candidatus Nitrosopumilus koreensis AR1. Acides Nucléiques Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725(IF:16.971)
[2] Duan XZ, Sun JT, Wang LT, et al. Une infection récente par Wolbachia modifie les communautés microbiennes dans les populations sauvages de Laodelphax striatellus. Microbiome. 2020 ; 8(1) : 104. Publié le 2 juillet 2020. doi : 10.1186/s40168-020-00878-x(IF : 11.607)
[3] Chanson B, Almatrafi E, Sang F et al.Gestion des sédiments traités au Fenton avec du biochar et du compost de fumier de mouton : effets sur les caractéristiques évolutives de la communauté bactérienne. J Environ Manage. 2022;316:115218. doi:10.1016/j.jenvman.2022.115218(IF:6.789)
[4] Huang C, Mei Q, Lou L, et al. Colite ulcéreuse en réponse à la transplantation de microbiote fécal via la modulation du microbiote intestinal et de l'équilibre des cellules Th17/Treg. Cells. 2022 ; 11(11) : 1851. Publié le 5 juin 2022. doi : 10.3390/cells11111851(IF : 6.600)
[5] Ghosh S, Yang X, Wang L, Zhang C, Zhao L. L'alimentation prébiotique en phase active modifie le microbiote intestinal, induit une atténuation indépendante du poids de la stéatose hépatique et du cholestérol sérique chez les souris nourries avec un régime riche en graisses. Comput Struct Biotechnol J. 2020 ; 19 : 448-458. Publié le 24 décembre 2020. doi : 10.1016/j.csbj.2020.12.011(IF : 6.018)
[6] Gao X, Yu B, Yu J, et al. Profilage développemental de la digestion des glucides alimentaires chez les porcelets. Front Microbiol. 2022 ; 13 : 896660. Publié le 29 avril 2022. doi : 10.3389/fmicb.2022.896660(IF : 5.640)
[7] Li P, Zhang Y, Yan F, Zhou X. Caractéristiques d'un bactériophage, vB_Kox_ZX8, isolé de Klebsiella oxytoca clinique et son effet thérapeutique sur la bactériémie chez la souris. Front Microbiol. 2021 ; 12 : 763136. Publié le 3 décembre 2021. doi : 10.3389/fmicb.2021.763136(IF : 5.640)
[8] Lin Z, Luo P, Huang D, Wu Y, Li F, Liu H. Stratégie multi-omique pour l'analyse de la toxicité de l'acrylamide dans le modèle Saccharomyces cerevisiae. Chem Biol Interact. 2021;349:109682. doi:10.1016/j.cbi.2021.109682(IF:5.194)
[9] Sun X, Lv W, Wang Y et al. Le gène Mrgprb2 joue un rôle dans les réactions anaphylactoïdes induites par l'injection de Houttuynia cordata. J Ethnopharmacol. 2022;289:115053. est ce que je:10.1016/j.jep.2022.115053(IF:4.360)
[10] Ma H, Lai B, Zan C, Di X, Zhu X, Wang K. GLO1 contribue à la résistance aux médicaments d'Escherichia coli en induisant le type PER de β-lactamases à spectre étendu. Infect Drug Resist. 2022 ; 15 : 1573-1586. Publié le 5 avril 2022. doi : 10.2147/IDR.S358578(IF : 4.003)
[11] Zhong Y, Zhao W, Tang Z, et al. Analyse transcriptomique comparative des différents stades de développement de l'ovaire chez l'écrevisse rouge des marais Procambarus clarkii. BMC Genomics. 2021;22(1):199. Publié le 21 mars 2021. doi:10.1186/s12864-021-07537-x(IF:3.969)
[12] Lian C, Yang H, Lan J, et al. Une analyse comparative des génomes chloroplastiques révèle des relations phylogénétiques et une variation intraspécifique chez la plante médicinale Isodon rubescens. PLoS One. 2022 ; 17(4) : e0266546. Publié le 6 avril 2022. doi : 10.1371/journal.pone.0266546(IF : 3.240)
[13] Diao G, Huang J, Zheng X et al. Prostaglandine L'E2 joue un double rôle dans la régulation de la migration des cellules dendritiques. Int J Mol Med. 2021;47(1):207-218. doi:10.3892/ijmm.2020.4801(IF:3.098)
[14] Bing XL, Zhao DS, Peng CW, Huang HJ, Hong XY. Similitudes et variations spatiales des communautés bactériennes et fongiques dans les populations de cicadelles du riz (Hemiptera : Delphacidae). Insect Sci. 2020;27(5):947-963. doi:10.1111/1744-7917.12782(IF:2.791)
[15] Li X, Zhou S, Zhang J, Zhou Z, Xiong Q. Changements directionnels dans la communauté bactérienne intestinale chez les larves de mouche soldat noire (Hermetia illucens). Animaux (Bâle). 2021 ; 11(12) : 3475. Publié le 6 décembre 2021. doi : 10.3390/ani11123475(IF : 2.752)
[16] Yang J, Peng Y, Kong W. Identification et séquence complète du génome du virus cryptique du mûrier 1. Arch Virol. 2022;167(2):687-690. doi:10.1007/s00705-021-05350-1(IF:2.574)
[17] Chang Y, Xia X, Sui L, et al. La colonisation endophyte des champignons entomopathogènes augmente la résistance des plantes aux maladies en modifiant la communauté bactérienne endophyte. J Basic Microbiol. 2021;61(12):1098-1112. doi:10.1002/jobm.202100494(SI:2.281)
[18] Ding CY, Ma YM, Li B, et al. Identification et analyse fonctionnelle des gènes différentiellement exprimés chez Myzus persicae (Hemiptera : Aphididae) en réponse au trans-anéthole. J Insect Sci. 2022;22(1):3. doi:10.1093/jisesa/ieab094(IF:1.857)
Paiement et sécurité
Vos informations de paiement sont traitées en toute sécurité. Nous ne stockons pas les détails de la carte de crédit ni accès aux informations de votre carte de crédit.
Enquête
Vous pouvez aussi aimer
FAQ
Le produit est destiné à des fins de recherche uniquement et n'est pas destiné à un usage thérapeutique ou diagnostique chez l'homme ou l'animal. Les produits et le contenu sont protégés par des brevets, des marques déposées et des droits d'auteur appartenant à Yeasen Biotechnology. Les symboles de marque indiquent le pays d'origine, pas nécessairement l'enregistrement dans toutes les régions.
Certaines applications peuvent nécessiter des droits de propriété intellectuelle tiers supplémentaires.
Yeasen se consacre à la science éthique, estimant que nos recherches doivent répondre à des questions cruciales tout en garantissant la sécurité et les normes éthiques.