Description
CHO Le kit de détection des résidus d'ADN des cellules hôtes est utilisé pour l'analyse quantitative de CHO résidus d'ADN de cellules hôtes dans des échantillons intermédiaires, des produits semi-finis et finis de divers produits biologiques.
Ce kit adopte la sonde fluorescente Taqman et la méthode de réaction en chaîne par polymérase (PCR), qui a une limite de détection minimale de niveau fg et peut détecter spécifiquement et rapidement le CHO résiduel ADN cellulaire. Le kit doit être utilisé avec le kit de préparation d'échantillons d'ADN résiduel (réf. 18461ES).
Produit Composants
| Non. | Nom | 41332ES50 (50T) | 41332ES60 (100T) |
| 41332-A | CHO PCR quantitative Mélanger | 0,75 mL | 1,5 mL |
| 41332-B | CHO Amorce et sonde Mélanger | 250 μL | 500 μL |
| 41332-C | Dilution de l'ADN Tampon | 2×1,8 mL | 4×1,8 mL |
| 41332-D | CHO Contrôle de l'ADN (30 ng/μL) | 25 μL | 50 μL |
Expédition et stockage
1. Expédié sur glace sèche et stocké à -20°C pour 2 année
Ce produit doit être conservé entre -25 et -15 ℃ pendant 2 ans.
Les produits 41332-A et 41332-B doivent être conservés à l'abri de la lumière.
Modèles d'instruments applicables
Inclure, mais sans s'y limiter :
Bio-Rad : Module optique CFX96.
Thermo Scientific : ABI 7500 ; ABI Quant Studio 5.
Instructions
- CHO Dilution standard d'ADN et préparation de la courbe standard
Le CHO Le contrôle de l'ADN a été dilué en gradient à l'aide du tampon de dilution de l'ADN fourni dans le kit*, et la dilution
la concentration est de 3 ng/μL, 300 pg/μL, 30 pg/μL, 3 pg/μL, 300 fg/μL, 30 fg/μL, 3 fg/μL.
Voir les instructions détaillées ci-dessous :
- Décongelez le contrôle ADN CHO et le tampon de dilution ADN sur de la glace. Après décongélation complète, vortexez doucement pour mélanger et centrifugez à basse vitesse pendant 10 secondes.
- Retirez sept tubes propres de 1,5 ml, marqués Std0, Std1, Std2, Std3, Std4, Std5, Std6.
- Ajoutez 90 μL de tampon de dilution d'ADN et 10 μL de contrôle CHODNA au tube à centrifuger de 1,5 mL étiqueté Std0, à savoir diluer à 3 ng/µL. Mélanger puis centrifuger pendant 10 secondes. Sous-emballer l'étalon d'ADN dilué et le conserver à court terme (pas plus de 3 mois) à -25~-15℃**Veuillez éviter les cycles de gel-dégel répétés.
- Ajouter 90 μL de tampon de dilution d'ADN dans d'autres tubes***, puis suivez la procédure ci-dessous pour les dilutions en série****.
| Tube | Taux de dilution | Concentration standard |
| Std1 | 10 μL de Std0 + 90 μL de tampon de dilution d'ADN | 300 pg/μL |
| Std2 | 10 μL de Std1 + 90 μL de tampon de dilution d'ADN | 30 pg/μL |
| Std3 | 10 μL de Std2 + 90 μL de tampon de dilution d'ADN | 3 pg/μL |
| Std4 | 10 μL de Std3 + 90 μL de tampon de dilution d'ADN | 300 fg/μL |
| Norme 5 | 10 μL de Std4 + 90 μL de tampon de dilution d'ADN | 30 fg/μL |
| Norme 6 | 10 μL Std5 + 90 μL de tampon de dilution d'ADN | 3 fg/μL |
Tableau 1 Dilution du gradient standard
*Trois puits répliqués sont nécessaires pour chaque concentration. La plage de détection est de 3 fg/μL~300pg/μL et cette plage peut être étendue.
**Afin de réduire le nombre de cycles de congélation-décongélation répétés et d'éviter la contamination, il est recommandé de stocker le contrôle ADN en aliquotes à -25~-15℃ pour la première fois.
***Une fois décongelé, le tampon de dilution d'ADN peut être conservé à 2-8°C pendant 7 jours. S'il n'est pas utilisé pendant une longue période, veuillez le conserver à -25~-15℃.
****Assurez-vous que le modèle est complètement mélangé, secouez doucement le mélange pendant 15 secondes à 1 minute pour chaque dilution de gradient.
- Préparation du contrôle de récupération par extraction (ERC)
Réglez la concentration d'ADN CHO dans l'ERC selon les besoins (l'échantillon ERC a été préparé avec 30 pg d'ADN CHO à titre d'exemple), comme suit :
- Ajoutez 100 μL d'échantillon de test dans un tube propre de 1,5 mL, puis ajoutez 10 μL d'étalon ADN CHO 3pg/μL (Std3) et mélangez bien, marqué ERC.
- Effectuer l’extraction d’ADN de l’échantillon ERC avec les échantillons de test pour préparer l’échantillon ERC purifié.
- Préparation de la solution de contrôle négatif (NCS)
Définissez le contrôle négatif dans l'expérience, les étapes de fonctionnement spécifiques sont les suivantes :
1) Ajoutez 100 μL de matrice d'échantillon (ou de tampon de dilution d'ADN) dans un tube propre de 1,5 mL, puis marqué comme NCS.
2) Effectuez l’extraction d’ADN de l’échantillon NCS avec les échantillons de test pour préparer l’échantillon NCS purifié.
- Préparation sans contrôle de modèle (NTC)
Définissez le contrôle sans modèle dans l'expérience, les étapes de fonctionnement spécifiques sont les suivantes :
1) Le NTC ne nécessite aucun prétraitement d’échantillon et peut être configuré au stade de la détection qPCR de l’ADN résiduel.
2) L'échantillon NTC dans chaque tube ou puits est de 20 μL de mélange (c'est-à-dire 15 μL de mélange CHO qPCR + 4 μL de mélange CHO Primer&probe + 1 μL IC) + 10 μL de tampon de dilution d'ADN. Il est recommandé de configurer trois puits répliqués.
- Système de réaction PCR
| Composant | Volume (μL) |
| CHO Mélange qPCR* | 15 |
| CHO Mélange d'amorces et de sondes | 5 |
| Modèle d'ADN | 10 |
| Volume total** | 30 |
Tableau 2 Système réactionnel
*Calculer le volume total de la réaction PCR en fonction du nombre de réactions : qPCR Mix = (nombre de réactions + 2) × (15 + 4 + 1) μL (y compris les pertes de deux puits de réaction). Il est recommandé d'effectuer plus de trois réplicats pour chaque échantillon dans l'expérience.
**Après avoir bouché le tube ou scellé la plaque, centrifugez le tube ou la plaque de réaction à basse vitesse pendant 10 secondes. Après avoir suffisamment secoué et mélangé pendant 5 secondes, répétez la centrifugation pour recueillir le liquide du couvercle ou de la paroi vers le fond. Évitez les bulles pendant le fonctionnement.
Voir le tableau ci-dessous pour la configuration de plaque recommandée :
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| UN | CNT |
| TS 1 | TS 1 | TS 1 |
| Norme 1 | Norme 1 | Norme 1 |
|
|
|
| B | CNT |
| TS 2 | TS 2 | TS 2 |
| Norme 2 | Norme 2 | Norme 2 |
|
|
|
| C | CNT |
| TS 3 | TS 3 | TS 3 |
| Norme 3 | Norme 3 | Norme 3 |
|
|
|
| D |
|
|
|
|
|
| Norme 4 | Norme 4 | Norme 4 |
|
|
|
| E | NCS |
| ERC 1 | ERC 1 | ERC 1 |
| Norme 5 | Norme 5 | Norme 5 |
|
|
|
| F | NCS |
| ERC 2 | ERC 2 | ERC 2 |
| Norme 6 | Norme 6 | Norme 6 |
|
|
|
| G | NCS |
| ERC 3 | ERC 3 | ERC 3 |
|
|
|
|
|
|
|
| H |
|
|
|
|
|
|
|
|
|
|
|
|
Tableau 3 Ordinateur sur la carte de référence
La disposition des plaques comprend : 6 Std (la courbe standard de 6 concentrations standard), 1 NTC (contrôle sans modèle), 1 NCS (solution de contrôle négatif), 3 TS (échantillons de test), 3 ERC (contrôle de récupération d'extraction).Trois puits répliqués pour chaque échantillon.
- Installation directives pour un instrument PCR(méthode en 2 étapes) (par exemple, instrument Thermo ABI 7500 qPCR, logiciel version 2.0)
Les instructions suivantes s'appliquent uniquement à l'instrument Thermo ABI 7500 qPCR (version logicielle 2.0). Si vous utilisez un autre instrument, reportez-vous au guide de l'instrument concerné pour obtenir des instructions de configuration.
1) Générez une nouvelle expérience, choisissez le modèle de quantification absolue ou défini par l'utilisateur.
2) Créez 1 sonde de détection, nommée « CHO-DNA », sélectionnez le fluorophore rapporteur comme « FAM » et éteignez le fluorophore comme « Aucun » ; Créez 1 sonde de détection supplémentaire, nommez-la « IC » et sélectionnez le fluorophore rapporteur « CY5 » et le fluorophore d'extinction « Aucun ». La fluorescence de référence est « ROX » (la fluorescence de référence peut être basée sur le modèle de l'instrument, etc., sélectionnez si vous devez l'ajouter).
3) Dans le volet « Échantillons », ajoutez toutes les informations sur les échantillons à tour de rôle. Sélectionnez ensuite les puits, choisissez la cible et les échantillons en conséquence. Définissez la tâche de CHO Norme ADN comme norme et attribuer les valeurs 300000, 30000, 3000, 300, 30, 3 (l'unité de concentration d'ADN dans chaque puits est fg/μL) dans la colonne Quantité et nommez les puits Std 1, norme 2, norme 3, norme 4, norme 5, Std 6, en conséquence. Définissez la tâche de NTC comme NTC. Définissez le NCS, le TS et l'ERC comme Inconnu, et nommez-les en fonction de la disposition des plaques ci-dessus. Cliquez ensuite sur Suivant.
4) Définissez le programme d’amplification : définissez le volume de réaction à 30 μL.
| Étape du cycle | Température (℃) | Temps | Cycles |
| Dénaturation initiale | 95℃ | 5 minutes | 1 |
| Dénaturation | 95℃ | 15 secondes | 40 |
| Recuit/Extension (Collecte de fluorescence) | 60℃ | 30 secondes |
Tableau 4 Procédure d'amplification
- Analyse des résultats de qPCR
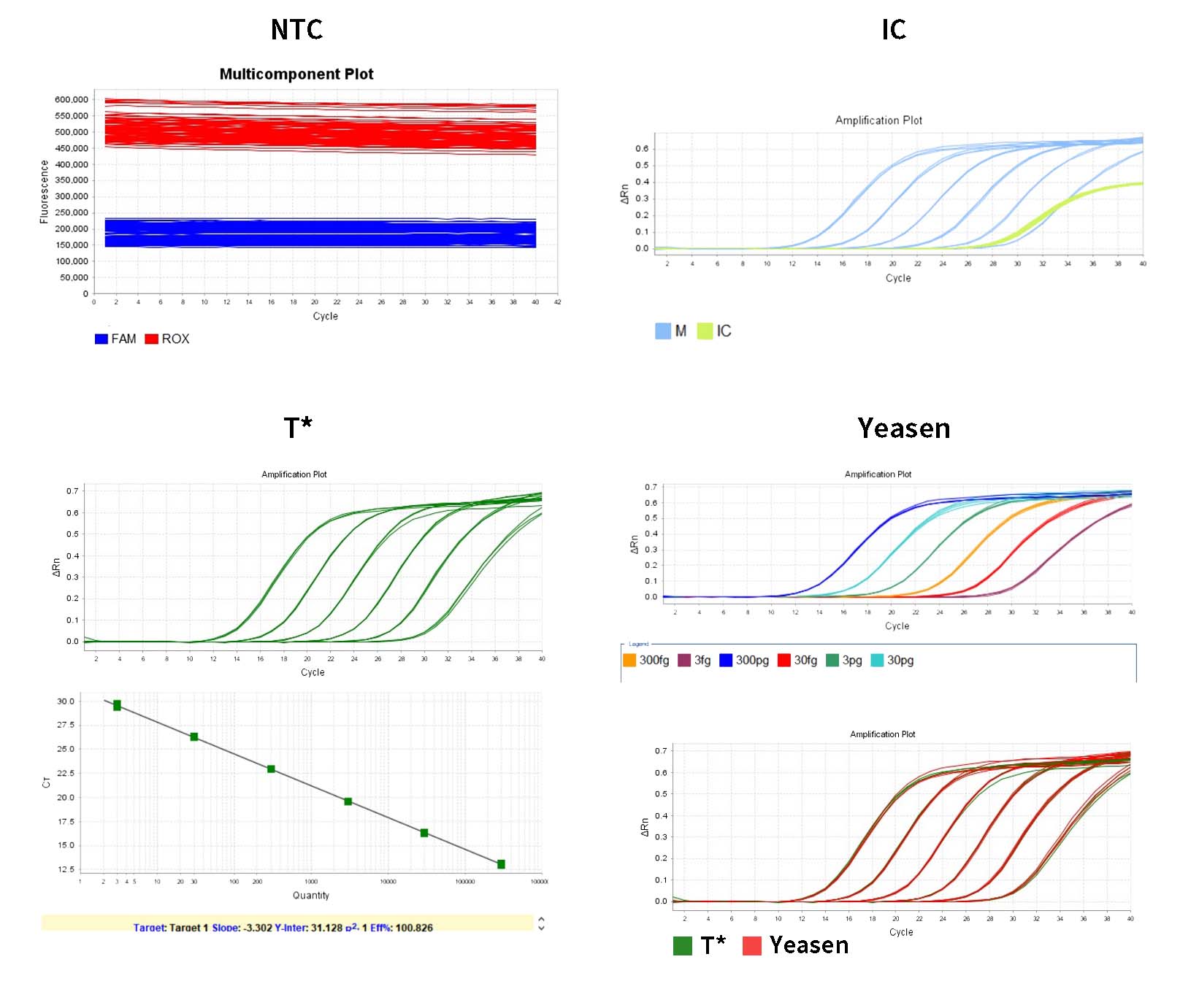
1) Le système indique automatiquement le seuil dans le panneau Amplification Plot d'Analyse. Le seuil donné par le système est parfois trop proche de la ligne de base, ce qui entraîne une grande différence de Ct entre les puits répliqués. Vous pouvez ajuster manuellement le seuil à une position appropriée et cliquer sur Analyser. Vous pouvez ensuite vérifier dans un premier temps si la courbe d'amplification est normale dans le graphique multicomposant.
2) Dans l'onglet Analyse des résultats, examinez le tracé de la courbe standard. Vérifiez les valeurs de R2, Efficacité, Pente et l'ordonnée à l'origine. Pour une courbe standard normale, R²>0,99, 90 %≤Eff%≤110 %, -3,6≤Pente≤-3,1.
3) Dans le volet « Afficher le tableau des puits » dans Analyse, les concentrations de chaque échantillon sont affichées en Quantité, l'unité est fg/μL, les unités peuvent être converties dans le rapport d'analyse.
4) Les réglages des paramètres de l'analyse des résultats doivent être basés sur le modèle spécifique et la version du logiciel utilisé et peuvent généralement être interprétés automatiquement par l'instrument.
5) Calculez le taux de récupération des pointes en fonction des résultats du test de l'échantillon TS à mesurer et de l'ERC de récupération des pointes de l'échantillon. Le taux de récupération des pointes doit être compris entre 50 % et 150 %. Formule du compteur de taux de récupération des pointes : Récupération (%) = {Sample spiked assay (eg.pg/μL) - Sample assay (eg.pg/μL)} x Volume d'élution (μL) / Valeur théorique de la quantité d'ADN ajoutée (par exemple pg) x 100 %.
6) La valeur Ct du contrôle négatif NCS doit être supérieure à la moyenne de la concentration la plus faible Ct de la norme.
- Le contrôle sans modèle NTC doit être indéterminé ou la valeur Ct ≥ 3
Remarques
- Ce produit est destiné à la recherche uniquement.
- Veuillez travailler avec des blouses de laboratoire et des gants jetables, pour votre sécurité.
3. Veuillez lire attentivement ce manuel avant d'utiliser ce réactif. L'expérience doit être standardisée, y compris la manipulation des échantillons, la préparation du système de réaction et l'ajout d'échantillons.
4. Assurez-vous que chaque composant est entièrement vortexé et centrifugé à basse vitesse avant utilisation.
Paiement et sécurité
Vos informations de paiement sont traitées en toute sécurité. Nous ne stockons pas les détails de la carte de crédit ni accès aux informations de votre carte de crédit.
Enquête
Vous pouvez aussi aimer
FAQ
Le produit est destiné à des fins de recherche uniquement et n'est pas destiné à un usage thérapeutique ou diagnostique chez l'homme ou l'animal. Les produits et le contenu sont protégés par des brevets, des marques déposées et des droits d'auteur appartenant à Yeasen Biotechnology. Les symboles de marque indiquent le pays d'origine, pas nécessairement l'enregistrement dans toutes les régions.
Certaines applications peuvent nécessiter des droits de propriété intellectuelle tiers supplémentaires.
Yeasen se consacre à la science éthique, estimant que nos recherches doivent répondre à des questions cruciales tout en garantissant la sécurité et les normes éthiques.