Hai imparato come creare una libreria PCR-Free?
Nel sequenziamento ad alto rendimento, la costruzione di librerie convenzionali richiede l'amplificazione PCR. Da un lato, serve ad amplificare campioni di DNA traccia e aumenta la resa della libreria. Dall'altro, può amplificare i segnali fluorescenti, rendendo più facile per i sequenziatori catturare e identificare i segnali fluorescenti per migliorare l'accuratezza del sequenziamento. Tuttavia, la PCR è come una "spada a doppio taglio". Mentre risolve il problema del basso volume iniziale del campione e amplifica il segnale fluorescente, introduce anche errori di amplificazione e distorsioni e non può presentare perfettamente la sequenza del genoma "True face". Inoltre, la polimerasi PCR ha una certa distorsione di amplificazione. Alcune regioni, in particolare GC alta o GC bassa, struttura secondaria e altre regioni, hanno una bassa efficienza di amplificazione e sono difficili da coprire. Introdurrà anche un gran numero di InDel errati e porterà un gran numero di duplicazioni, causando uno spreco di volume di dati del DNA e aumentando il costo del sequenziamento. Pertanto, l'introduzione della tecnologia PCR-Free non solo ha i vantaggi della PCR, ma supera anche le carenze della PCR. Non solo può portare librerie di alta qualità con elevata sensibilità e realizzare il rilevamento di campioni di traccia, ma ha anche un'elevata accuratezza e riduce il carico di lavoro di convalida del follow-up per gli studi sulle varianti. Quindi cos'è esattamente la libreria PCR-Free e quali sono i suoi vantaggi?
1. Che cos'è la libreria PCR-Free?
2. Quali sono i vantaggi della tecnologia PCR-Free?
3. Visualizzazione dei dati PCR-Free
4. Domande frequenti
5. Informazioni sul prodotto
6. Per quanto riguarda la lettura
1. Che cos'è la libreria PCR-Free?
La tecnologia di sequenziamento di nuova generazione è la tecnologia più comunemente utilizzata nella ricerca di sequenziamento ad alto rendimento nella moderna biologia molecolare. Il tradizionale sequenziamento di prima generazione non è più in grado di soddisfare pienamente le esigenze dei ricercatori. Il risequenziamento del genoma di organismi modello e il sequenziamento del genoma di organismi non modello richiedono costi maggiori. Tecnologia di sequenziamento bassa, a rendimento più elevato e più veloce, dando così vita alla tecnologia di sequenziamento di seconda generazione. Il principio fondamentale della tecnologia di sequenziamento di seconda generazione è il sequenziamento per sintesi, ovvero determinare la sequenza del DNA catturando le informazioni di base etichettate all'estremità appena sintetizzate. Il principio principale della tecnologia di sequenziamento di seconda generazione per il sequenziamento del DNA è prima frammentare il DNA, riparare l'estremità del DNA frammentato e quindi aggiungere adattatori specifici su entrambi i lati e quindi utilizzare metodi diversi per generare milioni di PCR fissate spazialmente. Per gli array clonali, i dati di sequenziamento vengono ottenuti utilizzando l'ibridazione dei primer e le reazioni di estensione enzimatica per creare immagini delle etichette fluorescenti incorporate da ciascuna reazione di estensione.
Il sequenziamento del DNA tramite sequenziamento di nuova generazione include principalmente due processi: preparazione della libreria e sequenziamento su macchina. Durante la preparazione della libreria, i frammenti genomici interrotti casualmente vengono in genere amplificati tramite PCR standard. Tuttavia, per alcuni modelli speciali, ci sono fattori come la complessa struttura secondaria o la scarsa stabilità termica, che influenzano la preferenza di amplificazione della PCR modello, quindi non tutte le sequenze genomiche possono essere riflesse in modo equo nella libreria di amplificazione PCR. Soprattutto per alcuni modelli con elevato contenuto di GC o AT, a volte è difficile utilizzare il metodo PCR per amplificare per la costruzione della libreria. Non vi è alcuna differenza tra la libreria senza PCR e la libreria PCR convenzionale quando si esegue il sequenziamento sulla macchina, tranne per il fatto che la PCR non viene eseguita durante il processo di costruzione della libreria. La libreria senza PCR può teoricamente migliorare la distribuzione delle letture dei dati e generare una copertura del genoma più uniforme.La libreria PCR-free è un supplemento al metodo di costruzione della libreria. Poiché viene preparata direttamente in una libreria di bordo senza amplificazione PCR, può migliorare la copertura di alcune regioni ad alto GC o ad alto AT, riducendo così le basi di errore introdotte da PCR, bias dei dati e ripetizione della sequenza.
I passaggi principali della costruzione di librerie NGS convenzionali sono la frammentazione del genoma, la riparazione delle estremità, l'aggiunta dell'adattatore, la PCR e l'amplificazione del segnale prima del sequenziamento. Quindi, che dire della costruzione di librerie senza PCR? Come suggerisce il nome, è un processo di costruzione di librerie che non richiede PCR. I passaggi principali della costruzione di librerie sono la frammentazione del genoma, la modifica delle estremità, l'aggiunta del linker e il controllo di qualità della libreria. Ma in realtà, questo può essere considerato solo come senza PCR nel processo di costruzione di librerie. Il vero senza PCR dovrebbe essere un processo dalla costruzione della libreria al sequenziamento senza amplificazione della libreria (come la tecnologia di sequenziamento a singola molecola) o una tecnologia di sequenziamento senza accumulo di errori di amplificazione PCR (come DNBseq di MGI).

Figura 1. Diagramma di flusso della costruzione di una biblioteca convenzionale e della costruzione di una biblioteca senza PCR
2. Quali sono i vantaggi della tecnologia PCR-Free?
Nel processo di costruzione di una libreria di routine, l'enzima di amplificazione può introdurre errori. Attraverso l'amplificazione ciclica, questi errori saranno accumulati e amplificati, riducendo la fedeltà della replicazione della sequenza di DNA. Inoltre, la DNA polimerasi ha anche una certa distorsione di amplificazione, specialmente per le regioni con grandi differenze nel contenuto di GC o nelle strutture secondarie, con conseguente bassa efficienza di amplificazione e scarsa uniformità di copertura; mentre introduce InDel sbagliati, porterà anche un gran numero di duplicazioni che causano spreco di volume di dati di DNA e aumentano i costi di sequenziamento. La tecnologia PCR-Free può ben evitare i problemi sopra menzionati introdotti dall'amplificazione PCR nella costruzione di librerie convenzionali. Nel processo di costruzione e sequenziamento di librerie, se c'è un processo di amplificazione PCR, avrà un impatto maggiore sull'uniformità della copertura del genoma. Per i modelli di DNA, alcuni hanno strutture secondarie complesse e alcuni hanno grandi differenze nella stabilità termica. Questi fattori influenzeranno l'efficienza dell'amplificazione PCR. Pertanto, nel processo di amplificazione PCR, non si può garantire che tutti i frammenti genomici possano ottenere la stessa efficienza di amplificazione, ma c'è un evidente bias di amplificazione, come una bassa copertura nelle regioni ad alto GC o basso GC del genoma. Tuttavia, non c'è PCR nell'intero processo di sequenziamento PCR-Free. Rispetto alla costruzione della libreria PCR, la copertura delle regioni ad alto GC e delle regioni di ripetizione AT/TA del genoma è migliorata. I vantaggi specifici sono i seguenti.
2.1 Processo di preparazione della biblioteca semplificato, con risparmio di tempo
La preparazione della libreria senza PCR elimina la necessità di amplificazione PCR e di altri passaggi, semplificando il processo di preparazione della libreria e risparmiando tempo.
2.2 Riduzione dei costi di preparazione della biblioteca
Nel processo di preparazione della libreria convenzionale, vengono utilizzati costosi enzimi ad alta fedeltà per l'amplificazione PCR per garantire l'autenticità della sequenza. Allo stesso tempo, PCR-Free elimina la fase di amplificazione PCR, riducendo i costi di preparazione della libreria.
2.3 Ridurre la distorsione dell'amplificazione
Gli enzimi di amplificazione del DNA hanno determinati bias di amplificazione, specialmente per i modelli con differenze significative nel contenuto di GC o nella struttura secondaria. Pertanto, PCR-Free può evitare efficacemente il bias di amplificazione causato dalla polimerasi.
2.4 Nessuna duplicazione del PCR
PCR-Free può ridurre le letture duplicate e aumentare i tassi di mappatura univoca e l'utilizzo efficace dei dati.
2.5 Errore ridotto
L'amplificazione PCR ha maggiori probabilità di introdurre errori di replicazione di inserzione e delezione, con conseguente aumento del tasso di errore nel sequenziamento Indel, mentre la PCR-Free può evitare efficacemente la generazione e l'accumulo di tali errori.
3. Visualizzazione dei dati PCR-Free
Kit di preparazione della libreria di DNA libero PCR Hieff NGS™ Ultima Pro V2 (Cat# 12196ES) ha un tasso di conversione della libreria migliore rispetto ai prodotti della concorrenza.
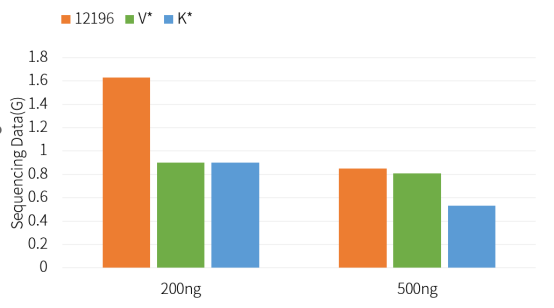
Figura 2. Quantità di dati di sequenziamento fuori macchina per la costruzione di librerie PCR-Free
4. Domande frequenti
D: Quali tipi di campioni sono adatti per le librerie senza PCR?
R: Per alcuni campioni con strutture secondarie complesse, elevata GC e preferenza per PCR, è possibile scegliere una libreria senza PCR da costruire.
D: Qual è la dimensione generale del frammento della libreria PCR-free?
R: La libreria PCR-free in sé non ha requisiti particolari per quanto riguarda la dimensione del frammento. La libreria del prodotto PCR, la libreria è costruita in base alla dimensione del prodotto PCR. Per la libreria genomica, si raccomanda di essere intorno a 350 bp, in modo che la qualità dei dati della libreria sia relativamente buona.
5. Informazioni sul prodotto
Il prodotto che Yeasen può fornire è illustrato nella Tabella 1.
Tabella 1. Informazioni sul prodotto
| Prodotto Nome | N. di cat. | Misurare |
| Kit di preparazione della libreria di DNA libero PCR Hieff NGS™ Ultima Pro V2 | 12196ES24/96 | 24/96T |
6. Per quanto riguarda la lettura
La soluzione per la preparazione della libreria NGS da campioni di DNA
Quanto ne sai sulla tecnologia NGS?
