Descrizione
Il kit di sintesi dell'RNA ad alto rendimento T7 ottimizza il sistema di reazione di trascrizione. Il kit può sintetizzare l'RNA a singolo filamento in modo efficiente utilizzando la RNA polimerasi T7, il DNA lineare a doppio filamento con la sequenza del promotore T7 come stampo, gli NTP come substrato per controllare la sequenza del DNA a valle del promotore. Durante la trascrizione, i nucleotidi modificati possono essere aggiunti al substrato per preparare l'RNA marcato con biotina o colorante.
Questo kit può sintetizzare trascrizioni lunghe e trascrizioni corte, l'RNA può essere prodotto a 100-200 μg con 1 μg di input di modello di DNA. L'RNA sintetizzato tramite trascrizione può essere utilizzato per varie applicazioni downstream, come la ricerca sulla struttura e la funzione dell'RNA, la protezione RNasi, l'ibridazione della sonda, l'RNAi, la microiniezione e in vitro traduzione.

Figura 1: In vitro Processo di trascrizione dell'RNA
Caratteristica
- Fino a 180 μg di RNA per reazione da 1 μg del modello di controllo
- Sistema di reazione ottimizzato per il processo IVT
- Diminuire la produzione di dsRNA
- Maggiore integrità e purezza dell'RNA
Applicazione
- In vitro Sintesi dell'RNA
Componenti
| Componenti n. | Nome | 10623ES50 (50 tonnellate) | 10623ES60 (100 tonnellate) | 10623ES70 (500 tonnellate) |
| 10623-A | Miscela di RNA polimerasi T7 | 100 microlitri | 200 microlitri | 1 ml |
| 10623-B | 10×tampone di trascrizione | 100 microlitri | 200 microlitri | 1 ml |
| 10623-C | ATP (100 mM) | 100 microlitri | 200 microlitri | 1 ml |
| 10623-D | CTP (100 mM) | 100 microlitri | 200 microlitri | 1 ml |
| 10623-E | GTP (100 mM) | 100 microlitri | 200 microlitri | 1 ml |
| 10623-F | Cavo UTP (100mM) | 100 microlitri | 200 microlitri | 1 ml |
| 10623-G | Modello di DNA di controllo (500ng/μL) | 10 microlitri | 20 microlitri | 100 microlitri |
Spedizione e stoccaggio
Trasporto di ghiaccio secco. Conservare a -15℃ ~ -25℃, valido per due anni.
Cifre
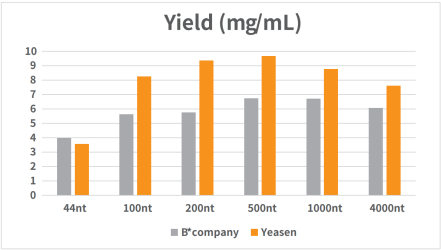
Figura 1. L'RNA standard è stato sintetizzato in vitro utilizzando il kit di sintesi dell'RNA T7.
La reazione è stata incubata in uno strumento PCR a 37℃ per 2 ore e poi purificata con sfere magnetiche (Cat#12602). Il risultato della resa è stato analizzato dallo spettrofotometro NanoDrop come mostrato nella Figura 1.
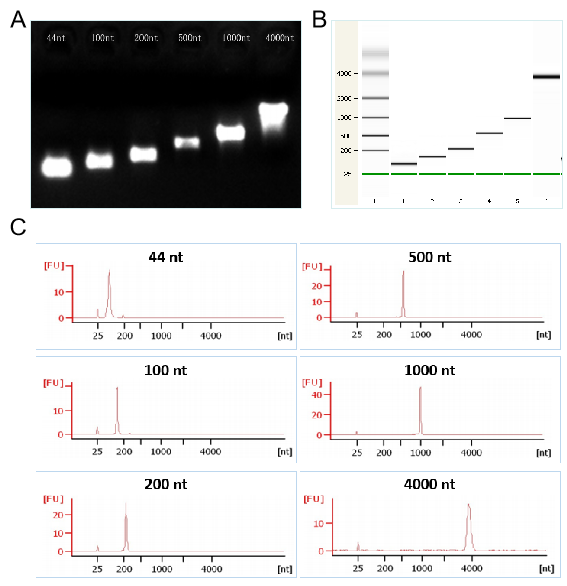
Figure 2. La dimostrazione della trascrizione di diverse lunghezze di RNA da parte del kit T7 rispettivamente nell'elettroforesi (Figure 2A), nel diagramma dell'elettroforesi capillare (Figure 2B) e nel cromatogramma (Figure 2C)
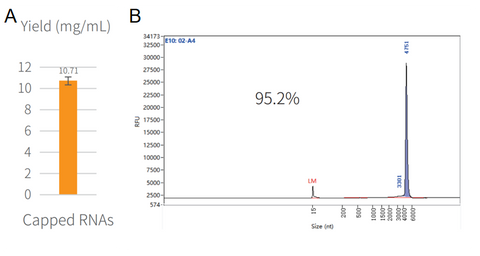
Figura 3. Sintesi dell'RNA cappato in vitro.
La reazione è stata incubata nello strumento PCR a 37℃ per 2 ore, quindi purificata con sfere magnetiche (Cat#12602). Il risultato della resa è stato analizzato dallo spettrofotometro NanoDrop come mostrato nella Figura 3A. Il risultato dell'integrità è stato analizzato tramite elettroforesi capillare come mostrato nella Figura 3B.
1. Bassa resa di trascrizione
La qualità del modello è strettamente correlata alla resa. Se la resa del gruppo sperimentale è significativamente inferiore a quella del gruppo di controllo, le possibili ragioni sono:
① il modello sperimentale contiene componenti inibitori;
② Il modello presenta qualche errore.
Suggerimenti:
① Ripulire il modello;
② Determinare la quantificazione del modello e la sua integrità;
③ Prolungare il tempo di reazione;
④ Aumentare la quantità di input del modello;
⑤ Provare altri promotori e RNA polimerasi.
2. Bassa resa delle trascrizioni brevi
Un breve frammento di inizio della trascrizione inibirà la reazione. Quando il prodotto di trascrizione è inferiore a 100 nt, estendere il tempo di reazione a 4-8 ore o aumentare la quantità di stampo a 2 μg aumenterà la resa di RNA.
3. La lunghezza della trascrizione dell'RNA è maggiore del previsto
Se l'elettroforesi mostra che la banda del prodotto è più grande della dimensione prevista, le possibili ragioni sono:
①Il modello del plasmide potrebbe non essere completamente linearizzato;
②L'estremità 3' del filamento sensoriale ha una struttura prominente;
③L'RNA ha una struttura secondaria non completamente denaturata.
Suggerimenti:
①Verificare se il modello è completamente linearizzato e, se necessario, eseguire un'ulteriore linearizzazione;
②Selezionare un enzima di restrizione adatto per evitare sporgenze 3' oppure utilizzare la DNA polimerasi Klenow Fragment/T4 per completare la trascrizione prima di procedere;
③Utilizzare gel denaturato per rilevare i prodotti dell'RNA.
4. La lunghezza della trascrizione dell'RNA è inferiore al previsto
Se l'elettroforesi mostra che la banda del prodotto è più piccola della dimensione prevista, le possibili ragioni sono:
①Il modello contiene una sequenza di terminazione simile alla RNA polimerasi T7;
②Il contenuto di GC nel modello è elevato.
Suggerimenti:
①Abbassare la temperatura di reazione (ad esempio, 30°C). A volte abbassare la temperatura può aumentare la lunghezza della trascrizione, ma ridurrà la resa. Oppure provare diverse RNA polimerasi per la trascrizione;
②Se il contenuto di GC del modello è elevato, utilizzare 42℃ per trascrivere, oppure aggiungere SSB per aumentare la resa e la lunghezza della trascrizione.
5. Elettroforesi di coda dei prodotti di trascrizione
Durante l'elettroforesi si verifica un fenomeno di scodamento.
Possibili motivi:
①Contaminato da RNasi durante l'operazione sperimentale;
②Modello di DNA contaminato da RNasi.
Suggerimenti:
①Utilizzare puntali per pipette e provette EP privi di RNasi, indossare guanti e maschere in lattice monouso e tutti i reagenti sono preparati con H2O priva di RNasi.
②Ripurificare il DNA stampo.
[1] Dong Z, Zheng N, Hu C, et al.Il microRNA-like RNA 8 (Nb-milR8) di Nosema bombycis aumenta la patogenicità fungina modulando l'espressione del gene BmPEX16 nel suo ospite, Bombyx mori. Microbiol Spectr. 2021;9(2):e0104821. doi:10.1128/Spectrum.01048-21(IF:7.171)
[2] Wang X, Tang S, Ye S, et al. Quantificazione ultrasensibile del miR-195-5p circolante con cascata di amplificazione dello spostamento a triplo filamento. Talanta. 2022;242:123300. doi:10.1016/j.talanta.2022.123300(IF:6.057)
Pagamento e sicurezza
Le informazioni di pagamento vengono elaborate in modo sicuro. Non archiviamo i dettagli della carta di credito né abbiamo accesso alle informazioni sulla tua carta di credito.
Indagine
Potrebbe piacerti anche
FAQ
Il prodotto è solo per scopi di ricerca e non è destinato all'uso terapeutico o diagnostico su esseri umani o animali. Prodotti e contenuti sono protetti da brevetti, marchi e copyright di proprietà di Yeasen Biotechnology. I simboli dei marchi indicano il paese di origine, non necessariamente la registrazione in tutte le regioni.
Alcune applicazioni potrebbero richiedere ulteriori diritti di proprietà intellettuale di terze parti.
Yeasen è un sostenitore della scienza etica, convinto che la nostra ricerca debba affrontare questioni critiche garantendo al contempo sicurezza e standard etici.

