Descrizione
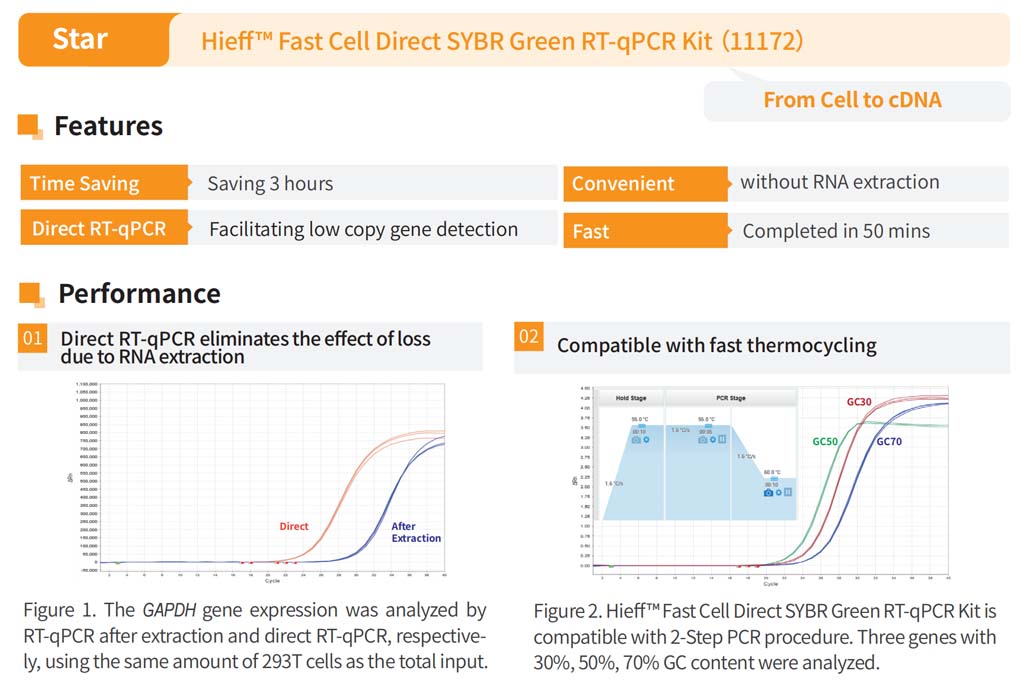
Capo™ Cellula veloce diretta Il kit RT-qPCR basato sul colorante SYBR Green è adatto per l'estrazione di RNA da tutti i tipi di cellule animali (come cellule della parete del lignaggio cellulare e cellule in sospensione, cellule di coltura primaria, varie cellule staminali, cellule iPS, ecc.), senza la necessità di estrarre RNA, e può essere utilizzato direttamente per l'analisi dell'espressione qPCR, che è breve, facile da usare e ha un basso tasso di errore. Ci vogliono solo 1,5 ore per completare i passaggi dalla preparazione del modello alla reazione di trascrizione inversa e all'analisi dell'espressione genica.
Il kit include reagenti per la trascrizione inversa e per il rilevamento della fluorescenza, che possono essere utilizzati per l'analisi dell'espressione genica senza dover acquistare reagenti aggiuntivi.
Specifiche
| N. di cat. | 11172ES40 / 11172ES60 |
| Misurare | 40 T/100 T |
Componenti
| Componenti n. | Nome | 11172ES40 | 11172ES60 |
| 11172-A | Tampone di lisi FCD | 2 ml. | 5 ml. |
| 11172-B | Tampone di lavaggio FCD | 8 ml | 20 ml. |
| 11172-C | Soluzione di arresto FCD | 100 microlitri | 250 microlitri |
| 11172-D | DNasi I | 80 microlitri | 200 microlitri |
| 11172-E | 4× Hifair™ FCD RT Mix | 200 microlitro | 500 microlitro |
| 11172-F | 2× Hieff™ FCD qPCR SYBR Master Mix | 2 ml | 5 ml |
| 11172-G | RNasi H libera2Lo | 2 ml | 5 ml |
Magazzinaggio
Il tampone di lisi FCD e il tampone di lavaggio FCD sono stati sciolti e conservati a 4°C per evitare contaminazioni. La soluzione di arresto FCD, la DNasi I, la miscela RT FCD 4× Hifair™, la miscela master qPCR SYBR 2× Hieff™ FCD devono essere conservate a una temperatura compresa tra -25 e -15℃.
Istruzioni
- Preparazione dei prodotti di scissione
1)1. Sciogliere il reagente a temperatura ambiente, capovolgerlo e mescolarlo delicatamente prima dell'uso, quindi utilizzarlo dopo una leggera centrifugazione per evitare la formazione di schiuma*.
*La mancata miscelazione dei reagenti, l'uso di un oscillatore per la miscelazione e la mancata configurazione dei reagenti sul ghiaccio possono portare a una riduzione delle prestazioni della reazione.
2) A seconda del tipo di cellula**, trasferire le cellule in una provetta da centrifuga e centrifugare a 5000 rpm per 2 min per raccogliere le cellule e aspirare bene il terreno di coltura. Se le cellule sono coltivate in piastre da 96 pozzetti, il terreno di coltura può essere aspirato direttamente.
3) Aggiungere 150 μL di tampone di lavaggio FCD in ciascun pozzetto, lavare le cellule soffiando, centrifugare a 5000 giri/min per 2 minuti e aspirare il tampone di lavaggio FCD***.
4) Aggiungere 48 μL di soluzione tampone di lisi FCD e 2 μL di soluzione DNasi Ⅰ a ciascun pozzetto, soffiare e mescolare a temperatura ambiente, quindi lasciare riposare per 5 minuti, quindi aggiungere 2.5 μL di soluzione di arresto FCD dopo l'incubazione****, quindi soffiare e mescolare per circa 5 volte per ottenere il prodotto di scissione*****.
** Il requisito di base per il numero di celle è 1 × 104 cellule per pozzetto e questo kit può essere utilizzato nell'intervallo di 1 × 103 - 1 × 106 cellule. Se il numero di cellule è maggiore, la quantità di soluzione tampone di lisi FCD e di soluzione di DNasiⅠ può essere aumentata proporzionalmente, come appropriato.
*** Le condizioni di centrifugazione variano da cella a cella, quindi centrifugare a una velocità adatta alle celle utilizzate.
**** Aggiungere 2,5 μL di soluzione bloccante FCD a 50 μL di lisato e aumentare la quantità di soluzione bloccante FCD secondo necessità.
***** Per la conservazione a lungo termine delle soluzioni dei prodotti di lisi cellulare, conservare a -20°C.
5) Sciogliere 4 x Hifair™ FCD RT Mix a temperatura ambiente e mescolare con leggera inversione, posizionare sul ghiaccio e configurare il sistema di reazione secondo la tabella seguente:
| Componenti | Volume (μL) | Concentrazione finale |
| 4× Hifair™ FCD RT Mix | 5 | 1× |
| prodotto di scollatura****** | X | X |
| RNasi H libera2Lo | Su A 20 | - |
*******Il livello di utilizzo consigliato è 2-5 μL, cercare di non superare il 45%.
- Trascrizione inversa
Pipettare e mescolare delicatamente la soluzione di reazione preparata sopra ed eseguire la reazione di trascrizione inversa secondo la procedura riportata nella tabella seguente:
| Temperatura | Tempo (minuto) |
| 55℃* | 15 minimo |
| 85℃ | 5 minimo |
* La temperatura di trascrizione inversa consigliata è di 55°C. Per modelli ad alto contenuto di GC o modelli complessi, la temperatura di trascrizione inversa può essere aumentata a 60°C. Il prodotto di trascrizione inversa può essere utilizzato direttamente per il rilevamento RT-qPCR a valle. Per evitare l'inibizione della reazione qPCR da parte del sistema di trascrizione inversa e per ottenere il valore Ct appropriato (10-35), il prodotto può essere diluito 10-1000 volte e quindi utilizzato. Se gli esperimenti a valle non vengono eseguiti per un breve periodo, può essere conservato a -20℃.
- PCR quantitativa a fluorescenza
1) Reazione sistema configurazione
Per la preparazione della soluzione di reazione (preparazione su ghiaccio) si raccomandano i seguenti rapporti.
| Componenti | Volume (μL) | Concentrazione finale |
| 2× Hieff™ FCD qPCR SYBR Master Mix | 10 | 1× |
| Primer diretto (10 μmol/L) | 0,4 | 0,2 μmol/L |
| Primer inverso (10 μmol/L) | 0,4 | 0.2 μmol/L |
| Prodotto di trascrizione inversa* | X | - |
| RNasi H libera2Lo | Su A 20 | - |
*Non aggiungere più di 1/10 del volume RT-qPCR del prodotto di trascrizione inversa. Un'elevata concentrazione di stampo porterà facilmente a un'amplificazione non specifica, diluizione appropriata di 5-50 volte. La quantità di stampo consigliata è 4 μL, cercare di non superare i 6 μL. Quando le prestazioni della reazione sono scarse, la concentrazione del primer può essere regolata nell'intervallo di 0,2-1,0 μmol/L.
2) Procedura di amplificazione PCR quantitativa a fluorescenza (metodo in due fasi)
| Ciclo fare un passo | Temperatura | Tempo | Cicli |
| Denaturazione iniziale | 95℃ | 30 secondo | 1 |
| Denaturazione | 95℃ | 10 secondo | 35-40 |
| Ricottura/Estensione* | 60℃ | 30 secondo | |
| Fase della curva di fusione | Impostazioni predefinite dello strumento | 1 | |
3)Procedura di amplificazione rapida per PCR quantitativa fluorescente (metodo in due fasi)
| Ciclo fare un passo | Temperatura | Tempo | Cicli |
| Denaturazione iniziale | 95℃ | 10 secondo | 1 |
| Denaturazione | 95℃ | 5 secondo | 40 |
| Ricottura/Estensione* | 60℃ | 10 secondo | |
| Fase della curva di fusione | Impostazioni predefinite dello strumento | 1 | |
* La temperatura di annealing/estensione e il tempo di estensione finale possono essere regolati in modo appropriato in base ai requisiti sperimentali. Il programma rapido è adatto per la maggior parte dei geni e il programma standard può essere provato per singoli geni complessi di struttura secondaria.
Appunti
- Questo prodotto è destinato esclusivamente alla ricerca.
- Per la vostra sicurezza, operate indossando camici da laboratorio e guanti monouso.
Versione EN20230908
Documenti:
11172-Hieff™ Fast Cell Direct. EN20230908.pdf
Pagamento e sicurezza
Le informazioni di pagamento vengono elaborate in modo sicuro. Non archiviamo i dettagli della carta di credito né abbiamo accesso alle informazioni sulla tua carta di credito.
Indagine
Potrebbe piacerti anche
FAQ
Il prodotto è solo per scopi di ricerca e non è destinato all'uso terapeutico o diagnostico su esseri umani o animali. Prodotti e contenuti sono protetti da brevetti, marchi e copyright di proprietà di Yeasen Biotechnology. I simboli dei marchi indicano il paese di origine, non necessariamente la registrazione in tutte le regioni.
Alcune applicazioni potrebbero richiedere ulteriori diritti di proprietà intellettuale di terze parti.
Yeasen è un sostenitore della scienza etica, convinto che la nostra ricerca debba affrontare questioni critiche garantendo al contempo sicurezza e standard etici.