Descrizione
Descrizione del prodotto
Hieff NGS™ Ultima Dual-mode RNA Library Prep Kit è un kit di costruzione di librerie di sequenziamento RNA completo per la piattaforma di sequenziamento Illumina® e MGI®, che include reagenti di frammentazione RNA, reagenti di trascrizione inversa, reagenti di sintesi ds-cDNA convenzionali e specifici per filamento e reagenti di amplificazione della libreria. La libreria di sequenziamento può essere costruita seguita dal kit di purificazione mRNA o dal kit di rimozione rRNA. Il modulo di sintesi a due filamenti è dotato di due buffer per soddisfare l'esigenza di una libreria convenzionale o di una libreria specifica per filamento. Tra questi, dTTP viene sostituito con dUTP nel buffer di sintesi a due filamenti specifico per filamento, quindi dUTP può essere aggiunto al secondo filamento di cDNA. La DNA polimerasi ad alta fedeltà utilizzata in questo kit non può amplificare il modello di DNA contenente uracile, ottenendo specificità per filamento. Tutti i reagenti forniti sono stati sottoposti a rigorosi controlli di qualità e verifica funzionale, garantendo la massima stabilità e riproducibilità della costruzione della libreria.
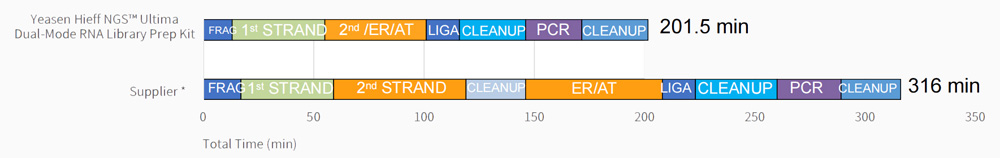
Flusso di lavoro
Componenti del prodotto
| Componenti | 12308ES24 | 12308ES96 | |||
| 12308-A | 2× Tampone Frag/Prime | 250 microlitri | 930 microlitri | ||
| 12308-B | Miscela enzimatica del 1° filamento | 48 microlitri | 192 microlitri | ||
| 12308-C | Reagente di specificità del filamento | 150 microlitri | 580 microlitri | ||
| 12308-D | Tampone del 2° filamento (dNTP) | 720 microlitri | 2×1440 μL | ||
| 12308-E | Tampone del 2° filamento (dUTP) | 720 microlitri | 2×1440 μL | ||
| 12308-F | Miscela enzimatica master 2nd Strand | 120 microlitri | 480 microlitri | ||
| 12308-G | Potenziatore della legatura | 720 microlitri | 2×1440 μL | ||
| 12308-H | Nuova DNA ligasi T4 | 120 microlitri | 480 microlitri | ||
| 12308-I | 2×Super Canace® II Mix ad alta fedeltà | 600 microlitri | 2×1200 μL | ||
| 12308-K | H2O senza nucleasi | 300 microlitri | 1000 microlitri | ||
Nota: questo kit è compatibile con entrambe le piattaforme Illumina e MGI, ma sono disponibili anche altre piattaforme Illumina o MGI Primer Mix (Cat# 13335 Primer Mix per Illumina e Cat# 13334 Primer Mix per MGI ) È necessario.
Spedizione e stoccaggio
I componenti del kit di preparazione della libreria mRNA Hieff NGS™ Ultima Dual-mode nella scatola I vengono spediti con impacchi di ghiaccio e possono essere conservati a 2-8°C per un anno.
I componenti del kit di preparazione della libreria mRNA Hieff NGS™ Ultima Dual-mode nella scatola II vengono spediti con ghiaccio secco e possono essere conservati a -20°C per un anno.
Attenzione
1 Operazione
1.1 Per la tua sicurezza e salute, indossa dispositivi di protezione individuale (DPI), come camici da laboratorio e guanti monouso, quando utilizzi questo prodotto. Questo prodotto è SOLO per uso di ricerca!
1.2 Scongelare i componenti a temperatura ambiente. Mescolare accuratamente capovolgendo più volte, centrifugare brevemente e mettere sul ghiaccio per l'uso.
1.3 Si raccomanda di eseguire ogni reazione in un termociclatore con coperchio riscaldato. Il termociclatore deve essere preriscaldato alla temperatura impostata prima dell'uso.
1.4 Sono necessarie forniture prive di contaminazione da RNasi e la pulizia regolare dell'area sperimentale.
1.5 Operazioni improprie possono molto probabilmente causare contaminazioni da aerosol, influenzando l'accuratezza del risultato. Si raccomanda l'isolamento fisico obbligatorio delle regioni di miscelazione della reazione PCR e delle regioni di analisi di purificazione del prodotto PCR. Dotato di apparecchiature quali pipette specializzate per la costruzione di librerie.
2 Legatura dell'adattatore
2.1 I kit Illumina o MGI Long Adapter (Barcoded Adapter) e i kit Short Adapter sono disponibili tra cui i clienti possono scegliere in base alle loro esigenze sperimentali.
2.2 Si raccomanda di selezionare adattatori commerciali di alta qualità. Se si selezionano adattatori fatti in casa, si prega di affidarsi a un'azienda con esperienza nella sintesi di primer NGS e di sottolineare la necessità di un rigoroso controllo della contaminazione. Inoltre, si raccomanda di preparare la soluzione di annealing del DNA in un banco pulito e di utilizzare un solo tipo di adattatore alla volta per evitare la contaminazione incrociata.
2.3 Scongelare gli adattatori sul ghiaccio o a 4°C; se si utilizza a temperatura ambiente, la temperatura del laboratorio non deve superare i 25°C per evitare che gli adattatori si denaturano.
2.4 La concentrazione dell'adattatore influenza direttamente l'efficienza della legatura e la resa della libreria. Il volume dell'adattatore aggiunto al kit è fissato a 5 μl. Si raccomanda di diluire gli adattatori con tampone 0,1×TE e gli adattatori diluiti possono essere conservati a 4°C per 48 ore. Nella tabella 1 è elencata la quantità di adattatore consigliata per diverse quantità di RNA in input.
Tabella 1-1 Quantità di adattatore Illumina consigliata per diversi input di RNA
| Inserisci RNA totale | Illumina Concentrazione delle scorte dell'adattatore |
| 10 ng | 1 micron |
| 100 ng | 1,5 micron |
| 500 ng | 3 micron |
| ≥1 μg | 5 micron |
Tabella 1-2 Quantità di adattatore MGI® consigliata per diversi RNA di input
| Inserisci RNA totale | MGI Concentrazione delle scorte dell'adattatore |
| 100-499 non | 2 micron |
| 500-4000 ng | 5 micron |
*L'utilizzo dell'adattatore può essere regolato in base ai diversi tipi di campioni di RNA totale e alla quantità di input.
3 Amplificazione della libreria
3.1 Sulla base della DNA polimerasi di prima generazione, la DNA polimerasi ad alta fedeltà nel kit ha notevolmente migliorato la sua uniformità di amplificazione e non presenta alcun bias di amplificazione.
3.2 Se l'adattatore indicizzato (noto anche come adattatore lungo o adattatore Y grande) viene legato al DNA target, è possibile utilizzare la miscela di primer fornita in questo kit per l'amplificazione; se per la legatura del DNA viene utilizzato un "adattatore corto" o un "adattatore Y piccolo", sono necessari primer indice per l'amplificazione.
3.3 I numeri dei cicli di amplificazione devono essere rigorosamente controllati. Un'amplificazione insufficiente può portare a una bassa resa della libreria; una sovraamplificazione può introdurre un aumento di bias, errori, letture duplicate, prodotti chimerici e accumulo di mutazioni di espansione. La Tabella 2 elenca i numeri di cicli raccomandati per l'amplificazione PCR.
Tabella 2 Numero di cicli raccomandato per generare la libreria di RNA*
| Inserisci RNA totale | Numero di cicli | |
| Non-stranded | Bloccato | |
| 10 ng | 15 | 15 |
| 100 ng | 14 | 14 |
| 500 ng | 12 | 13 |
| 1 μg | 11 | 12 |
Nota:*La resa della libreria non è correlata solo alla quantità di input e al numero di cicli di amplificazione ma anche influenzato dalla qualità dei campioni, dalle condizioni di frammentazione e dalle condizioni di ordinamento. Nel processo di costruzione della biblioteca, scegliere le condizioni più appropriate in base alla situazione effettiva.
4 Pulizia del DNA basata su perle e selezione delle dimensioni
4.1 Ci sono diversi passaggi nel processo di costruzione della libreria che richiedono perle magnetiche per la purificazione del DNA. Consigliamo le perle di selezione del DNA Hieff NGS™ (Yeasen Cat#12601) o le perle magnetiche AMPure® XP (Beckman Cat#A63880) per la purificazione del DNA e la selezione delle dimensioni.
4.2 Le sfere magnetiche devono essere portate a temperatura ambiente prima dell'uso, altrimenti la resa diminuirà e l'effetto di selezione delle dimensioni sarà compromesso.
4.3 Le sfere magnetiche devono essere mescolate bene tramite vortex o pipettaggio prima dell'uso.
4.4 Non aspirare le microsfere durante il trasferimento del surnatante, anche tracce di microsfere possono influire sulle reazioni successive.
4.5 L'etanolo all'80% deve essere preparato fresco, altrimenti l'efficienza del recupero ne risentirà.
4.6 Le perle magnetiche devono essere asciugate a temperatura ambiente prima di eluire il prodotto. Una secchezza insufficiente causerà facilmente l'influenza dell'etanolo residuo sulle reazioni successive; una secchezza eccessiva causerà la rottura delle perle magnetiche e ridurrà la resa di purificazione. Normalmente, l'essiccazione a temperatura ambiente per 3-5 minuti è sufficiente per consentire alle perle di asciugarsi completamente.
4.7 Se necessario, i campioni di DNA purificati o selezionati in base alle dimensioni eluiti nel tampone TE possono essere conservati a 4°C per 1-2 settimane o a -20°C per un mese.
5 Analisi della qualità della biblioteca
5.1 Normalmente, la qualità della libreria costruita può essere valutata tramite la distribuzione della lunghezza e il rilevamento della concentrazione.
5.2 Rilevamento della concentrazione della libreria: metodi basati su coloranti fluorescenti del DNA a doppio filamento, come Qubit®, PicoGreen®, ecc.; quantificazione assoluta basata su qPCR.
5.3 I metodi basati sulla rilevazione spettrale, come NanoDrop®, ecc., non sono applicabili alla rilevazione della concentrazione della libreria.
5.4 qPCR è raccomandato per il rilevamento della concentrazione della libreria: tramite Qubit®, PicoGreen® e altri metodi basati su coloranti fluorescenti del DNA a doppio filamento, non è possibile distinguere efficacemente tra prodotti legati agli adattatori a un'estremità, prodotti non legati agli adattatori a entrambe le estremità e altri prodotti a doppio filamento incompleti. La quantificazione assoluta di qPCR si basa sul principio di amplificazione PCR, che quantifica solo la libreria completa dell'adattatore a entrambe le estremità del campione (la libreria che può essere sequenziata), escludendo l'interferenza di librerie non sequenzianti che non sono legate all'adattatore alle estremità a singola estremità o a doppia estremità.
5.5 La rilevazione della distribuzione della lunghezza della libreria può essere eseguita da Agilent Bioanalyzer 2100 e da altre apparecchiature basate sul principio dell'elettroforesi capillare o della microfluidica.
Documenti:
Citazioni e riferimenti:
[1] Decifrare il microbioma intestinale della carpa erbivora attraverso un approccio multi-omico
M Li, H Liang, H Yang, Q Ding, R Xia, J Chen, W Zhou… - Microbioma, 2024 IF:19.4
[2] Lo screening CRISPR in pool identifica i corpi P come repressori della transizione epitelio-mesenchimale del cancro
L Fang, L Zhang, M Wang, Y He, J Yang, Z Huang… - Ricerca sul cancro, 2024 IF:12.7
[3] Il peptide 1 derivato da Klotho inibisce la senescenza cellulare nel rene fibrotico ripristinando l'espressione di Klotho tramite regolazione post-trascrizionale
X Zhang, L Li, H Tan, X Hong, Q Yuan, FF Hou… - Teranostica, 2024 IF:12.4
[4] Cellule staminali parzialmente rivestite con esoscheletro per il ripristino del miocardio infartuato
H He, Y Yuan, Y Wu, J Lu, X Yang, K Lu… - Materiali avanzati, 2023 IF: 29.4
[5] Innovazione genomica e riorganizzazione normativa durante l’evoluzione del genere di cotone Gossypium
M Wang, J Li, Z Qi, Y Long, L Pei, X Huang… - Nature Genetics, 2022 IF:41.379
[6] Gli alleli di rischio MTMR3 migliorano l'immunità IgA indotta dal recettore Toll-like 9 nella nefropatia da IgA
Y Wang, T Gan, S Qu, L Xu, Y Hu, L Liu, S Shi, J Lv… - Kidney International, 2023 IF:19.6
[7] Completamento diviso degli editor di base per ridurre al minimo le modifiche fuori bersaglio
X Xiong, K Liu, Z Li, FN Xia, XM Ruan, X He, JF Li - Piante della natura, 2023 IF:18.6
[8] Le vescicole della membrana esterna batterica ingegnerizzate che incapsulano gli adenovirus oncolitici migliorano l'efficacia della viroterapia del cancro aumentando l'autofagia delle cellule tumorali
W Ban, M Sun, H Huang, W Huang, S Pan… - Nature Communications, 2023 IF:16.6
[9] La resistenza delle cellule cancerose all’IFNγ può verificarsi tramite un’attività migliorata del percorso di riparazione delle rotture a doppio filamento
T Han, X Wang, S Shi, W Zhang, J Wang, Q Wu… - Ricerca sull'immunologia del cancro, 2023 IF: 12.0
[10] Terapia combinata basata su un sistema di nano-rilascio biomimetico a doppio bersaglio per superare la resistenza al cisplatino nel carcinoma epatocellulare
Y Huang, Q Kou, Y Su, L Lu, X Li, H Jiang… - Journal of Nanobiotechnology, 2023 IF: 10.9
[11] PIAS3 promuove la ferroptosi regolando TXNIP tramite il percorso di segnalazione TGF-β nel carcinoma epatocellulare
W Bao, J Wang, K Fan, Y Gao, J Chen - Ricerca farmacologica, 2023 IF:9.3
[12] Identificazione di bersagli proteici leganti l'RNA con HyperTRIBE in Saccharomyces cerevisiae
W Piao, C Li, P Sun, M Yang, Y Ding, W Song… - International Journal of Molecular Science, 2023 IF:5.6
[13] Il microbiota regola la transizione del ciclo vitale e la dinamica dei nematociti nelle meduse
S Peng, L Ye, Y Li, F Wang, T Sun, L Wang, W Hao… - Iscience, 2023 SE: 5.08
[14] I profili del trascrittoma e dei miRNA rivelano la rete regolatrice e i regolatori chiave della formazione dello xilema secondario nel pioppo “84K”
H Wang, P Zhao, Y He, Y Su, X Zhou… - Rivista internazionale di scienza molecolare, 2023 IF:5.6
Pagamento e sicurezza
Le informazioni di pagamento vengono elaborate in modo sicuro. Non archiviamo i dettagli della carta di credito né abbiamo accesso alle informazioni sulla tua carta di credito.
Indagine
Potrebbe piacerti anche
FAQ
Il prodotto è solo per scopi di ricerca e non è destinato all'uso terapeutico o diagnostico su esseri umani o animali. Prodotti e contenuti sono protetti da brevetti, marchi e copyright di proprietà di Yeasen Biotechnology. I simboli dei marchi indicano il paese di origine, non necessariamente la registrazione in tutte le regioni.
Alcune applicazioni potrebbero richiedere ulteriori diritti di proprietà intellettuale di terze parti.
Yeasen è un sostenitore della scienza etica, convinto che la nostra ricerca debba affrontare questioni critiche garantendo al contempo sicurezza e standard etici.

