Descrizione
Hieff NGSTM Il kit di preparazione della libreria RNA EvoMax (dUTP) è un premiscelato, actinomicina D libero e specifico del filamento totale Libreria di sequenziamento dell'RNA preparare kit compatibile con le piattaforme Illumina e MGI. Questo prodotto è disponibile in due tipologie: tubo o kit di sigillatura, e questo kit è più conveniente sia tramite dispositivo automatico di movimentazione dei liquidi o manipolazione manuale Il kit contiene reagenti di frammentazione dell'RNA, reagenti di trascrizione inversa, reagenti di sintesi del ds-cDNA specifico per filamento e reagenti di amplificazione della libreria. Può essere collegato al kit di purificazione dell'mRNA o al kit di rimozione dell'rRNA per fare ricerche su mRNA o lncRNA. Questo prodotto ottimizza il modulo di trascrizione inversa per ottenere una libreria ad alta specificità di catena senza actinomicina D, che garantisce la sicurezza degli sperimentatori in misura maggiore. Tutti i reagenti sono stati sottoposti a rigorosi controlli di qualità e convalida funzionale per massimizzare la stabilità e la ripetibilità della libreria preparazione.
Specifiche
| N. di cat. | 12340ES24/12340ES96/12340ES97/12340ES98 |
| Misurare | 24 T / 96 T / 96 T (automazione) / 96 T (piastra) |
Componenti
| Componenti n. | Nome | 12340ES24 | 12340ES96 | 12340ES97 (automazione) | 12340ES98 (Piatto )** |
| 12340-A | Tampone di frammentazione/innesco | 450 microlitri | 2×900 μL | 2×1064 μL | 8×266 μL |
| 12340-B | 1° Modulo di Reazione 2.0 | 192 microlitri | 768 microlitri | 960 microlitri | 8×120 μL |
| 12340-C | 2° Modulo di Reazione (dUTP) | 840 microlitri | 3×1120 μL | 3×1280 μL | 8×480 μL |
| 12340-D | Modulo di reazione di legatura | 840 microlitri | 3×1120 μL | 3×1280 μL | 8×480 μL |
| 12340-E | 2×Super Canace® II Mix ad alta fedeltà | 600 microlitri | 2×1200 μL | 2×1360 μL | 8×340 μL |
| * | Miscela di primer* | / | / | / | / |
Nota: * La notazione indica che il componente non è incluso nel kit. La miscela di primer è richiesta se si utilizzano gli adattatori completi, altrimenti non è't. Questo kit è compatibile con le piattaforme Illumina e MGI, ma necessita di una miscela di primer aggiuntiva (Cat # 13334 Primer Mix per MGITM e Cat # 13335 Primer Mix per Illumina®) per la piattaforma Illumina® o MGI se vengono utilizzati gli adattatori completi.
Figura
Disposizione del gruppo di reagenti della piastra.

Figura 1: disposizione dei reagenti del kit della libreria della piastra di tenuta

Figura 2. Componenti semplificati di Kit di preparazione della libreria RNA EvoMax.
Magazzinaggio
Questo prodotto deve essere conservato a -25~-15℃ per 1 anni.
Istruzioni
1. La quantità consigliata da aggiungere in un sistema da 20 μL è 0,1-1 unità (U), e la quantità di input può essere regolata in base ai risultati effettivi.
2. In base alle esigenze dell'esperimento, la concentrazione finale di dUTP può essere regolata tra 0,2 e 0,6 mM, ed è possibile aggiungere selettivamente 0,2 mM di dTTP.
3. Tempo di reazione a 25~37℃ può essere regolato entro 5~10 minuti in base alle esigenze sperimentali.
Appunti
1. Operazione
1) Si prega di operare con camici da laboratorio e guanti monouso,per la tua sicurezza.
2) Scongelare i componenti a temperatura ambiente. Mescolare accuratamente capovolgendo più volte, centrifugare brevemente e mettere su ghiaccio per l'uso.
3) Si raccomanda di eseguire ogni reazione in un termociclatore con coperchio riscaldato. Il termociclatore deve essere preriscaldato alla temperatura impostata prima dell'uso.
4) Sono necessarie forniture prive di contaminazione da RNasi e la pulizia regolare dell'area sperimentale. RNAZap di ThermoFisherTM è stato raccomandato l'uso di uno spray ad alta efficienza per la rimozione degli acidi nucleici, per eliminare la contaminazione da RNasi.
5) Operazioni improprie possono molto probabilmente causare contaminazioni da aerosol, influenzando l'accuratezza del risultato. Si raccomanda l'isolamento fisico obbligatorio delle regioni di miscelazione della reazione PCR e delle regioni di analisi della purificazione del prodotto PCR. Dotato di attrezzature quali pipette specializzate per la costruzione di biblioteche.
6) Questo reagente è monouso. L'uso multiplo è severamente vietato.
7) Questo prodotto è destinato esclusivamente alla ricerca.
2. Legatura dell'adattatore
1) I kit Illumina o MGI Long Adapter (Barcoded Adapter) e short Adapter sono disponibili tra cui i clienti possono scegliere in base alle proprie esigenze sperimentali.
2) Si raccomanda di selezionare adattatori commerciali di alta qualità. Se si selezionano adattatori fatti in casa, si prega di affidarsi a un'azienda con esperienza nella sintesi di primer NGS e di sottolineare la necessità di un rigoroso controllo della contaminazione. Inoltre, si raccomanda di preparare la soluzione di annealing del DNA in un banco pulito e di utilizzare un solo tipo di adattatore alla volta per evitare la contaminazione incrociata.
3) Si prega di scongelare gli adattatori sul ghiaccio o a 4°C; quando si utilizza a temperatura ambiente, la temperatura del laboratorio non deve superare i 25°C per evitare che gli adattatori si denaturano.
4) La concentrazione dell'adattatore influisce direttamente sull'efficienza della legatura e sulla resa della libreria. Il volume dell'adattatore aggiunto al kit è fissato a 5 μl. Si raccomanda di diluire gli adattatori con tampone 0,1×TE e gli adattatori diluiti possono essere conservati a 4°C per 48 ore. Tabella 1 elenca la quantità di adattatore consigliata per diverse quantità di RNA in input.
Tabella 1-1 Quantità di adattatore Illumina consigliata per diversi input di RNA
| Inserisci RNA totale | Concentrazione delle scorte dell'adattatore Illumina® |
| 10 ng | 1 micron |
| 100 ng | 1,5 micron |
| 500 ng | 3 micron |
| ≥1 μg | 5 micron |
Tabella 1-2 Quantità di adattatore MGI consigliata per diversi RNA di input
| Inserisci RNA totale | Concentrazione di stock di adattatore MGI® |
| 10~99 ng | 1 micron |
| 100~499 ng | 2 micron |
| 500~4000 ng | 5 micron |
* L'utilizzo dell'adattatore può essere regolato in base ai diversi tipi di campioni di RNA totale e alla quantità di input
3.Amplificazione della libreria
1) Sulla base della DNA polimerasi di prima generazione, la DNA polimerasi ad alta fedeltà nel kit ha notevolmente migliorato la sua uniformità di amplificazione e non presenta alcuna distorsione di amplificazione.
2) I numeri dei cicli di amplificazione devono essere rigorosamente controllati. Un'amplificazione insufficiente può portare a una bassa resa della libreria; una sovra-amplificazione può introdurre un aumento di bias, errori, letture duplicate, prodotti chimerici e accumulo di mutazioni di espansione. Nella Tabella 2 sono elencati i numeri di cicli raccomandati per l'amplificazione PCR.
3) Il numero di cicli consigliato nella Tabella 2 può soddisfare la stragrande maggioranza dei requisiti di preparazione della libreria. Se il campione è di scarsa qualità (ad esempio campioni FFPE con grave degradazione), il numero di cicli può essere aumentato in modo appropriato, in base alla situazione effettiva. Prestare attenzione a notare che l'mRNA varia da specie e tessuti diversi, il numero di cicli di amplificazione deve essere regolato.
Tabella 2 Input Volume totale di RNA e cicli di amplificazione raccomandati Tabella *
| Inserisci RNA totale | Numero di cicli |
| 10 ng | 16 |
| 100 ng | 14 |
| 500 ng | 12 |
| 1 μg | 11 |
[Nota]: *La resa della libreria non è solo correlata alla quantità di input e al numero di cicli di amplificazione, ma è anche influenzata dalla qualità dei campioni, dalle condizioni di frammentazione e dalle condizioni di ordinamento. Nel processo di costruzione della libreria, scegliere le condizioni più appropriate in base alla situazione effettiva.
4. Pulizia del DNA basata su microsfere e selezione delle dimensioni
1) Ci sono diversi passaggi nel processo di costruzione della libreria che richiedono perle magnetiche per la purificazione del DNA. Consigliamo le perle di selezione del DNA Hieff NGS™ (Yeasen Cat#12601) o le perle magnetiche AMPure® XP (Beckman Cat#A63880) per la purificazione del DNA e la selezione delle dimensioni.
2) La perla magnetica deve essere bilanciata a temperatura ambiente prima dell'uso, altrimenti la resa diminuirà e l'effetto di selezione delle dimensioni sarà influenzato.
3) Prima dell'uso, le sfere magnetiche devono essere mescolate bene tramite vortex o pipettaggio.
4) Non aspirare le microsfere durante il trasferimento del surnatante, anche tracce di microsfere potrebbero influire sulle reazioni successive.
5) L'etanolo all'80% deve essere preparato fresco, altrimenti l'efficienza del recupero ne risentirà.
6) Le perle magnetiche devono essere asciugate a temperatura ambiente prima di eluire il prodotto. Una secchezza insufficiente causerà facilmente l'alterazione delle reazioni successive da parte dei residui di etanolo; una secchezza eccessiva causerà la rottura delle perle magnetiche e ridurrà la resa di purificazione. Normalmente, l'essiccazione a temperatura ambiente per 3-5 minuti è sufficiente per consentire alle perle di asciugarsi completamente.
7) Se necessario, i campioni di DNA purificati o selezionati in base alle dimensioni eluiti nel tampone TE possono essere conservati a 4°C per 1-2 settimane o a -20°C per un mese.
5.Analisi della qualità della biblioteca
In genere, la qualità della libreria costruita può essere valutata mediante il rilevamento della concentrazione e il rilevamento della distribuzione della lunghezza.
6. Altro materiale
1) Kit di arricchimento dell'mRNA: Hieff NGS® mRNA Isolation Master Kit V2 (Yeasen Cat # 12629).
2) Kit di rimozione dell'rRNA: Hieff NGS® MaxUp Human rRNA Depletion Kit (rRNA e ITS / ETS) (Yeasen Cat # 12257) o altro kit di rimozione dell'rRNA.
3) Purificazione dell'RNA da sfere magnetiche: Hieff NGS® RNA Cleaner (Yeasen Cat # 12602) o altri prodotti equivalenti.
4) Perle magnetiche purificate dal DNA: perle di selezione del DNA Hieff NGS® (Yeasen Cat # 12601) o perle AMPure® XP (A63880) o altri prodotti equivalenti.
5) Controllo qualità dell'RNA: Agilent 2100 Bioanalyzer RNA 6000 Nano / Pico Chip o altri prodotti equivalenti.
6) Adattatori: adattatore completo per uso Illumina® (cod. cat. 13519-13520 o altro equivalente) o adattatore completo per uso MGI® (cod. cat. 13360-13362 o altro equivalente).
7. Controllo di qualità della biblioteca
Chip Agilent 2100 Bioanalyzer DNA 1000/chip ad alta sensibilità o altri prodotti equivalenti; reagenti per la quantificazione della libreria.
8. Altri materiali
Etanolo anidro, acqua ultrapura sterile, testa della pistola a basso assorbimento, provetta PCR, telaio magnetico, strumento PCR, ecc.
Diagramma di flusso

Figura 1: Processo di costruzione della libreria RNA
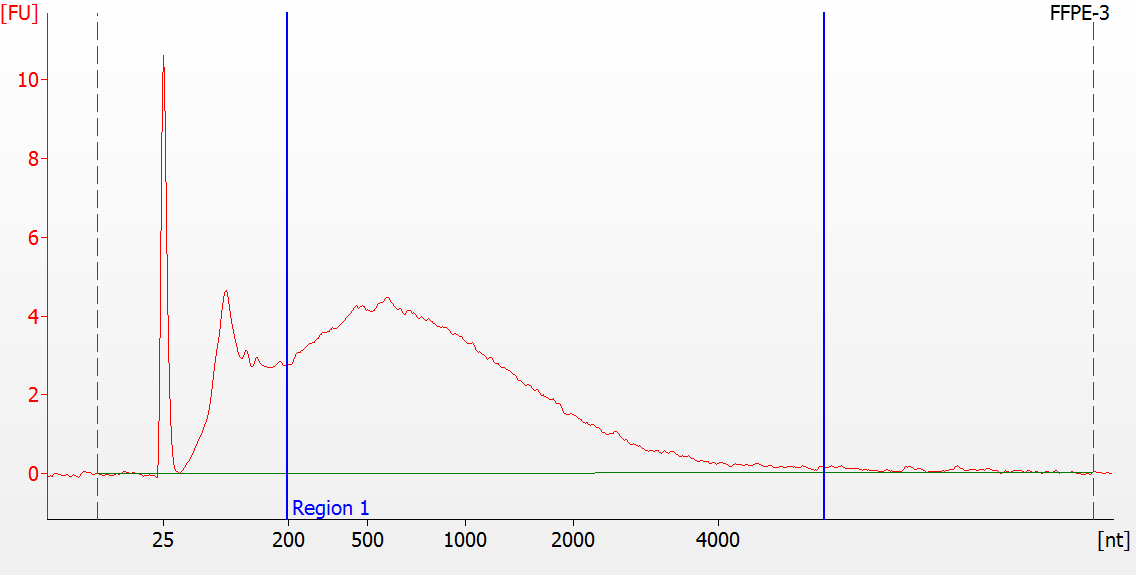
Preparazione della libreria RNA per campioni FFPE di diversa qualità
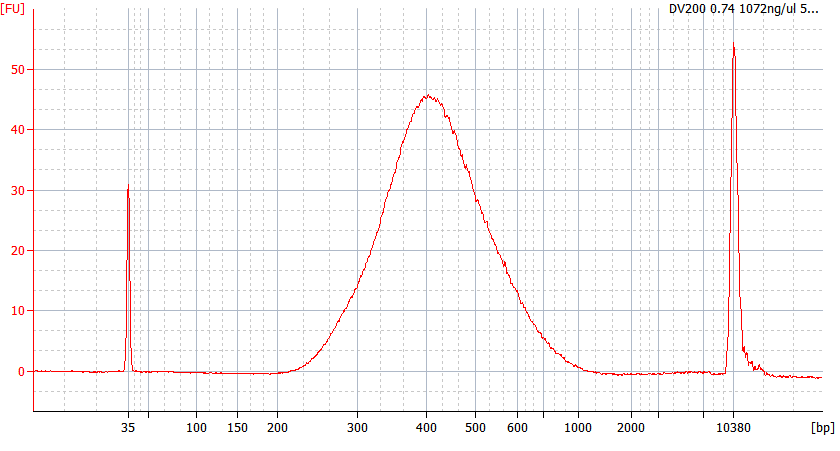
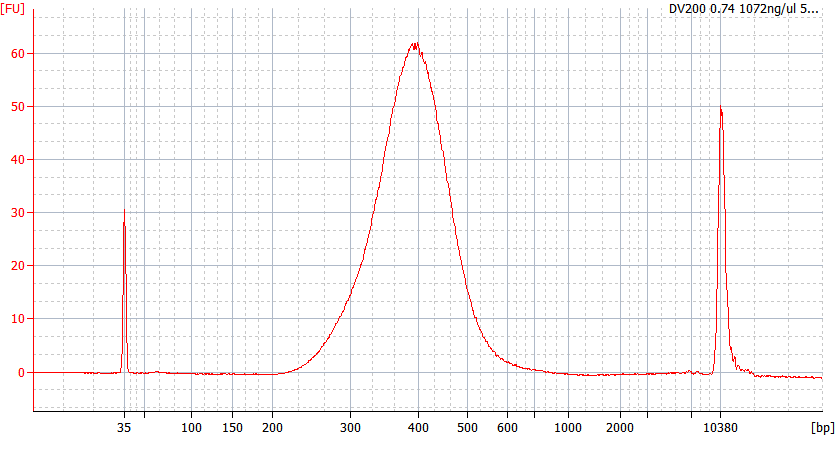
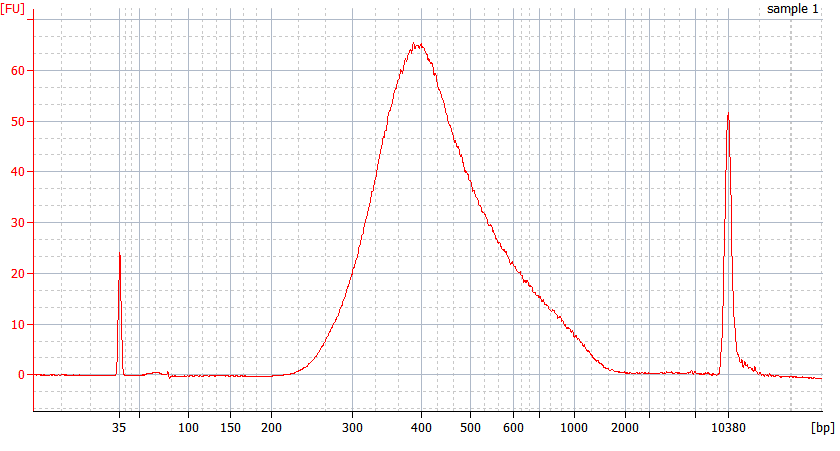
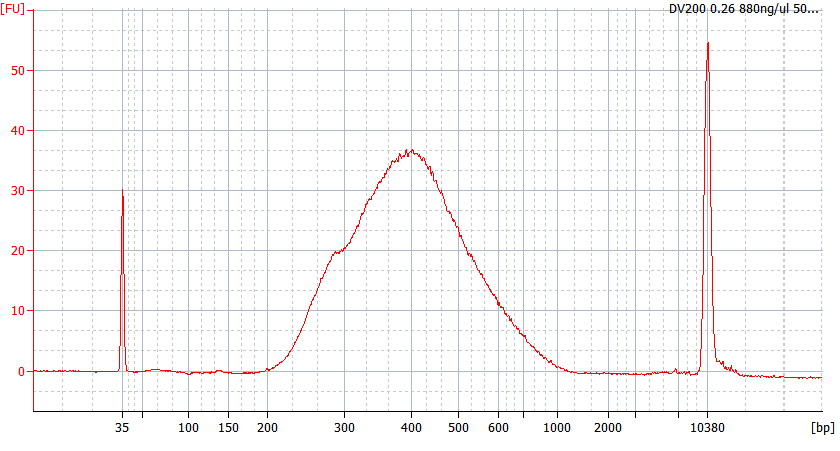
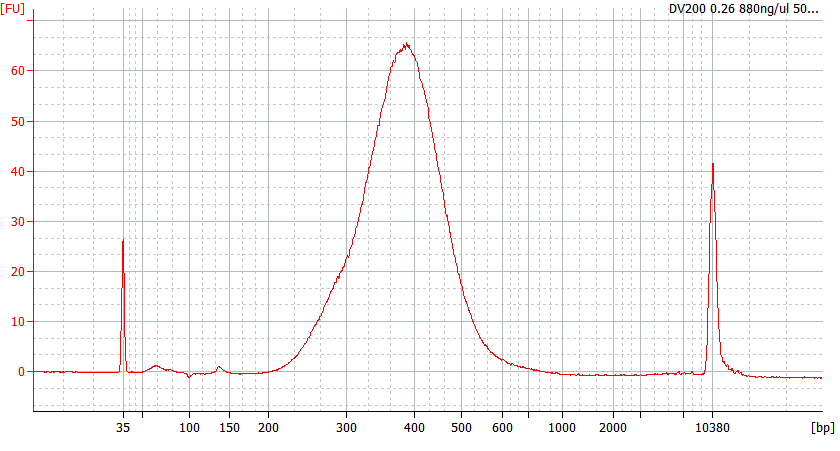
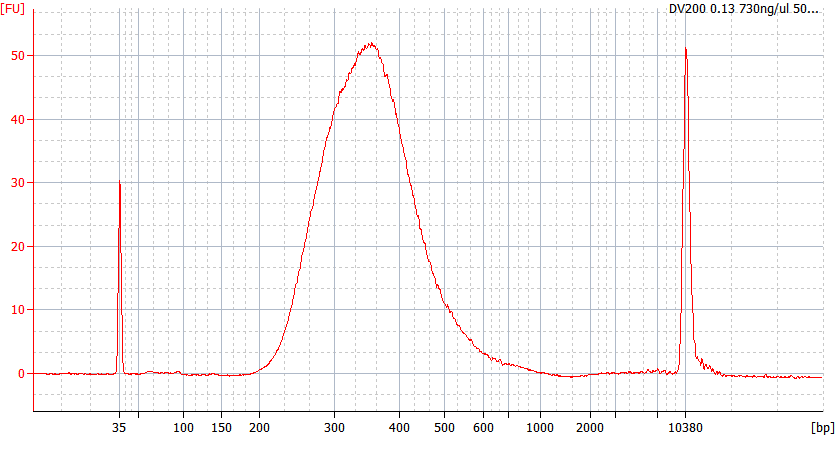
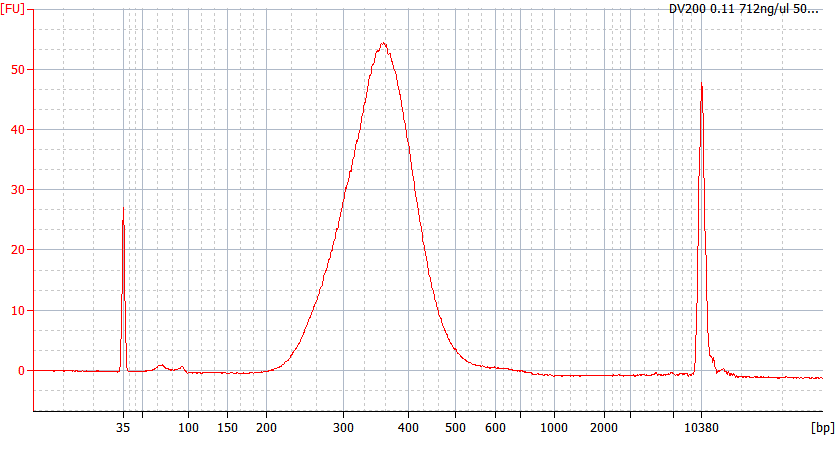
Per l'RNA FFPE gravemente degradato (DV 200 <50%) e per campioni con basso input, raccomandiamo un protocollo di doppia purificazione dopo la legatura dell'adattatore per ridurre la perdita della libreria.
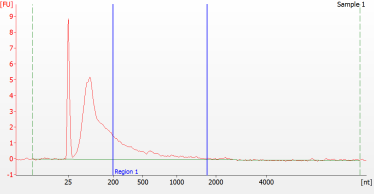
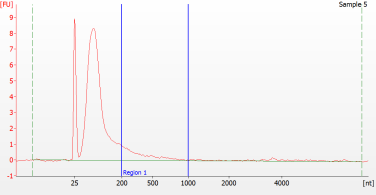
Modelli di picco di campioni FFPE di diversa qualità
Pagamento e sicurezza
Le informazioni di pagamento vengono elaborate in modo sicuro. Non archiviamo i dettagli della carta di credito né abbiamo accesso alle informazioni sulla tua carta di credito.
Indagine
Potrebbe piacerti anche
FAQ
Il prodotto è solo per scopi di ricerca e non è destinato all'uso terapeutico o diagnostico su esseri umani o animali. Prodotti e contenuti sono protetti da brevetti, marchi e copyright di proprietà di Yeasen Biotechnology. I simboli dei marchi indicano il paese di origine, non necessariamente la registrazione in tutte le regioni.
Alcune applicazioni potrebbero richiedere ulteriori diritti di proprietà intellettuale di terze parti.
Yeasen è un sostenitore della scienza etica, convinto che la nostra ricerca debba affrontare questioni critiche garantendo al contempo sicurezza e standard etici.