Descrizione
Le perle di selezione del DNA Hieff NGS™ sono preparate in base al principio SPRI (Solid Phase Reverse Immobilization) e sono applicabili per la purificazione del DNA e la selezione delle dimensioni durante la preparazione di librerie di sequenziamento di nuova generazione (NGS). Le perle di selezione del DNA Hieff NGS™ sono compatibili con vari kit di preparazione di librerie di DNA e RNA e sono una buona alternativa a Perle AMPure.
Componenti
| Componenti n. | Nome | 12601ES08 | 12601ES56 | 12601ES75 |
| 12601 | Perle di selezione del DNA Hieff NGS™ | 5 ml | 60 ml | 450 ml |
Specifiche
| Linea di prodotti | Perle di pulizia e selezione del DNA |
| Materiale di partenza | Il DNA |
| Compatibilità | Il DNA |
| Tecnologia di isolamento | Perlina magnetica |
| Tipo di prodotto finale | Il DNA |
| Per l'uso con (applicazione) | DNA celan up, selezione delle dimensioni del DNA |
Spedizione e stoccaggio
Le perle vengono spedite con ghiaccio e possono essere conservate a una temperatura compresa tra 2°C e 8°C per un anno.
Istruzioni
- 1. Preparazione
Equilibrare le perle di selezione a temperatura ambiente per almeno 30 minuti prima dell'uso.
- 2. Selezione della dimensione del DNA
Il flusso operativo della selezione delle dimensioni è illustrato nella Figura 1 e il protocollo è il seguente.
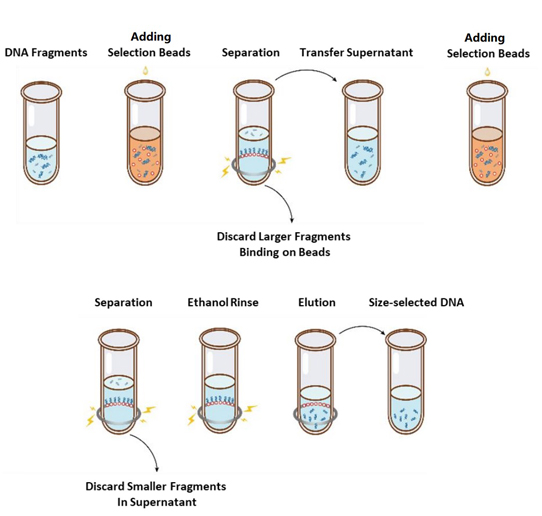
Figura 1. Diagramma di flusso della selezione delle dimensioni del DNA
2.1 Mescolare accuratamente le perle agitandole con un vortex o pipettandole su e giù ogni volta prima dell'uso.
2.2 Aggiungere il primo giro di sfere di selezione al campione (fare riferimento alla Tabella 1). Mescolare accuratamente agitando o pipettando su e giù almeno 10 volte.
2.3 Incubare a temperatura ambiente per 5 minuti.
2.4 Centrifugare brevemente la provetta e posizionarla su un supporto magnetico. Quando la soluzione è limpida (circa 5 min), trasferire il surnatante in una nuova provetta per PCR.
2.5 Aggiungere il secondo giro di sfere di selezione al campione dal passaggio 2.4 secondo la Tabella 1. Mescolare accuratamente agitando con vortex o pipettando su e giù almeno 10 volte.
2.6 Incubare a temperatura ambiente per 5 minuti.
2.7 Centrifugare brevemente la provetta e posizionarla su un supporto magnetico. Quando la soluzione è limpida (circa 5 min), aspirare il surnatante e scartarlo.
2.8 Mantenere la provetta nel supporto magnetico e aggiungere 200 μL di etanolo all'80% appena preparato senza toccare le perle, incubare a temperatura ambiente per 30 sec. Aspirare l'etanolo e scartarlo.
2.9 Ripetere il passaggio 2.8 una volta per un totale di due lavaggi.
2.10 Rimuovere l'etanolo residuo con puntali per pipette da 10 µL. Tenere la provetta nel supporto magnetico, asciugare all'aria le perle di selezione con il coperchio aperto finché non compaiono delle crepe (circa 5 min).
Nota: non far seccare troppo le perle di selezione. Ciò potrebbe causare una riduzione del recupero del DNA target.
2.11 Rimuovere la provetta dal supporto magnetico. Aggiungere una quantità appropriata di ddH2O (≥20 µL) e mescolare accuratamente agitando o pipettando su e giù almeno 10 volte.
2.12 Incubare a temperatura ambiente per 5 minuti.
Centrifugare brevemente la provetta e posizionarla sul supporto magnetico. Quando la soluzione è limpida (circa 5 minuti), trasferire 20 μL del surnatante in una nuova provetta.
- 3. Condizioni consigliate per la selezione delle dimensioni del DNA
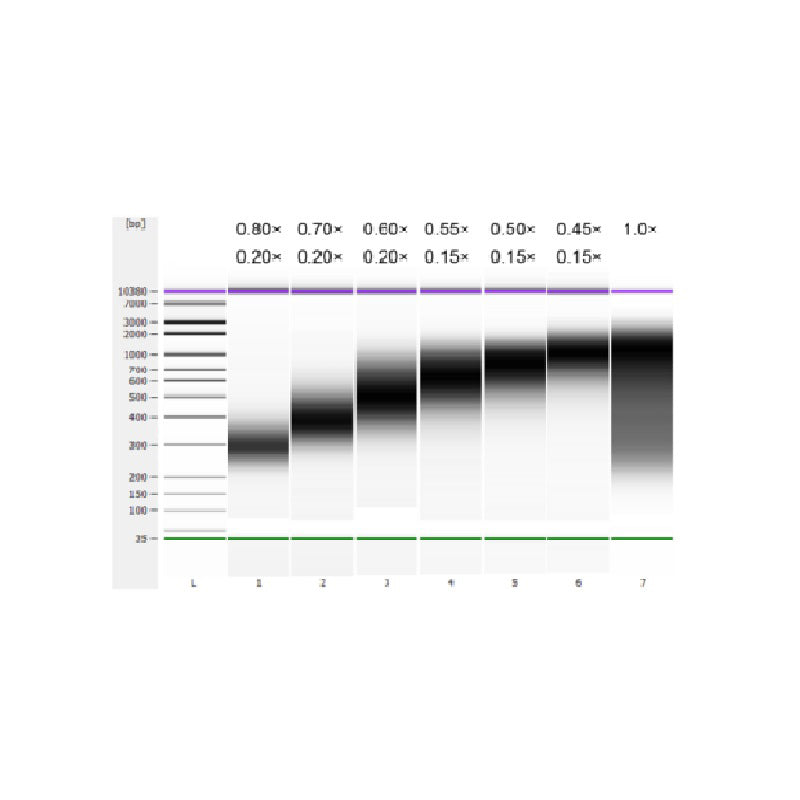
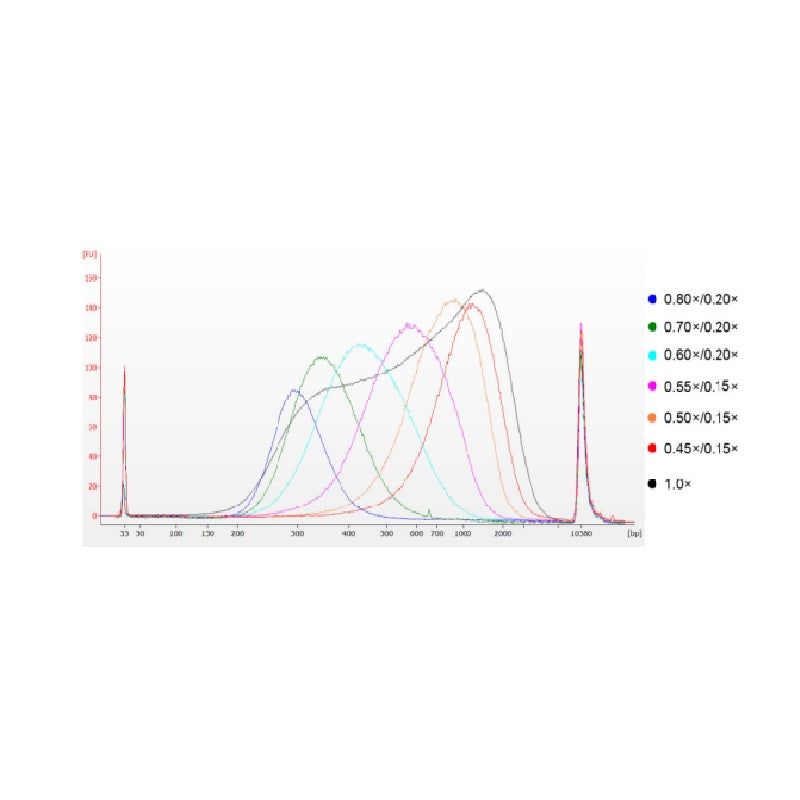
Il DNA del timo del vitello è stato frammentato mediante sonicazione per preparare un frammento di 100-1.000 bp e sono stati eseguiti due cicli di selezione delle dimensioni secondo la Tabella 1. I risultati sono stati analizzati utilizzando Agilent 2100 Bioanalyzer (Figura 2).
Tabella 1. Condizioni consigliate per la selezione delle dimensioni del DNA
| Lunghezza del frammento di DNA | 250-350 punti base | 320-420 anni fa | 450-550 anni fa | 550-700 anni fa | 700-900 anni fa | 800-1.000 punti base |
| Rapporto delle perle: DNA per il 1° Round | 0,80× | 0,70× | 0,60× | 0,55× | 0,50× | 0,45× |
| Rapporto delle perle: DNA per il 2° Girare | 0,20× | 0,20× | 0,20× | 0,15× | 0,15× | 0,15× |
Nota: "×" nella tabella indica il volume del campione di DNA. Ad esempio, se la lunghezza dell'inserto della libreria è 250 bp e il volume del campione di DNA è 100 μL, il volume delle sfere magnetiche utilizzate nel primo round di ordinamento è 0,80×100 μL=80 μL; il volume delle sfere magnetiche utilizzate nel secondo round di ordinamento è 0,20×100 μL=20 μL.
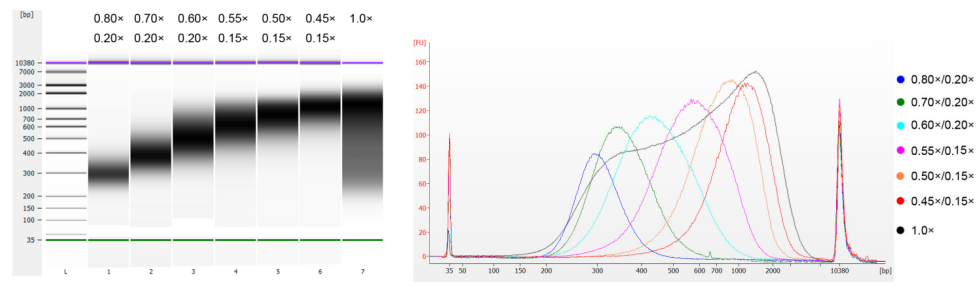
Figura 2. Elettroferogramma del chip di DNA ad alta sensibilità Agilent 2100
Note:
1. Per la vostra sicurezza e salute, vi preghiamo di indossare camici da laboratorio e guanti monouso durante il funzionamento.

Citato da "Integrazione specifica della sequenza da parte della famiglia 1 casposasi da Candidatus Nitrosopumilus koreensis AR1. Acidi nucleici Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725"

Citato da "Recente infezione da Wolbachia altera le comunità microbiche nelle popolazioni selvatiche di Laodelphax striatellus. Microbioma. 2020;8(1):104. Pubblicato il 2 luglio 2020. doi:10.1186/s40168-020-00878-x"
[1] Wang X, Yuan Q, Zhang W, et al. Integrazione specifica della sequenza da parte della famiglia 1 casposasi da Candidatus Nitrosopumilus koreensis AR1. Acidi nucleici Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725(IF:16.971)
[2] Duan XZ, Sun JT, Wang LT, et al. Una recente infezione da Wolbachia altera le comunità microbiche nelle popolazioni selvatiche di Laodelphax striatellus. Microbioma. 2020;8(1):104. Pubblicato il 2 luglio 2020. doi:10.1186/s40168-020-00878-x(IF:11.607)
[3] Canzone B, Almatrafi E, Sang F, et al.Gestione dei sedimenti trattati con Fenton con biochar e compost di letame di pecora: effetti sulle caratteristiche evolutive della comunità batterica. J Environ Manage. 2022;316:115218. doi:10.1016/j.jenvman.2022.115218(IF:6.789)
[4] Huang C, Mei Q, Lou L, et al. Colite ulcerosa in risposta al trapianto di microbiota fecale tramite modulazione del microbiota intestinale e dell'equilibrio delle cellule Th17/Treg. Cellule. 2022;11(11):1851. Pubblicato il 5 giugno 2022. doi:10.3390/cells11111851(IF:6.600)
[5] Ghosh S, Yang X, Wang L, Zhang C, Zhao L. L'alimentazione prebiotica in fase attiva altera il microbiota intestinale, induce un'alleviamento indipendente dal peso della steatosi epatica e del colesterolo sierico nei topi alimentati con una dieta ricca di grassi. Comput Struct Biotechnol J. 2020;19:448-458. Pubblicato il 24 dicembre 2020. doi:10.1016/j.csbj.2020.12.011(IF:6.018)
[6] Gao X, Yu B, Yu J, et al. Profilazione dello sviluppo della digestione dei carboidrati dietetici nei suinetti. Front Microbiol. 2022;13:896660. Pubblicato il 29 aprile 2022. doi:10.3389/fmicb.2022.896660(IF:5.640)
[7] Li P, Zhang Y, Yan F, Zhou X. Caratteristiche di un batteriofago, vB_Kox_ZX8, isolato da Klebsiella oxytoca clinica e suo effetto terapeutico sulla batteriemia dei topi. Front Microbiol. 2021;12:763136. Pubblicato il 3 dicembre 2021. doi:10.3389/fmicb.2021.763136(IF:5.640)
[8] Lin Z, Luo P, Huang D, Wu Y, Li F, Liu H. Strategia basata su multi-omica per l'analisi della tossicità dell'acrilamide nel modello Saccharomyces cerevisiae. Chem Biol Interact. 2021;349:109682. doi:10.1016/j.cbi.2021.109682(IF:5.194)
[9] Sun X, Lv W, Wang Y, et al. Il gene Mrgprb2 svolge un ruolo nelle reazioni anafilattoidi indotte dall'iniezione di Houttuynia cordata. J Etnofarmaco. 2022;289:115053. doi:10.1016/j.jep.2022.115053(IF:4.360)
[10] Ma H, Lai B, Zan C, Di X, Zhu X, Wang K. GLO1 contribuisce alla resistenza ai farmaci di Escherichia coli attraverso l'induzione del tipo PER di β-lattamasi a spettro esteso. Infect Drug Resist. 2022;15:1573-1586. Pubblicato il 5 aprile 2022. doi:10.2147/IDR.S358578(IF:4.003)
[11] Zhong Y, Zhao W, Tang Z, et al. Analisi trascrittomica comparativa delle diverse fasi di sviluppo dell'ovaio nel gambero rosso Procambarus clarkii. BMC Genomics. 2021;22(1):199. Pubblicato il 21 marzo 2021. doi:10.1186/s12864-021-07537-x(IF:3.969)
[12] Lian C, Yang H, Lan J, et al. L'analisi comparativa dei genomi dei cloroplasti rivela relazioni filogenetiche e variazioni intraspecifiche nella pianta medicinale Isodon rubescens. PLoS One. 2022;17(4):e0266546. Pubblicato il 6 aprile 2022. doi:10.1371/journal.pone.0266546(IF:3.240)
[13] Diao G, Huang J, Zheng X et al. Prostaglandina E2 svolge un duplice ruolo nella regolazione della migrazione delle cellule dendritiche. Int J Mol Med. 2021;47(1):207-218. doi:10.3892/ijmm.2020.4801(IF:3.098)
[14] Bing XL, Zhao DS, Peng CW, Huang HJ, Hong XY. Somiglianze e variazioni spaziali delle comunità batteriche e fungine nelle popolazioni di cicalina del riso (Hemiptera: Delphacidae). Insect Sci. 2020;27(5):947-963. doi:10.1111/1744-7917.12782(IF:2.791)
[15] Li X, Zhou S, Zhang J, Zhou Z, Xiong Q. Cambiamenti direzionali nella comunità batterica intestinale nelle larve della mosca soldato nera (Hermetia illucens). Animali (Basilea). 2021;11(12):3475. Pubblicato il 6 dicembre 2021. doi:10.3390/ani11123475(IF:2.752)
[16] Yang J, Peng Y, Kong W. Identificazione e sequenza completa del genoma del virus criptico del gelso 1. Arch Virol. 2022;167(2):687-690. doi:10.1007/s00705-021-05350-1(IF:2.574)
[17] Chang Y, Xia X, Sui L, et al. La colonizzazione endofitica di funghi entomopatogeni aumenta la resistenza alle malattie delle piante modificando la comunità batterica endofitica. J Basic Microbiol. 2021;61(12):1098-1112. doi:10.1002/jobm.202100494(IF:2.281)
[18] Ding CY, Ma YM, Li B, et al. Identificazione e analisi funzionale dei geni differenzialmente espressi in Myzus persicae (Hemiptera: Aphididae) in risposta al trans-anetolo. J Insect Sci. 2022;22(1):3. doi:10.1093/jisesa/ieab094(IF:1.857)
Pagamento e sicurezza
Le informazioni di pagamento vengono elaborate in modo sicuro. Non archiviamo i dettagli della carta di credito né abbiamo accesso alle informazioni sulla tua carta di credito.
Indagine
Potrebbe piacerti anche
FAQ
Il prodotto è solo per scopi di ricerca e non è destinato all'uso terapeutico o diagnostico su esseri umani o animali. Prodotti e contenuti sono protetti da brevetti, marchi e copyright di proprietà di Yeasen Biotechnology. I simboli dei marchi indicano il paese di origine, non necessariamente la registrazione in tutte le regioni.
Alcune applicazioni potrebbero richiedere ulteriori diritti di proprietà intellettuale di terze parti.
Yeasen è un sostenitore della scienza etica, convinto che la nostra ricerca debba affrontare questioni critiche garantendo al contempo sicurezza e standard etici.