Descrizione
Hieff NGSTM Italiano: Le perle di selezione del DNA vengono preparate in base al principio SPRI (Solid Phase Reverse Immobilization) ed è applicabile per la purificazione del DNA e la selezione delle dimensioni durante la preparazione di librerie di sequenziamento di nuova generazione (NGS). Hieff NGSTM Italiano: Le DNA Selection Beads sono compatibili con vari kit di preparazione di librerie di DNA e RNA. Questo prodotto è fabbricato in una struttura ultra-pulita con un elevato livello di pulizia e può essere utilizzato per la fase di purificazione del DNA durante i processi di rilevamento dei patogeni.
Specifiche
| Cat.Nlo. | 17266È03 /17266ES75 |
| Misurare | 1 ml/450 ml |
Componenti
| Componenti n. | Nome | 17266È03 | 17266ES75 |
| 17266 | Hieff NGS UCF.ME Perle di selezione del DNA | 1 ml | 450 ml |
Magazzinaggio
Questo prodotto deve essere conservato a 2~8℃ per 18 mesi.
Spedizione e stoccaggio
Le perle vengono spedite con ghiaccio e possono essere conservate a una temperatura compresa tra 2°C e 8°C per un anno.
Istruzioni
- 1. Preparazione
Equilibrare le perle di selezione a temperatura ambiente per almeno 30 minuti prima dell'uso.
- 2. Selezione della dimensione del DNA
Il flusso operativo della selezione delle dimensioni è illustrato nella Figura 1 e il protocollo è il seguente.
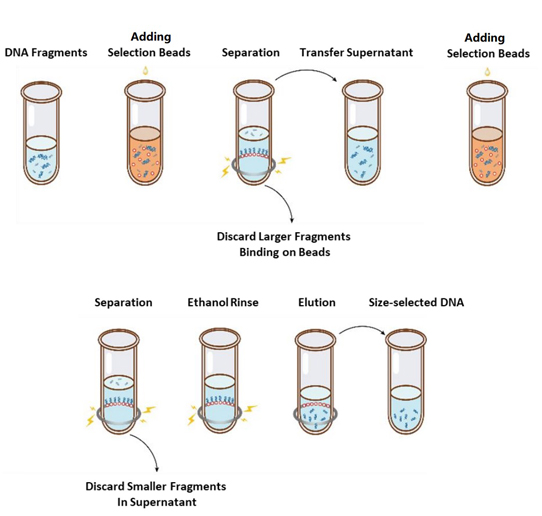
Figura 1. Diagramma di flusso della selezione delle dimensioni del DNA
2.1 Mescolare accuratamente le perle agitandole con un vortex o pipettandole su e giù ogni volta prima dell'uso.
2.2 Aggiungere il primo giro di sfere di selezione al campione (fare riferimento alla Tabella 1). Mescolare accuratamente agitando o pipettando su e giù almeno 10 volte.
2.3 Incubare a temperatura ambiente per 5 minuti.
2.4 Centrifugare brevemente la provetta e posizionarla su un supporto magnetico. Quando la soluzione è limpida (circa 5 min), trasferire il surnatante in una nuova provetta per PCR.
2.5 Aggiungere il secondo giro di sfere di selezione al campione dal passaggio 2.4 secondo la Tabella 1. Mescolare accuratamente agitando con vortex o pipettando su e giù almeno 10 volte.
2.6 Incubare a temperatura ambiente per 5 minuti.
2.7 Centrifugare brevemente la provetta e posizionarla su un supporto magnetico. Quando la soluzione è limpida (circa 5 min), aspirare il surnatante e scartarlo.
2.8 Mantenere la provetta nel supporto magnetico e aggiungere 200 μL di etanolo all'80% appena preparato senza toccare le perle, incubare a temperatura ambiente per 30 sec. Aspirare l'etanolo e scartarlo.
2.9 Ripetere il passaggio 2.8 una volta per un totale di due lavaggi.
2.10 Rimuovere l'etanolo residuo con puntali per pipette da 10 µL. Tenere la provetta nel supporto magnetico, asciugare all'aria le perle di selezione con il coperchio aperto finché non compaiono delle crepe (circa 5 min).
Nota: non far seccare troppo le perle di selezione. Ciò potrebbe causare una riduzione del recupero del DNA target.
2.11 Rimuovere la provetta dal supporto magnetico. Aggiungere una quantità appropriata di ddH2O (≥20 µL) e mescolare accuratamente agitando o pipettando su e giù almeno 10 volte.
2.12 Incubare a temperatura ambiente per 5 minuti.
Centrifugare brevemente la provetta e posizionarla sul supporto magnetico. Quando la soluzione è limpida (circa 5 minuti), trasferire 20 μL del surnatante in una nuova provetta.
- 3. Condizioni consigliate per la selezione delle dimensioni del DNA
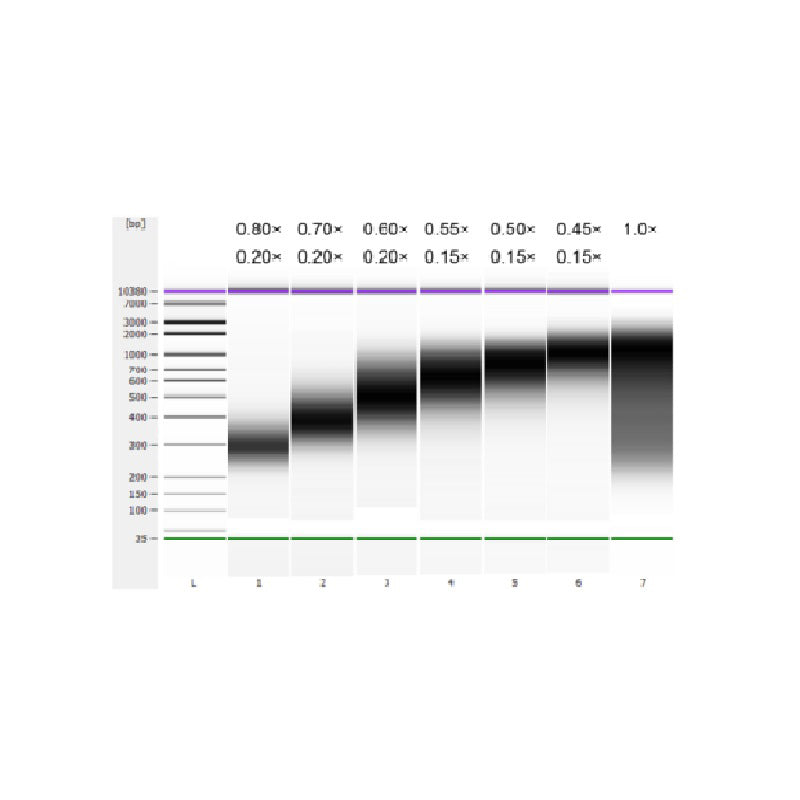
Il DNA del timo del vitello è stato frammentato mediante sonicazione per preparare un frammento di 100-1.000 bp e sono stati eseguiti due cicli di selezione delle dimensioni secondo la Tabella 1. I risultati sono stati analizzati utilizzando Agilent 2100 Bioanalyzer (Figura 2).
Tabella 1. Condizioni consigliate per la selezione delle dimensioni del DNA
| Lunghezza del frammento di DNA | 250-350 punti base | 320-420 anni fa | 450-550 anni fa | 550-700 anni fa | 700-900 anni fa | 800-1.000 punti base |
| Rapporto delle perle: DNA per il 1° Round | 0,80× | 0,70× | 0,60× | 0,55× | 0,50× | 0,45× |
| Rapporto delle perle: DNA per il 2° Girare | 0,20× | 0,20× | 0,20× | 0,15× | 0,15× | 0,15× |
Nota: "×" nella tabella indica il volume del campione di DNA. Ad esempio, se la lunghezza dell'inserto della libreria è 250 bp e il volume del campione di DNA è 100 μL, il volume delle sfere magnetiche utilizzate nel primo round di ordinamento è 0,80×100 μL=80 μL; il volume delle sfere magnetiche utilizzate nel secondo round di ordinamento è 0,20×100 μL=20 μL.
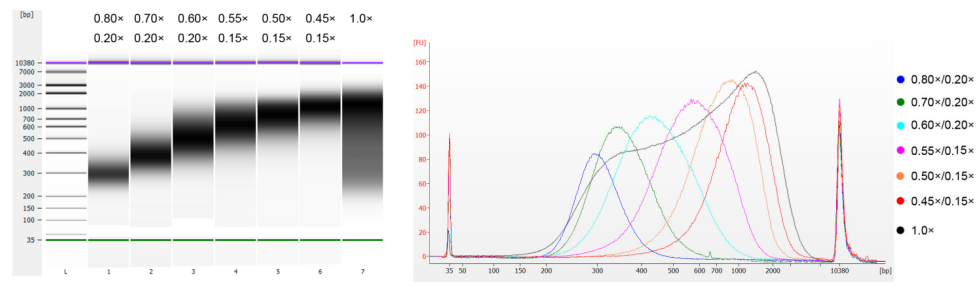
Figura 2. Elettroferogramma del chip di DNA ad alta sensibilità Agilent 2100
Note:1. Per la vostra sicurezza e salute, vi preghiamo di indossare camici da laboratorio e guanti monouso durante il funzionamento.
Pagamento e sicurezza
Le informazioni di pagamento vengono elaborate in modo sicuro. Non archiviamo i dettagli della carta di credito né abbiamo accesso alle informazioni sulla tua carta di credito.
Indagine
Potrebbe piacerti anche
FAQ
Il prodotto è solo per scopi di ricerca e non è destinato all'uso terapeutico o diagnostico su esseri umani o animali. Prodotti e contenuti sono protetti da brevetti, marchi e copyright di proprietà di Yeasen Biotechnology. I simboli dei marchi indicano il paese di origine, non necessariamente la registrazione in tutte le regioni.
Alcune applicazioni potrebbero richiedere ulteriori diritti di proprietà intellettuale di terze parti.
Yeasen è un sostenitore della scienza etica, convinto che la nostra ricerca debba affrontare questioni critiche garantendo al contempo sicurezza e standard etici.