Descrizione
Il kit di rilevamento del micoplasma GMyc-PCR utilizza principalmente il metodo PCR per rilevare l'infezione da micoplasma di vari materiali biologici (come colture cellulari, secrezioni animali sperimentali, siero animale, ecc.). Combina diversi vantaggi: sensibile, specifico, rapido e può essere rilevato direttamente con i supernatanti delle colture cellulari. Questo prodotto rileva il micoplasma in materiali biologici come le cellule coltivate mediante il metodo PCR. I primer utilizzati sono progettati in base alla regione conservata della sequenza 16S-23S rRNA del micoplasma e amplificano specificamente solo il DNA del micoplasma, con elevata sensibilità e specificità di rilevamento. L'amplificazione PCR e l'analisi dell'elettroforesi richiedono solo poche ore e l'operazione è comoda e semplice.
La coltura cellulare è un esperimento comune nella ricerca sulle scienze della vita. A differenza di altri metodi sperimentali comunemente usati, la coltura cellulare è un processo dinamico continuo e le cellule spesso rispondono a errori di manipolazione o contaminanti che spesso mostrano stati cellulari anomali o aspetto del mezzo. Se è contaminata da micoplasmi, la morfologia cellulare non presenta cambiamenti evidenti ed è facile trascurarla. Spesso non viene individuata finché l'inquinamento non è molto grave. Possono esserci centinaia di micoplasmi sulla membrana cellulare contaminata, questi micoplasmi competono per i nutrienti e rilasciano metaboliti tossici, influenzando seriamente i risultati sperimentali.
Studi hanno dimostrato che almeno 20 tipi di micoplasma possono contaminare le cellule, tra cui i più comuni sono: Mycoplasma orale (M. orale), Mycoplasma arginina (M. arginino), Mycoplasma hyorhinis (M. hyorhinis), Mycoplasma fermentum (M. fermentano), Mycoplasma hominis (M. uomo), Mycoplasma salivarius (M. salivario), Mycoplasma polmonare (M. polmone) e Mycoplasma pera (M. pirum). Il tasso di contaminazione da micoplasma delle cellule coltivate varia dal 4% al 92%. Le fonti di contaminazione includono l'ambiente di lavoro, l'operatore stesso (alcuni micoplasmi sono la normale flora del corpo umano), il terreno di coltura, il siero, la contaminazione incrociata delle cellule, l'attrezzatura sperimentale e la contaminazione utilizzata del tessuto o dell'organo originale da cui sono state preparate le cellule.
Identificare la causa sottostante dei problemi durante la coltura cellulare è un compito difficile e che richiede molto tempo, in cui si dovrebbero sospettare eventuali cambiamenti improvvisi e sono necessarie buone pratiche di test e test regolari per la contaminazione da micoplasma. Esistono molti metodi per la rilevazione del micoplasma, come la coltura diretta, la colorazione a fluorescenza del DNA, ELISA e metodi PCR.
Caratteristica
- I primer utilizzati sono stati progettati in base alla regione conservata della sequenza dell'rRNA 16S-23S del Mycoplasma
- Solo il DNA del micoplasma amplificato specificamente
- Ottima sensibilità e specificità
- Sono state rilevate più di 20 specie di micoplasmi
Applicazione
- Rilevamento del micoplasma
Componenti
| Componenti n. | Nome | 40601ES10 (10 analisi) | 40601ES20 (20 analisi) |
| 40601-A | Miscela PCR GMyc-1st | 250 µl | 2×250 µL |
| 40601-B | Miscela di PCR GMyc-2nd | 250 µl | 2×250 µL |
| 40601-C | Modello di controllo positivo N | 20 µl | 20 µl |
[Note] 1. Quando non viene utilizzato per un lungo periodo, può essere conservato congelato a -85~-65℃.
2. La reazione PCR è estremamente sensibile.Per evitare falsi positivi, quando si aggiungono i campioni, alla fine viene aggiunto un controllo positivo.
Magazzinaggio
Questo prodotto può essere conservato a -25~-15℃ per 18 mesi. Se non viene utilizzato per un lungo periodo, si prega di tenerlo al riparo dalla luce.
Cifre
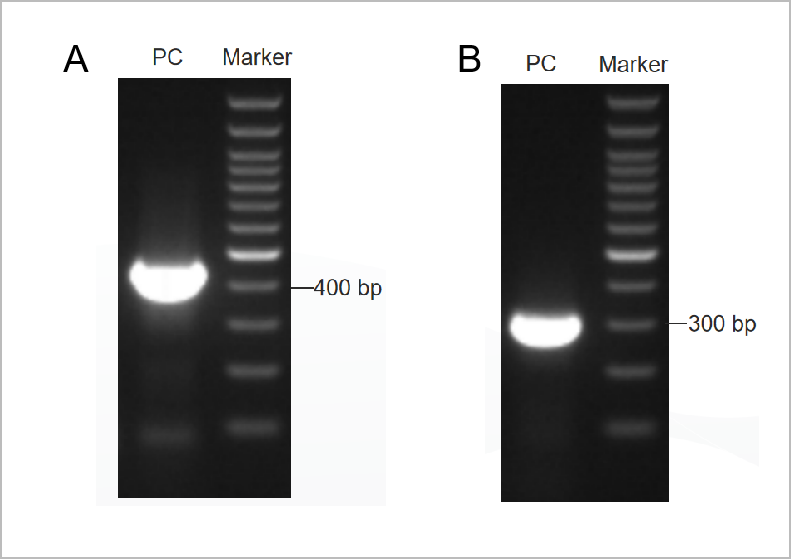
Figura 1. Il risultato dell'elettroforesi del PCR Mycoplama Test Kit.
Il primo elettroforetagramma circolare è stato mostrato nella Figura 1A e il secondo elettroforetagramma circolare è stato mostrato nella Figura 1B. (M: marcatore da 1 kb, 1-2: controlli positivi, 3: controllo negativo)
[1] Rao XS, Cong XX, Gao XK, et al. La fosforilazione mediata da AMPK migliora l'auto-inibizione di TBC1D17 per promuovere l'assorbimento del glucosio dipendente da Rab5. Cell Death Differ. 2021;28(12):3214-3234. doi:10.1038/s41418-021-00809-9(IF:15.828)
[2] Guo F, Li L, Li J, et al. Sequenziamento multi-omico a singola cellula di embrioni precoci di topo e cellule staminali embrionali. Cell Res. 2017;27(8):967-988. doi:10.1038/cr.2017.82(IF:15.606)
[3] Hao Y, He B, Wu L, et al. La traslocazione nucleare di p85β promuove la tumorigenesi del cancro mutante del dominio elicoidale PIK3CA. Nat Commun. 2022;13(1):1974. Pubblicato il 13 aprile 2022. doi:10.1038/s41467-022-29585-x(IF:14.919)
[4] Shu X, Liu M, Lu Z, et al. La mappatura dell'intero genoma rivela che la deossiuridina è arricchita nel DNA centromerico umano. Nat Chem Biol. 2018;14(7):680-687. doi:10.1038/s41589-018-0065-9(IF:13.843)
[5] Li X, Xiong X, Wang K, et al. La mappatura dell'intero trascrittoma rivela un metiloma N(1)-metiladenosina reversibile e dinamico. Nat Chem Biol. 2016;12(5):311-316. doi:10.1038/nchembio.2040(IF:12.709)
[6] Sun L, Yang X, Huang X, et al. La 2-idrossilazione degli acidi grassi reprime la tumorigenesi e la metastasi del colon-retto tramite l'asse trascrizionale YAP. Cancer Res. 2021;81(2):289-302. doi:10.1158/0008-5472.CAN-20-1517(IF:12.701)
[7] Sun Z, Zhang Z, Wang QQ, Liu JL. L'inattivazione combinata di CTPS1 e ATR è sinteticamente letale per le cellule tumorali che sovraesprimono MYC. Cancer Res. 2022;82(6):1013-1024. doi:10.1158/0008-5472.CAN-21-1707(IF:12.701)
[8] Song J, Zhuang Y, Zhu C, et al. Ruoli differenziali del PUS10 umano nell'elaborazione dei miRNA e nella pseudouridilazione del tRNA. Nat Chem Biol. 2020;16(2):160-169. doi:10.1038/s41589-019-0420-5(IF:12.154)
[9] He B, Pan H, Zheng F, et al. Il lungo RNA non codificante LINC00930 promuove la glicolisi tumorale mediata da PFKFB3 e la proliferazione cellulare nel carcinoma nasofaringeo. J Exp Clin Cancer Res. 2022;41(1):77. Pubblicato il 24 febbraio 2022. doi:10.1186/s13046-022-02282-9(IF:11.161)
[10] Tang B, Liu BH, Liu ZY, Luo MY, Shi XH, Pang DW. Punti quantici con un rivestimento zwitterionico anfifilico compatto. Interfacce ACS Appl Mater. 2022;14(24):28097-28104. doi:10.1021/acsami.2c04438(IF:9.229)
[11] Huang C, Zhang Z, Chen L, et al.L'acetilazione nei domini N- e C-terminali di Src regola ruoli distinti della tumorigenesi mediata da STAT3. Cancer Res. 2018;78(11):2825-2838. doi:10.1158/0008-5472.CAN-17-2314(IF:9.130)
[12] Wu X, Yu M, Zhang Z, et al. DDB2 regola la replicazione del DNA attraverso la degradazione indipendente da PCNA di CDT2. Cell Biosci. 2021;11(1):34. Pubblicato l'8 febbraio 2021. doi:10.1186/s13578-021-00540-5(IF:7.133)
[13] Wang J, Zhang Y, Liu X, Liu H. Ottimizzazione della terapia adattiva basata sulla raggiungibilità della sottopopolazione resistente al tumore. Tumori (Basilea). 2021;13(21):5262. Pubblicato il 20 ottobre 2021. doi:10.3390/cancers13215262(IF:6.639)
[14] Feng W, Liu R, Xie X, et al. La SUMOilazione dell'α-tubulina è una nuova modifica che regola la dinamica dei microtubuli. J Mol Cell Biol. 2021;13(2):91-103. doi:10.1093/jmcb/mjaa076(IF:6.216)
[15] Yu M, Hu X, Yan J, Wang Y, Lu F, Chang J. L'inibitore RIOK2 NSC139021 esercita effetti antitumorali sul glioblastoma inducendo l'arresto del ciclo cellulare e l'apoptosi mediati da Skp2. Biomedicines. 2021;9(9):1244. Pubblicato il 17 settembre 2021. doi:10.3390/biomedicines9091244(IF:6.081)
[16] Ren S, Cai Y, Hu S, et al. La berberina esercita attività antitumorale nel linfoma diffuso a grandi cellule B modulando l'asse c-myc/CD47. Biochem Pharmacol. 2021;188:114576. doi:10.1016/j.bcp.2021.114576(IF:5.858)
[17] Wen F, Sun X, Sun C, et al. TAGLN è sottoregolato dalla degradazione proteasomiale mediata da TRAF6 nelle cellule del cancro alla prostata. Mol Cancer Res. 2021;19(7):1113-1122. doi:10.1158/1541-7786.MCR-20-0513(IF:5.852)
[18] Tang B, Sun EZ, Zhang ZL, et al. Il dominio del colesterolo sequestrato dalla sfingomielina recluta la proteina legante la formina 17 per restringere le fossette rivestite di clatrina nell'ingresso del virus dell'influenza. J Virol. 2022;96(5):e0181321. doi:10.1128/JVI.01813-21(IF:5.103)
[19] Hu J, Ren W, Qiu W, et al. Generazione di una linea di cellule staminali pluripotenti indotte (XDCMHi001-A) da un paziente con spondilite anchilosante con mutazione JAK2. Stem Cell Res. 2020;45:101788. doi:10.1016/j.scr.2020.101788(IF:4.495)
[20] Xiao S, Yao X, Ye J, Tian X, Yin Z, Zhou L. La modifica epigenetica facilita l'espressione aberrante della prolina sintasi PYCR1 nel cancro gastrico [pubblicato online prima della stampa, 30 maggio 2022]. Biochim Biophys Acta Gene Regul Mech. 2022;1865(6):194829. doi:10.1016/j.bbagrm.2022.194829(IF:4.490)
[21] Wang J, Zhang Y, Liu X, Liu H. Il trattamento periodico fisso è efficace per il sistema tumorale senza informazioni complete?. Cancer Manag Res. 2021;13:8915-8928. Pubblicato il 30 novembre 2021. doi:10.2147/CMAR.S339787(IF:3.989)
[22] Yang X, Ren S, Rehman ZU, et al. Caratterizzazione molecolare, espressione e identificazione funzionale della chinasi legante TANK 1 (TBK1) della mucca (Bos taurus) e della capra (Capra hircus). Dev Comp Immunol. 2022;133:104444. doi:10.1016/j.dci.2022.104444(IF:3.636)
[23] Zheng D, Chang X, Liu Y, et al. Il 2-metossi-5((3,4,5-trimetosifenil)seleninil) fenolo inverte la migrazione cellulare e l'invasione indotte da EGF attraverso la down-regulation di MDM2 nelle linee cellulari del cancro al seno. Cancer Biol Ther. 2019;20(4):513-523. doi:10.1080/15384047.2018.1537578(IF:3.373)
[24] Xu F, Zhang S, Liu Z, et al. TEX9 ed eIF3b agiscono in sinergia funzionale per promuovere la progressione del carcinoma squamocellulare esofageo. BMC Cancer. 2019;19(1):875. Pubblicato il 3 settembre 2019. doi:10.1186/s12885-019-6071-9(IF:2.933)
[25] Pan H, Sun L, Wang W, et al. RNA non codificante lungo nel siero LOC553103 come biomarcatore diagnostico e prognostico non specifico per i tipi comuni di cancro umano. Clin Chim Acta. 2020;508:69-76. doi:10.1016/j.cca.2020.05.017(IF:2.615)
[26] Li N, Lin SM, Li Y, Sun J, Zhang L, Chen M.Una linea di cellule staminali pluripotenti indotte (GZHMCi004-A) derivata da un feto con mutazione eterozigote G380R nel gene FGFR3 che causa acondroplasia. Stem Cell Res. 2021;53:102322. doi:10.1016/j.scr.2021.102322(IF:2.020)
[27] Luo Q, Wei C, Long Y, et al. Generazione di una linea di cellule staminali embrionali umane knockout ELTD1 mediante il sistema iCRISPR/Cas9. Stem Cell Res. 2021;53:102350. doi:10.1016/j.scr.2021.102350(IF:2.020)
[28] Liu YQ, Ling TW, Wang HY, Yang YH, Song WJ, Wang TC. Generazione di una linea di cellule staminali pluripotenti indotte senza integrazione (LZUSHI001-A) da un paziente epilettico con mutazione DGKG. Stem Cell Res. 2022;61:102768. doi:10.1016/j.scr.2022.102768(IF:2.020)
[29] Chen M, Lin SM, Li N, Li Y, Li Y, Zhang L. Una linea di cellule staminali pluripotenti indotte (GZHMCi003-A) derivata da un feto con delezione eterozigote dell'esone 3 nel gene RUNX2 che causa displasia cleidocranica. Stem Cell Res. 2021;51:102166. doi:10.1016/j.scr.2021.102166(IF:2.020)
[30] Xu Y, Wang X, Qiu T, et al. Generazione di una linea di cellule staminali pluripotenti indotte (FDCHI007-A) derivata da un paziente con encefalopatia epilettica e dello sviluppo di tipo 31 portatrice di c.545C eterozigote > Una mutazione nel gene DNM1. Stem Cell Res. 2022;60:102709. doi:10.1016/j.scr.2022.102709(IF:2.020)
[31] Fan T, He J, Wang Y, Yu J, Sun W. Generazione di una linea di cellule staminali pluripotenti indotte (FDCHi006-A) da una bambina di 7 anni con pubertà precoce centrale. Stem Cell Res. 2021;56:102542. doi:10.1016/j.scr.2021.102542(IF:2.020)
[32] Gong X, Zheng Z, Yang T, Zheng H, Xiao X, Jia N. Generazione di una linea iPSC corretta dal gene isogenico (OGHFUi001-A-1) da un paziente con encefalopatia epilettica infantile precoce di tipo 1 (EIEE1) con una mutazione emizigote R330L nel gene ARX. Stem Cell Res. 2022;60:102693. doi:10.1016/j.scr.2022.102693(IF:2.020)
[33] Jia N, Gong X, Chen J, et al. Generazione di una linea di cellule staminali pluripotenti indotte (OGHFUi001-A) da un'encefalopatia epilettica infantile precoce di tipo 1 con mutazione ARX. Stem Cell Res. 2021;53:102367. doi:10.1016/j.scr.2021.102367(IF:2.020)
[34] Zhu W, Zhou Y, Wang Q, et al. Generazione di una linea di cellule staminali pluripotenti indotte umane (iPSC) da fibroblasti cutanei di un paziente portatore di una mutazione E363Q nel gene PSEN1. Stem Cell Res. 2022;61:102769. doi:10.1016/j.scr.2022.102769(IF:2.020)
[35] Luo F, Long K, Li X, et al. La carenza di LRRC8A attenua la necrosi indotta dall'ipossia nelle cellule 3T3-L1. Biosci Biotechnol Biochem. 2020;84(6):1139-1145. doi:10.1080/09168451.2020.1730689(IF:1.516)
Pagamento e sicurezza
Le informazioni di pagamento vengono elaborate in modo sicuro. Non archiviamo i dettagli della carta di credito né abbiamo accesso alle informazioni sulla tua carta di credito.
Indagine
Potrebbe piacerti anche
FAQ
Il prodotto è solo per scopi di ricerca e non è destinato all'uso terapeutico o diagnostico su esseri umani o animali. Prodotti e contenuti sono protetti da brevetti, marchi e copyright di proprietà di Yeasen Biotechnology. I simboli dei marchi indicano il paese di origine, non necessariamente la registrazione in tutte le regioni.
Alcune applicazioni potrebbero richiedere ulteriori diritti di proprietà intellettuale di terze parti.
Yeasen è un sostenitore della scienza etica, convinto che la nostra ricerca debba affrontare questioni critiche garantendo al contempo sicurezza e standard etici.

