Descrizione
Trasmettitore Hieff™ Liposomal Transfection Reagent è un versatile reagente di transfezione liposomiale, adatto per la transfezione di DNA, RNA e oligonucleotidi, con elevata efficienza di transfezione per la maggior parte delle cellule eucariotiche. La sua formula unica consente di aggiungerlo direttamente al mezzo e la presenza di siero non influisce sull'efficienza di transfezione, il che riduce i danni alle cellule causati dalla rimozione del siero. Non è necessario rimuovere il complesso acido nucleico-Hieff Trans™ o sostituirlo con un mezzo fresco dopo la transfezione e può anche essere rimosso dopo 4-6 ore.
Trasmettitore Hieff™ è fornito in forma liquida sterile. Solitamente, per la trasfezione di piastre a 24 pozzetti, circa 1,5 μL ogni volta, 1 mL di Hieff Trans™ può effettuare circa 660 trasfezioni; per una piastra a 6 pozzetti, circa 6 μL ogni volta, 1 mL di Hieff Trans™ può effettuare circa 660 trasfezioni. 160 trasfezioni.
Caratteristica
- Efficienza eccezionale: prestazioni di trasfezione superiori nell'ampia gamma di linee cellulari, inclusa la trasfezione transitoria e la trasfezione stabile
- Ampia adattabilità: eccellente efficienza di trasfezione in una varietà di linee cellulari e alti livelli di espressione proteica ricombinante
- Bassa tossicità: le attività delle diverse cellule sono pressoché inalterate dai reagenti di trasfezione di YEASEN
- Funzionamento semplice: efficacia comprovata in presenza di siero: elimina la necessità di cambiare terreno dopo la trasfezione
- Conveniente: effetto di trasfezione competitivo con prezzi più accessibili
Applicazione
- Trasfezione cellulare
Specificazione
| Modulo | Liquido |
| Compatibile con il siero | SÌ |
| Tipo di cellula | Linee cellulari stabilite |
| Tipo di campione | DNA plasmidico, siRNA sintetico |
| Tecnica di trasfezione | Trasfezione basata sui lipidi |
Componenti
| Componenti n. | Nome | 40802ES02 | 40802ES03 | 40802ES08 |
| 40802 | Traduzione di Hieff™ Reagente di trasfezione liposomiale | 0,5 ml | 1 ml | 5×1 ml. |
Spedizione e stoccaggio
Il prodotto viene spedito con ghiaccio e può essere conservato a 2-8ºC per un anno. Non congelare!
Cifre

Citato da: Communications Biology volume 5, Numero articolo: 1296 (2022)
- Dimostrazione dell'effetto di trasfezione
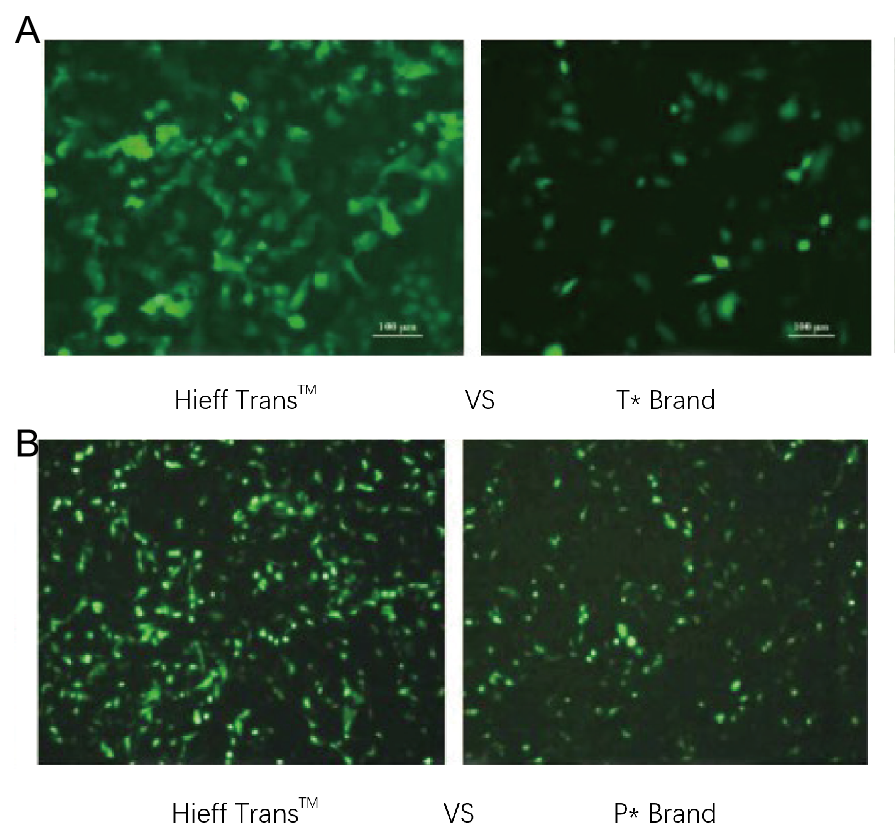
Figura 1. Il reagente di trasfezione liposomiale Hieff Trans™ supera le prestazioni del reagente di trasfezione del marchio concorrente (marchio T* nella Figura 1a, marchio P* nella Figura 1b).
Ogni reagente è stato utilizzato per trasfettare la linea cellulare target (linea cellulare Hela nella Figura 1a, linea cellulare DF-1 nella Figura 1b) in un formato a 96 pozzetti. L'espressione GFP è stata analizzata 48 ore dopo la trasfezione. Il reagente di trasfezione liposomiale Hieff Trans™ ha fornito un'efficienza di trasfezione GFP superiore rispetto ai prodotti di marca della concorrenza.
- Linee cellulari convalidate
| Prodotto Nome | Capo TransTMLiposomiale Trasfezione Reagente | |||||
| convalidato Cella Linee | 293T | Calu 1 | HEK293 | HO1980 | N2A | STUDENTI |
| 293F | CHO-K1 | Modello HEK293T | HUVEC | NCI-H1975 | U-87 | |
| 293 piedi | COS-7 | Ciao | Modello MCF10A | NIH-3T3 | Vero | |
| 3T3 | DF-1 | Epatite C | Il MCF-7 | Neuro-2a | WEHI | |
| A549 | H1299 | Epatite B | Codice MDA-MB-231 | PC-12 | Modello WRL-68 | |
| BV-2 | H520 | Epa1-6 | Codice articolo: MDA-MB-231-LM2-4175 | Raw264.7 | ||
| B50 | HaCaT | Epatite G2 | MDCK | SGC-7901 | ||
| C2C12 | HCT116 | HK2 | Meccanismo Energetico | T47D | … | |
Figura 2. Elenco delle linee cellulari trasfettate con successo con Hieff Trans™ Reagente di trasfezione liposomiale (in continuo aggiornamento).

Citato da "Optogenetic control of RNA function and metabolism using engineered light-switchable RNA-binding proteins. Nat Biotechnol. 2022 3 gennaio. doi: 10.1038/s41587-021-01112-1"

Citato da "Structural insight into BRCA1-BARD1 complex recruitment to corrupt chromatin. Mol Cell. 2021 Jul 1;81(13):2765-2777.e6. doi: 10.1016/j.molcel.2021.05.010."
Citato da "L'asse UBQLN2-HSP70 riduce gli aggregati di poli-Gly-Ala e allevia i difetti comportamentali nel modello animale C9ORF72. Neuron. 2021 16 giugno;109(12):1949-1962.e6. doi: 10.1016/j.neuron.2021.04.023."
Trasmettitore Hieff™ Reagente di trasfezione liposomiale Domande frequenti
(1) D: Il siero può essere presente durante la preparazione del complesso reagente di trasfezione dell'acido nucleico?
A: La presenza di siero influirà sulla formazione dei liposomi. Si raccomanda di utilizzare un mezzo privo di siero (solitamente un mezzo MEM) quando si preparano complessi di reagenti di trasfezione di acidi nucleici.
(2) D: Il reagente di trasfezione può essere congelato?
R: No. Questo reagente deve essere conservato a una temperatura compresa tra 2 e 8 ℃ e bisogna fare attenzione a non aprire ripetutamente il tappo per un lungo periodo, poiché l'apertura prolungata del tappo causerà l'ossidazione dei liposomi e influenzerà l'efficienza della trasfezione.
(3) D: A cosa dovrei prestare attenzione quando utilizzo il reagente di trasfezione dell'acido nucleico liposomiale Hieff Trans™?
A: 1) Durante l'operazione di trasfezione, è meglio che la confluenza cellulare raggiunga l'80%-95% e la densità di placcatura specifica venga determinata in base alla situazione delle cellule;
2) L'utilizzo di DNA ad elevata purezza aiuta a ottenere una maggiore efficienza di trasfezione;
3) Durante la preparazione dei complessi di trasfezione, è necessario diluire il DNA e i reagenti di trasfezione con un mezzo privo di siero;
4) Non è possibile aggiungere antibiotici al terreno durante la trasfezione;
5) La concentrazione del DNA e la quantità di reagente liposomiale cationico devono essere ottimizzate per il primo utilizzo per ottenere la massima efficienza di trasfezione. Il rapporto tra DNA e reagente di trasfezione è in genere si consiglia un rapporto di 1:2-1:3.
(4) D: È necessario terminarlo dopo la trasfezione?
R: Non è necessario. I complessi liposomiali sono stabili per 6 ore. Se il mezzo cellulare non viene cambiato prima della trasfezione, per garantire i nutrienti richiesti per la normale crescita cellulare, è necessario cambiare mezzo dopo 4-6 ore. Tuttavia, se il mezzo è stato cambiato prima della trasfezione, non è necessario cambiare mezzo dopo la trasfezione dei liposomi.
(5) D: A cosa dovrei prestare attenzione se voglio migliorare l'efficienza della trasfezione?
A: a: La densità delle cellule al momento della trasfezione è del 90%-95%.
b: Durante la trasfezione, utilizzare un mezzo MEM privo di siero per le diluizioni di acidi nucleici e liposomi.
c: Il mezzo può essere cambiato 4-6 ore dopo la trasfezione.
(6) D: È possibile eseguire la co-trasfezione di DNA e siRNA? Qual è l'effetto?
R: È possibile eseguire la co-transfezione, ma si raccomanda di eseguire la transfezione separata e la transfezione del DNA dovrebbe essere eseguita 6 ore dopo l'siRNA. Se si opera insieme, l'efficienza della transfezione dell'siRNA sarà peggiore.
(7) D: Il reagente di trasfezione può essere utilizzato per la trasfezione del confezionamento lentivirale?
R: Il confezionamento lentivirale è possibile, ma la sua efficienza non è necessariamente correlata all'efficienza della trasfezione, bensì anche alla selezione dei plasmidi di confezionamento e al rapporto tra i plasmidi.
(8) D: Il reagente di trasfezione dell'acido nucleico liposomiale Hieff Trans™ può essere utilizzato per la trasfezione di cellule in sospensione?
A: Hieff Trans™ Liposome Nucleic Acid Transfection Reagent può essere utilizzato per la trasfezione di cellule in sospensione, vedere Protocollo per i dettagli. Inoltre, abbiamo anche lanciato un reagente di trasfezione specificamente per cellule in sospensione (Cat. n. 40805, Liposome nucleic acid transfection reagent for suspension cells).
[1] Liu R, Yang J, Yao J, Zhao Z, He W, Su N, Zhang Z, Zhang C, Zhang Z, Cai H, Zhu L, Zhao Y, Quan S, Chen X, Yang Y. Controllo optogenetico della funzione e del metabolismo dell'RNA utilizzando proteine leganti l'RNA commutabili dalla luce ingegnerizzate. Nat Biotechnol. 3 gennaio 2022. doi: 10.1038/s41587-021-01112-1. Epub anticipato. PMID: 34980910. (IF:54.908)
[2] Zhou J, Chen P, Wang H, Liu H, Li Y, Zhang Y, Wu Y, Paek C, Sun Z, Lei J, Yin L. Varianti Cas12a progettate per un effetto off-target inferiore su tutto il genoma attraverso un rigoroso riconoscimento PAM. Mol Ther. 2022 5 gennaio;30(1):244-255. doi: 10.1016/j.ymthe.2021.10.010. Epub 20 ottobre 2021. PMID: 34687846; PMCID: PMC8753454. (IF:11.454)
[3] Chen S, Cao X, Zhang J, Wu W, Zhang B, Zhao F. circVAMP3 guida la separazione di fase di CAPRIN1 e inibisce il carcinoma epatocellulare sopprimendo la traduzione di c-Myc. Adv Sci (Weinh).2022 marzo;9(8):e2103817. doi: 10.1002/advs.202103817. Epub 2022 24 gennaio. PMID: 35072355; PMCID: PMC8922094. (IF:16.808)
[4] Zhang Y, Yu X, Sun R, Min J, Tang X, Lin Z, Xie S, Li X, Lu S, Tian Z, Gu C, Teng L, Yang Y. Il fattore di splicing arginina/serina-ricco 8 promuove la malignità del mieloma multiplo e la lesione ossea attraverso lo splicing alternativo di CACYBP e la comunicazione cellulare basata sugli esosomi. Clin Transl Med. 2022 febbraio;12(2):e684. doi: 10.1002/ctm2.684. PMID: 35184390. (IF:11.492)
[5] Tang X, Deng Z, Ding P, Qiang W, Lu Y, Gao S, Hu Y, Yang Y, Du J, Gu C. Una nuova proteina codificata da circHNRNPU promuove la progressione del mieloma multiplo regolando il microambiente del midollo osseo e lo splicing alternativo. J Exp Clin Cancer Res. 2022 marzo 8;41(1):85. doi: 10.1186/s13046-022-02276-7. PMID: 35260179. (IF:11.161)
[6] Hua Z, Wei R, Guo M, Lin Z, Yu X, Li X, Gu C, Yang Y. YTHDF2 promuove la proliferazione delle cellule del mieloma multiplo tramite l'asse STAT5A/MAP2K2/p-ERK. Oncogene. 2022 marzo;41(10):1482-1491. doi: 10.1038/s41388-022-02191-3. Epub 2022 gennaio 24. PMID: 35075244. (IF:9.867)
[7] Liang Y, Lu Q, Li W, Zhang D, Zhang F, Zou Q, Chen L, Tong Y, Liu M, Wang S, Li W, Ren X, Xu P, Yang Z, Dong S, Zhang B, Huang Y, Li D, Wang H, Yu W. Riattivazione del soppressore tumorale nel cancro al seno mediante commutazione dell'enhancer attraverso la rete NamiRNA. Nucleic Acids Res. 2021 7 settembre;49(15):8556-8572. doi: 10.1093/nar/gkab626. PMID: 34329471; PMCID: PMC8421228. (SE:16.9)
[8] Dai L, Dai Y, Han J, Huang Y, Wang L, Huang J, Zhou Z. Approfondimenti strutturali sul reclutamento del complesso BRCA1-BARD1 nella cromatina danneggiata. Mol Cell. 2021 1 luglio;81(13):2765-2777.e6. doi: 10.1016/j.molcel.2021.05.010. Epub 2021 7 giugno. PMID: 34102105. (IF:17.97)
[9] Zhang K, Wang A, Zhong K, Qi S, Wei C, Shu X, Tu WY, Xu W, Xia C, Xiao Y, Chen A, Bai L, Zhang J, Luo B, Wang W, Shen C. L'asse UBQLN2-HSP70 riduce gli aggregati di poli-Gly-Ala e allevia i difetti comportamentali nel modello animale C9ORF72. Neuron. 2021 16 giugno;109(12):1949-1962.e6. doi: 10.1016/j.neuron.2021.04.023. Epub 2021 14 maggio. PMID: 33991504. (IF:17.17)
[10] Liang Y, Lu Q, Li W, Zhang D, Zhang F, Zou Q, Chen L, Tong Y, Liu M, Wang S, Li W, Ren X, Xu P, Yang Z, Dong S, Zhang B, Huang Y, Li D, Wang H, Yu W. Riattivazione del soppressore tumorale nel cancro al seno mediante commutazione dell'enhancer attraverso la rete NamiRNA. Nucleic Acids Res. 2021 7 settembre;49(15):8556-8572. doi: 10.1093/nar/gkab626. PMID: 34329471; PMCID: PMC8421228. (IF:16.9)
[11] Li T, Chen X, Qian Y, Shao J, Li X, Liu S, Zhu L, Zhao Y, Ye H, Yang Y. Un dispositivo optogenetico sintetico basato su BRET per l'espressione pulsatile del transgene che consente l'omeostasi del glucosio nei topi. Nat Commun. 27 gennaio 2021;12(1):615. doi: 10.1038/s41467-021-20913-1. PMID: 33504786; PMCID: PMC7840992. (IF:14.92)
[12] Pan Y, He X, Li C, Li Y, Li W, Zhang H, Wang Y, Zhou G, Yang J, Li J, Qu J, Wang H, Gao Z, Shen Y, Li T, Hu H, Ma H. L'attività neuronale recluta l'asse CRTC1/CREB per guidare l'autofagia dipendente dalla trascrizione per il mantenimento dell'LTD in fase tardiva. Cell Rep. 2021 20 luglio;36(3):109398. doi: 10.1016/j.celrep.2021.109398. PMID: 34289350. (IF:9.42)
[13] Liu H, Xing R, Ou Z, Zhao J, Hong G, Zhao TJ, Han Y, Chen Y. Il recettore accoppiato alla proteina G GPR17 inibisce lo sviluppo del glioma aumentando la produzione di ROS mediata dal complesso repressivo Polycomb 1. Cell Death Dis. 2021 12 giugno;12(6):610. doi: 10.1038/s41419-021-03897-0. PMID: 34120140; PMCID: PMC8197764. (IF:8.463)
[14] Fan Y, Wang J, Jin W, Sun Y, Xu Y, Wang Y, Liang X, Su D. CircNR3C2 promuove l'effetto soppressivo del tumore mediato da HRD1 tramite spugnatura di miR-513a-3p nel cancro al seno triplo negativo. Mol Cancer. 2 febbraio 2021;20(1):25. doi: 10.1186/s12943-021-01321-x.ID PMI: 33530981; ID PMC: PMC7851937. (IF:27.403)
[15] Gu C, Wang Y, Zhang L, Qiao L, Sun S, Shao M, Tang X, Ding P, Tang C, Cao Y, Zhou Y, Guo M, Wei R, Li N, Xiao Y, Duan J, Yang Y. AHSA1 è un promettente bersaglio terapeutico per la proliferazione cellulare e la resistenza agli inibitori del proteasoma nel mieloma multiplo. J Exp Clin Cancer Res. 2022 6 gennaio;41(1):11. doi: 10.1186/s13046-021-02220-1. PMID: 34991674; PMCID: PMC8734095. (IF:11.161)
[16] Luo Q, Wu X, Zhao P, Nan Y, Chang W, Zhu X, Su D, Liu Z. OTUD1 attiva l'apoptosi indipendente dalla caspasi e dipendente dalla caspasi promuovendo la traslocazione nucleare dell'AIF e la degradazione di MCL1. Adv Sci (Weinh). 8 febbraio 2021; 8 (8): 2002874. doi: 10.1002/advs.202002874. PMID: 33898171; PMCID: PMC8061361. (IF: 15,84)
[17] Luo Q, Wu X, Nan Y, Chang W, Zhao P, Zhang Y, Su D, Liu Z. TRIM32/USP11 bilancia la stabilità di ARID1A e lo stato oncogenico/soppressivo del tumore del carcinoma a cellule squamose. Cell Rep. 2020 7 gennaio;30(1):98-111.e5. doi: 10.1016/j.celrep.2019.12.017. PMID: 31914402. (IF:9.42)
[18] Sun X, Peng X, Cao Y, Zhou Y, Sun Y. L'ADNP promuove la differenziazione neurale modulando la segnalazione Wnt/β-catenina. Nat Comune. 2020 12 giugno;11(1):2984. doi: 10.1038/s41467-020-16799-0. PMID: 32533114; ID PMC: PMC7293280. (IF:14.911)
[19] Yang X, Wang H, Xie E, Tang B, Mu Q, Song Z, Chen J, Wang F, Min J. Il ricollegamento della segnalazione ERBB3 ed ERK conferisce resistenza all'inibizione di FGFR1 nel cancro gastrointestinale che ospita una mutazione ERBB3-E928G. Protein Cell. 2020 dicembre; 11 (12): 915-920. doi: 10.1007/s13238-020-00749-z. PMID: 32632529; PMCID: PMC7719122. (IF: 14.872)
[20] Chen, T., Chen, Y., Chen, H. et al. Nanomacchina a doppio enzima che cammina senza vincoli sul DNA per l'imaging intracellulare di microRNA poco espressi. Nano Res. 12, 1055–1060 (2019). https://doi.org/10.1007/s12274-019-2344-5 (SE:8.21)
[21] Zhang X, Qi Z, Yin H, Yang G. L'interazione tra la segnalazione p53 e Ras controlla la resistenza al cisplatino tramite la regolazione dell'apoptosi e dell'autofagia mediata da HDAC4 e HIF-1α. Teranostics. 30 gennaio 2019;9(4):1096-1114. doi: 10.7150/thno.29673. PMID: 30867818; PMCID: PMC6401400. (IF:8.12)
[22] Zou Y, Wang A, Shi M, Chen X, Liu R, Li T, Zhang C, Zhang Z, Zhu L, Ju Z, Loscalzo J, Yang Y, Zhao Y. Analisi di paesaggi redox e dinamiche in cellule viventi e in vivo utilizzando sensori fluorescenti codificati geneticamente. Nat Protoc. 2018 ottobre;13(10):2362-2386. doi: 10.1038/s41596-018-0042-5. PMID: 30258175; PMCID: PMC6714056. (IF:13.49)
[23] Zhang K, Zhao X, Chen X, Wei Y, Du W, Wang Y, Liu L, Zhao W, Han Z, Kong D, Zhao Q, Guo Z, Han Z, Liu N, Ma F, Li Z. Effetti terapeutici migliorati degli esosomi derivati da cellule staminali mesenchimali con un idrogel iniettabile per il trattamento dell'ischemia degli arti posteriori. Interfacce ACS Appl Mater. 12 settembre 2018; 10 (36): 30081-30091. doi: 10.1021/acsami.8b08449. Epub 29 agosto 2018. PMID: 30118197. (IF: 8.09)
[24] Hao H, Hu S, Chen H, Bu D, Zhu L, Xu C, Chu F, Huo X, Tang Y, Sun X, Ding BS, Liu DP, Hu S, Wang M. La perdita di CXCR7 endoteliale compromette l'omeostasi vascolare e il rimodellamento cardiaco dopo infarto miocardico: implicazioni per la scoperta di farmaci cardiovascolari. Circolazione. 28 marzo 2017;135(13):1253-1264. doi: 10.1161/CIRCULATIONAHA.116.023027. Epub 2 febbraio 2017. PMID: 28154007. (IF:18.881)
Pagamento e sicurezza
Le informazioni di pagamento vengono elaborate in modo sicuro. Non archiviamo i dettagli della carta di credito né abbiamo accesso alle informazioni sulla tua carta di credito.
Indagine
Potrebbe piacerti anche
FAQ
Il prodotto è solo per scopi di ricerca e non è destinato all'uso terapeutico o diagnostico su esseri umani o animali. Prodotti e contenuti sono protetti da brevetti, marchi e copyright di proprietà di Yeasen Biotechnology. I simboli dei marchi indicano il paese di origine, non necessariamente la registrazione in tutte le regioni.
Alcune applicazioni potrebbero richiedere ulteriori diritti di proprietà intellettuale di terze parti.
Yeasen è un sostenitore della scienza etica, convinto che la nostra ricerca debba affrontare questioni critiche garantendo al contempo sicurezza e standard etici.