Descrizione
HEK293 Il kit di rilevamento dei residui di DNA delle cellule ospiti viene utilizzato per l'analisi quantitativa di HEK293 Il DNA della cellula ospite è presente in campioni intermedi, semilavorati e prodotti finiti di vari prodotti biologici.
Questo kit adotta la sonda fluorescente Taqman e il metodo della reazione a catena della polimerasi (PCR), che ha un limite minimo di rilevamento del livello fg e può rilevare in modo specifico e rapido il residuo HEK293 DNA cellulare. L'uso combinato dell'enzima UDG e dUTP può eliminare la contaminazione causata dai prodotti di amplificazione. Il kit deve essere utilizzato insieme al kit di preparazione del campione di DNA residuo (Cat# 18461ES).
Specifiche
| N. di cat. | 41331ES50/ 41331ES60 |
| Misurare | 50 tonnellate / 100 tonnellate |
Componenti
| Componenti n. | Nome | 41331ES50 | 41331ES60 |
| 41331-A | HEK293 Miscela qPCR (UDG più) | 0,75 ml. | 1.5 ml. |
| 41331-B | HEK293 Miscela Primer&Sonda | 250 microlitri | 500 microlitri |
| 41331-C | Tampone di diluizione del DNA | 2×1.8 ml. | 4×1.8 ml. |
| 41331-D | HEK293 Controllo del DNA (30 ng/μL) | 25 microlitri | 50 microlitri |
Magazzinaggio
Questo prodotto deve essere conservato a una temperatura compresa tra -25 e -15 °C per 2 anni.
Sia il 41331-A che il 41331-B devono essere conservati al riparo dalla luce.
Modelli di strumenti applicabili
Includono ma non sono limitati a:
Bio-Rad: Modulo ottico CFX96.
Termografo: ABI 7500; ABI Quant Studio 5.
Istruzioni
- HEK293Diluizione standard del DNA e preparazione della curva standard
L'HEK293 Il controllo del DNA è stato diluito in gradiente utilizzando il tampone di diluizione del DNA fornito nel kit*e la diluizione
la concentrazione è 300 pg/μL, 30 pg/μL, 3 pg/μL, 300 fg/μL, 30 fg/μL.
Vedi istruzioni dettagliate qui sotto:
- Scongelare il controllo HEK293DNA e il tampone di diluizione del DNA su ghiaccio. Dopo lo scongelamento completo, agitare delicatamente per mescolare e centrifugare a bassa velocità per 10 secondi.
- Estrarre sei provette pulite da 1,5 ml, contrassegnate con Std0, Std1, Std2, Std3, Std4, Std5.
- Aggiungere 90 μL di tampone di diluizione del DNA e 10 μL di controllo HEK293DNA alla provetta per microcentrifuga da 1,5 mL etichettata Std0, vale a dire diluire a 3 ng/μL. Mescolare e quindi centrifugare per 10 sec. Sottoconfezionare lo standard di DNA diluito e conservarlo a breve termine (non più di 3 mesi) a -25~-15℃**Si prega di evitare ripetuti cicli di congelamento e scongelamento.
- Aggiungere 90 μL di tampone di diluizione del DNA nelle altre provette***, quindi seguire la procedura sottostante per le diluizioni seriali****.
| Tubo | Rapporto di diluizione | Concentrazione standard |
| Standard1 | 10 μL Std0 + 90 μL Tampone di diluizione del DNA | 300 pg/μl |
| Standard2 | 10 μL Std1 + 90 μL tampone di diluizione del DNA | 30 pg/μl |
| Standard3 | 10 μL Std2 + 90 μL tampone di diluizione del DNA | 3 pg/μl |
| Standard4 | 10 μL Std3 + 90 μL tampone di diluizione del DNA | 300 fg/μl |
| Standard5 | 10 μL Std4 + 90 μL tampone di diluizione del DNA | 30 fg/μl |
Tabella 1 Diluizione del gradiente standard
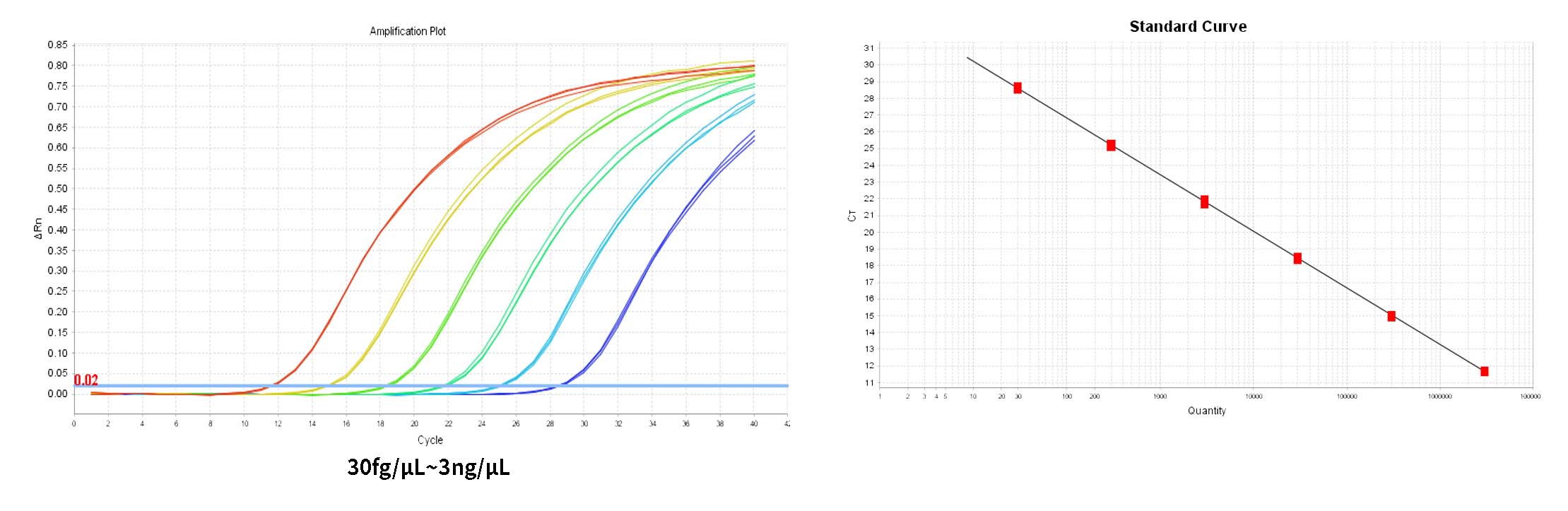
*Sono necessari tre pozzetti replicati per ogni concentrazione. L'intervallo di rilevamento è 30 fg/μL~300pg/μL e questo intervallo può essere ampliato.
**Per ridurre il numero di congelamenti-scongelamenti ripetuti ed evitare contaminazioni, si consiglia di conservare il controllo del DNA in aliquote a una temperatura compresa tra -25 e -15℃ per la prima volta.
***Una volta scongelato, il tampone di diluizione del DNA può essere conservato a 2-8°C per 7 giorni; se non utilizzato per un lungo periodo, conservarlo a una temperatura compresa tra -25 e -15°C.
****Assicurarsi che il modello sia completamente miscelato, agitare delicatamente la miscela per 15 secondi a 1 minuto per ogni diluizione del gradiente.
- Preparazione del controllo di recupero dell'estrazione (ERC)
Impostare la concentrazione di DNA HEK293 in ERC secondo necessità (il campione ERC è stato preparato con 30 pg di DNA HEK293 come esempio), come segue:
- Aggiungere 100 μL di campione di prova in una provetta pulita da 1,5 mL, quindi aggiungere 10 μL di standard di DNA HEK293 3pg/μL (Std3) e mescolare bene, contrassegnando come ERC.
- Eseguire l'estrazione del DNA dal campione di ERC insieme ai campioni di prova per preparare il campione di ERC purificato.
- Preparazione della soluzione di controllo negativo (NCS)
Impostato il controllo negativo nell'esperimento, i passaggi operativi specifici sono i seguenti:
1) Aggiungere 100 μL di matrice campione (o tampone di diluizione del DNA) in una provetta pulita da 1,5 mL, quindi contrassegnare come NCS.
2) Eseguire l'estrazione del DNA dal campione NCS insieme ai campioni di prova per preparare il campione NCS purificato.
- Preparazione senza controllo del modello (NTC)
Impostando il controllo "Nessun modello" nell'esperimento, i passaggi operativi specifici sono i seguenti:
1) L'NTC non richiede alcun pretrattamento del campione e può essere configurato nella fase di rilevamento del DNA residuo tramite qPCR.
2) Il campione NTC in ogni provetta o pozzetto è 20 μL di Mix (ovvero 15 μL di HEK293 qPCR Mix + 5 μL di HEK293 Primer&probe Mix) + 10 μL di DNA Dilution Buffer. Si consiglia di configurare tre pozzetti replicati.
- Sistema di reazione PCR
| Componente | Volume (μL) |
| HEK293 Miscela qPCR (più UDG)* | 15 |
| HEK293 Miscela Primer&Sonda | 5 |
| Modello di DNA | 10 |
| Volume totale** | 30 |
Tabella 2 Sistema di reazione
*Calcolare il volume totale della reazione PCR in base al numero di reazioni: qPCR Mix =(numero di reazioni+2) × (15+5) μL (incluse le perdite di due pozzetti di reazione). Si raccomandano più di tre repliche per ogni campione nell'esperimento.
**Dopo aver tappato la provetta o sigillato la piastra, centrifugare la provetta o la piastra di reazione a bassa velocità per 10 secondi. Dopo aver agitato e mescolato a sufficienza per 5 secondi, ripetere la centrifuga per raccogliere il liquido dal coperchio o dalla parete sul fondo. Evitare bolle durante l'operazione.
Per la configurazione consigliata della piastra, vedere la tabella seguente:
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| UN | NTC |
| TS1 - Italiano | TS1 - Italiano | TS1 - Italiano |
| Livello 1 | Livello 1 | Livello 1 |
|
|
|
| B | NTC |
| TS2 (Italiano) | TS2 (Italiano) | TS2 (Italiano) |
| Livello 2 | Livello 2 | Livello 2 |
|
|
|
| C | NTC |
| TS3 in Italiano | TS3 in Italiano | TS3 in Italiano |
| Livello 3 | Livello 3 | Livello 3 |
|
|
|
| D |
|
|
|
|
|
| Livello 4 | Livello 4 | Livello 4 |
|
|
|
| E | NCS |
| ERC 1 | ERC 1 | ERC 1 |
| Livello 5 | Livello 5 | Livello 5 |
|
|
|
| F | NCS |
| ERC 2 | ERC 2 | ERC 2 |
|
|
|
|
|
|
|
| G | NCS |
| ERC 3 | ERC 3 | ERC 3 |
|
|
|
|
|
|
|
| H |
|
|
|
|
|
|
|
|
|
|
|
|
Tabella 3 Scheda di riferimento del computer acceso
La disposizione delle piastre include: 5 Std (curva standard di 5 concentrazioni standard), 1 NTC (nessun controllo del modello), 1 NCS (soluzione di controllo negativo), 3 TS (campioni di prova), 3 ERC (controllo del recupero dell'estrazione).Tre pozzetti replicati per ogni campione.
- Impostare linee guida per uno strumento PCR(metodo in 2 fasi) (ad esempio strumento Thermo ABI 7500 qPCR, software versione 2.0)
Le seguenti istruzioni si applicano solo allo strumento Thermo ABI 7500 qPCR (versione software 2.0). Se si utilizza uno strumento diverso, fare riferimento alla guida dello strumento applicabile per le linee guida di configurazione.
1) Genera un nuovo esperimento, scegli il modello di quantificazione assoluta o definito dall'utente.
2) Crea 1 sonda di rilevamento, denominata "HEK293-DNA", seleziona il fluoroforo reporter come "FAM" e disattiva il fluoroforo come "Nessuno". La fluorescenza di riferimento è "ROX" (la fluorescenza di riferimento può essere basata sul modello dello strumento, ecc., seleziona se devi aggiungerla).
3) Nel riquadro 'Samples', aggiungi tutte le informazioni sui campioni a turno. Quindi seleziona i pozzetti, scegli il target e i campioni di conseguenza. Imposta l'attività di HEK293 Standard del DNA come standard e assegnare i valori 300000, 30000, 3000, 300, 30 (l'unità di concentrazione del DNA in ogni pozzetto è fg/μL) nella colonna Quantità e denominare i pozzetti Std 1, Standard 2, Standard 3, Standard 4, Standard 5, corrispondentemente. Imposta il compito di NTC come NTC. Imposta NCS, TS ed ERC come Sconosciuto, e nominarli in base al layout della Piastra di cui sopra corrispondentemente. Quindi fare clic su Avanti.
4) Impostare il programma di amplificazione: impostare il volume di reazione su 30 μL.
| Fase del ciclo | Temperatura (℃) | Tempo | Cicli |
| Digestione del prodotto di amplificazione | 37℃ | 5 minuti | 1 |
| Denaturazione iniziale | 95℃ | 10 minuti | 1 |
| Denaturazione | 95℃ | 15 secondi | 40 |
| Ricottura/Estensione (raccolta di fluorescenza) | 60℃ | 30 secondi |
Tabella 4 Procedura di amplificazione
- Analisi dei risultati qPCR
1) Il sistema fornirà automaticamente la soglia nel pannello Amplification Plot di Analysis. La soglia fornita dal sistema è talvolta troppo vicina alla linea di base, con conseguente grande differenza in Ct tra i pozzetti replicati. È possibile regolare manualmente la soglia in una posizione appropriata e fare clic su Analyze. Quindi è possibile inizialmente verificare se la curva di amplificazione è normale in Multicomponent Plot.
2) Nella scheda Analisi dei risultati, rivedere il grafico della curva standard. Verificare i valori per R2, Efficienza, Pendenza e intercetta Y. Per una curva standard normale, R²>0,99, 90% ≤ Eff% ≤ 110%, -3,6 ≤ Pendenza ≤-3,1.
3) Nel riquadro "Visualizza tabella pozzetti" in Analisi, le concentrazioni di ciascun campione sono mostrate in Quantità, l'unità è fg/μL e le unità possono essere convertite nel report del test.
4) Le impostazioni dei parametri dell'analisi dei risultati devono essere basate sul modello specifico e sulla versione del software utilizzata e possono generalmente essere interpretate automaticamente dallo strumento.
5) Calcolare il tasso di recupero dei picchi in base ai risultati del test del campione TS da misurare e al campione di recupero dei picchi ERC, il tasso di recupero dei picchi deve essere compreso tra il 50% e il 150%. Formula del misuratore del tasso di recupero dei picchi: Recupero (%) = {Sample spiked assay (eg.pg/μL) - Sample assay (eg.pg/μL)} x Volume di eluizione (μL) / Valore teorico della quantità di DNA aggiunta (ad esempio pg) x 100%。
6) Il valore Ct del controllo negativo NCS deve essere maggiore della media della concentrazione più bassa Ct dello standard.
- Il controllo NTC senza modello deve essere indeterminato o il valore Ct ≥3
Appunti
- Questo prodotto è destinato esclusivamente alla ricerca.
- Per la vostra sicurezza, operate indossando camici da laboratorio e guanti monouso.
3. Si prega di leggere attentamente questo manuale prima di utilizzare questo reagente e di standardizzare l'esperimento, inclusa la manipolazione del campione, la preparazione del sistema di reazione e l'aggiunta del campione.
4. Assicurarsi che ogni componente sia completamente agitato e centrifugato a bassa velocità prima dell'uso.
Pagamento e sicurezza
Le informazioni di pagamento vengono elaborate in modo sicuro. Non archiviamo i dettagli della carta di credito né abbiamo accesso alle informazioni sulla tua carta di credito.
Indagine
Potrebbe piacerti anche
FAQ
Il prodotto è solo per scopi di ricerca e non è destinato all'uso terapeutico o diagnostico su esseri umani o animali. Prodotti e contenuti sono protetti da brevetti, marchi e copyright di proprietà di Yeasen Biotechnology. I simboli dei marchi indicano il paese di origine, non necessariamente la registrazione in tutte le regioni.
Alcune applicazioni potrebbero richiedere ulteriori diritti di proprietà intellettuale di terze parti.
Yeasen è un sostenitore della scienza etica, convinto che la nostra ricerca debba affrontare questioni critiche garantendo al contempo sicurezza e standard etici.