DNase I en hun toepassingen in de biomedische wetenschap
Deoxyribonuclease I (DNase I) is een endonuclease, de toepassing ervan is niet alleen om RNA-integriteit te behouden, maar ook voor DNA-voetafdrukanalyse, generatie van willekeurige DNA-bibliotheken, vermindering van kleverigheid in cellysaten of proteïne-extracten, enz. Kortom, DNase I kan worden gebruikt in bijna elke toepassing die enzymsplitsingen van DNA vereist. Hieronder volgt een gedetailleerde introductie tot DNase I en de specifieke toepassing ervan.
1. Wat is DNase I?
2. DNase I voor de bereiding van DNA-vrije RNA-extractie
3. DNase I voor in vitro transcriptie om template DNA te verwijderen
4. DNase I voor rRNA-verwijdering
5. DNase I voor DNA-labeling
6. Andere toepassingen
7. DNase I productselectiegids
1. Wat is DNase I?
Desoxyribonuclease I (DNase I) is een niet-specifieke endonuclease die enkel- of dubbelstrengs DNA kan verteren, dat aanwezig is in verschillende weefsels en lichaamsvloeistoffen. Het kan fosfodiësterbindingen hydrolyseren om mono- en oligodeoxynucleotiden te produceren die een 5'-fosfaatgroep en een 3'-OH-groep bevatten. Het optimale werk-pH-bereik van DNase I is 7-8, de activiteit ervan is afhankelijk van Ca2+ en kan worden geactiveerd door divalente metaalionen zoals Mn2+, Mg2+, Zn2+, enz. In aanwezigheid van Mg2+ knipt DNase I willekeurig elke plaats van dubbelstrengs DNA; in aanwezigheid van Mn2+ kan DNase I dubbelstrengs DNA op dezelfde plaats knippen om een stomp uiteinde of een 1-2 nucleotide kleverige uiteinden overhang te vormen.
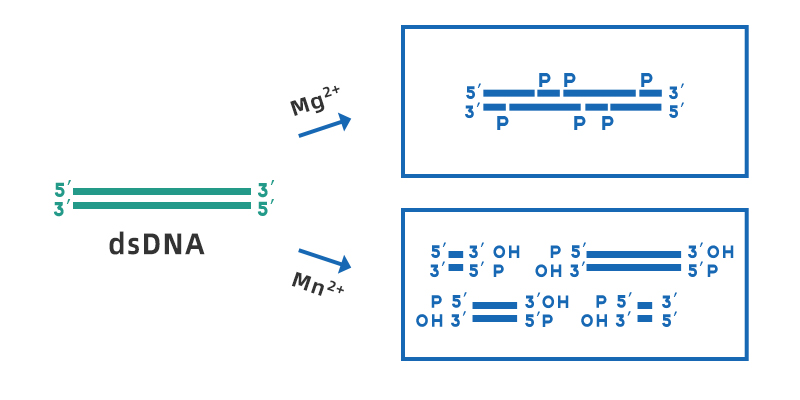
Figuur 1. Schematisch diagram van de splitsing van dsDNA door DNase I in aanwezigheid van Mg2+ en Mn2+.
Hoewel DNase I-splitsingen over het algemeen worden beschouwd als niet-specifieke splitsingen, is het waarschijnlijker dat DNase I inwerkt op bepaalde sequentiefragmenten, zoals de minor groove-regio, en is het gevoeliger voor splitsingen van purine-pyrimidinesequenties. Wanneer DNase I echter inwerkt op heterogeen dsDNA, worden alle vier de basen gesplitst en zal het effect op een specifieke base niet meer dan 3 keer groter zijn dan dat van andere basen.
2. DNase I voor de bereiding van DNA-vrije RNA-extractie
Bij biologische experimenten is de eerste stap het voorbereiden van nucleïnezuur om verschillende functies van RNA te bestuderen. Omdat DNA en RNA echter vaak samen worden vrijgegeven tijdens het cellyseproces, kan de interferentie in de twee niet worden vermeden, ongeacht welke extractieoplossing wordt gebruikt. Daarom moeten specifieke enzymen worden gebruikt om de interferentie te verwijderen. Voor hoogwaardige RNA-extractie wordt DNase I gebruikt om resterend DNA uit het monster te verwijderen.
DNase I kan dubbelstrengs en enkelstrengs DNA afbreken tot oligonucleotiden en enkelvoudige nucleotiden, en het DNA in het RNA-preparaatproduct kan effectief worden afgebroken. DNase I wordt vervolgens geïnactiveerd door verhitting met een stopbuffer. Tijdens het verhittingsproces kan de haarspeldstructuur van het RNA-molecuul worden geopend, wat de directe toegang van het RNA tot het reverse transcriptieproces vergemakkelijkt.
De kwaliteit van RNA zal de experimentele gegevens in grote mate direct beïnvloeden. Over het algemeen kunnen gDNA-residuen niet volledig worden vermeden tijdens RNA-extractie, daarom wordt over het algemeen aanbevolen om RNA-monsters te behandelen met DNase I om resterend gDNA te verteren voordat u downstream-toepassingen uitdaagt (bijv. mRNA-expressieanalyse, transcriptoomanalyse, enz.). De stap van het verteren van gDNA kan worden uitgevoerd tijdens RNA-extractie, na RNA-extractie of vóór RNA-reverse transcriptie.Volgens de productpositionering worden de producten geleverd door
Tabel 1: Lijst met producten gerelateerd aan DNA-verwijdering van RNA-extractie of vóór reverse transcriptie
| Productpositionering | Productnaam | Kat # |
| RNA-extractie | TRIeasy™ Totaal RNA-extractiereagens [navragen] | 10606ES |
| 19221ES | ||
| MolPure™ Plant Plus RNA-kit [navragen] | 19292ES | |
| MolPure™ Virale DNA/RNA-kit [navragen] | 19321ES | |
| gDNA-verwijdering | 10325ES | |
| Omgekeerde transcriptie | Hifair™Ⅲ1st Strand cDNA Synthesis SuperMix voor qPCR (gDNA digester plus) | 11141ES |
| qPCR | 11184ES |
3. DNase I voor in vitro transcriptie om template DNA te verwijderen
In vitro transcriptie (IVT) gebruikt voornamelijk DNA als template, plus bijbehorende substraten en buffers om RNA te verkrijgen via in vitro transcriptie. Bij in vitro transcriptie-experimenten worden RNA-polymerasen zoals T7, T3 en SP6 vaak gebruikt voor RNA-synthese. Het gesynthetiseerde RNA kan DNA-residuen bevatten. Het verwijderen van de DNA-residuen is gunstig voor de ontwikkeling van downstream experimenten. Bijvoorbeeld, in de mRNA-vaccinontwikkelingsfase is het verwijderen van residuen een cruciale stap, wat de moeilijkheid van downstream zuivering kan verminderen en de zuiverheid van het product kan verhogen. De DNA-template wordt doorgaans verwijderd met behulp van Recombinant DNase I (RNase-vrij).Volgens het mRNA-syntheseproces worden de producten geleverd door
| mRNA-syntheseproces | Productnaam | Kat# |
| Sjabloonvoorbereiding | Hieff Canace™ Plus Hoogwaardige DNA-polymerase [navragen] | 10148ES |
| 10922ES | ||
| 10125ES | ||
| FuniCut™BsaI [navragen] | 15005ES | |
| FuniCut™ XbaI [navragen] | 15033ES | |
| BspQI[vragen] [navragen] | 16215ES | |
| In vitro transcriptie | 10623ES | |
| 10624ES | ||
| T7 RNA-polymerase (50 U/μL)[navragen] | 10618ES | |
| 10133ES | ||
| 10620ES | ||
| 10621ES | ||
| Verwijder sjabloon-DNA | 10611ES | |
| mRNA-modificatie | 10614ES | |
| 10612ES | ||
| 10132ES | ||
| 10619ES | ||
| mRNA-zuivering | 12602ES |
4. DNase I voor rRNA-verwijdering
In vivo is rRNA zeer overvloedig en zeer conservatief, wat van weinig belang is voor het verkrijgen van biologische informatie. Daarom wordt rRNA vaak als eerste verwijderd bij de constructie en sequentiebepaling van een RNA-bibliotheek.Momenteel is de verwijderingsmethode van rRNA voornamelijk RNase H-digestie. De belangrijkste stappen van enzymatisch-gebaseerde rRNA-uitputting worden weergegeven in figuur 2: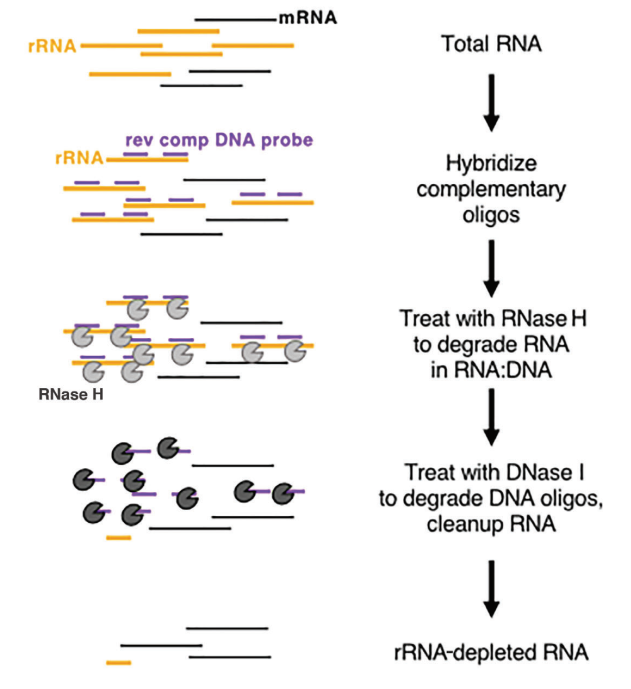
Figuur 2: Schematisch diagram van het principe van enzymatisch-gebaseerde rRNA-uitputting (Baldwin, A. et al. 2021, Current Protocols)
Eerst totale RNA extraheren, dan enkelstrengs DNA-sonde hybridiseren met rRNA, rRNA-specifieke enkelstrengs DNA-sonde ontwerpen en synthetiseren, dan RNase H gebruiken om het gehybridiseerde rRNA af te breken, en DNase I gebruiken om de DNA-sonde af te breken. Ten slotte het niet-rRNA RNA-sjabloon achterlaten. De producten gerelateerd aan rRNA-verwijdering geleverd door
| mRNA-syntheseproces | Productnaam | Kat# |
| Mens/Muis/Rat rRNA-uitputting | Hoog NGS™ MaxUp rRNA-uitputtingskit (mens/muis/rat) MaxUp [navragen] | 12253ES |
| Uitputting van rRNA in planten | 12254ES | |
| Verwijdering van ribosomaal RNA en 45S ITS/ETS-regio's uit menselijk totaal RNA | 12257ES | |
| Afbraak van rRNA | 12906ES | |
| DNA-sonde-afbraak | 10325ES |
5.DNase I voor DNA-labeling
De nick-translatie is een van de meest gebruikte deoxyribonucleïnezuur-probe-labelingsmethoden in het laboratorium. Deze methode maakt gebruik van verschillende enzymatische activiteiten van DNA-polymerase I om gelabelde deoxyribonucleoside-trifosfaten op te nemen in nieuw gesynthetiseerde DNA-ketens. Zo worden uniform gelabelde DNA-probes voor hoge specifieke activiteit gesynthetiseerd. De kenmerken van nick-translatie zijn snelle, eenvoudige, doelbewuste, hoge specificiteit en uniform gelabelde probes, die geschikt zijn voor langer dubbelstrengs DNA.De methode wordt gerealiseerd door de gecombineerde werking van DNase I en E. coli DNA Polymerase I. De belangrijkste stappen van DNA-labeling door nick-translatie worden weergegeven in figuur 3:
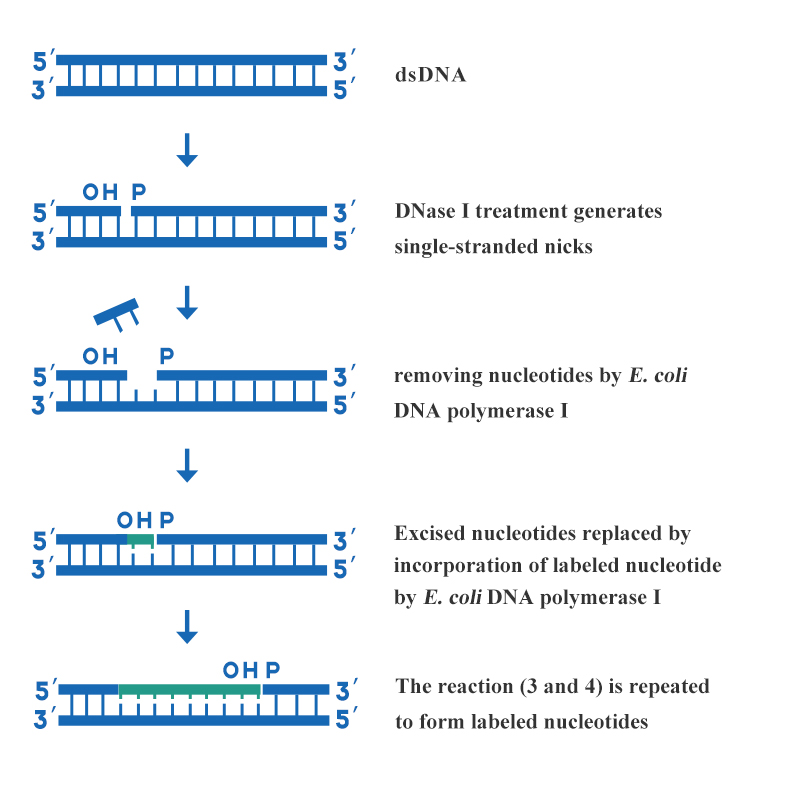
Figuur 3: Schematisch diagram van DNA-labeling door nick-translatie
Een geschikte concentratie DNase I wordt gebruikt om meerdere enkelstrengs gaten te creëren op elke streng van het dubbelstrengs DNA dat gelabeld moet worden, en de 3' hydroxyl terminus wordt gevormd bij de kloof. Gebruik de 5'→3' exonuclease activiteit van E. coli DNA Polymerase I om een nucleotide te knippen van het 5' uiteinde van de nick, en tegelijkertijd de 5'→3' polymerase activiteit van E. coli DNA Polymerase I introduceert een nucleotide gelabeld met het 3' uiteinde van de kloof om de kloof te repareren. Terwijl de kloof langs de DNA streng beweegt, worden de gelabelde nucleotiden opgenomen in de nieuw gesynthetiseerde streng.De producten met betrekking tot DNA-labeling die worden geleverd door
| Productpositionering | Productnaam | Kat# |
| Normaal | Deoxyribonuclease I (DNase I) uit runderpancreas [navragen] | 10607ES/10608ES |
| RNase vrij | 10325ES | |
| E. coli bron | 12903ES |
6. Andere toepassingen
De bovenstaande zijn enkele veelgebruikte toepassingen. Meer toepassingen van DNase I zijn onder andere de volgende, zoals DNase I footprinting assay en DNase I hypersensitive sites. DNase I footprinting assay is een detectiemethode die de bindingssites van DNA-bindende eiwitten op DNA nauwkeurig kan identificeren. Wanneer een eiwit zich bindt aan een DNA-fragment, kan het de bindingssite beschermen tegen beschadiging door DNase I en blijven de DNA-fragmenten achter na enzymdigestie ("footprint"), en kan de sequentie ervan worden bepaald. In de gelafbeelding zijn er geen banden waar het DNA zich bindt aan het eiwit. Klik op de link om meer te lezen.DNase I overgevoelige sites verwijzen naar splitsingen op een klein aantal specifieke sites wanneer chromatine wordt behandeld met lage DNase I en deze specifieke sites worden DNase I overgevoelige sites genoemd. Het principe is dat wanneer een gen zich in een transcriptioneel actieve staat bevindt, het chromatine dat het gen bevat aanzienlijk gevoeliger is voor DNase-afbraak dan de inactieve regio. Klik op de link om meer te lezen.
7. DNase I productselectiegids
| Productnaam (Cat#) | Productpositionering | Aanbevolen toepassingen |
| DNase I uit runderpancreas (CAT#10607,10608)[navragen] | RNase verwijderd, niet gedetecteerd | Wordt vooral gebruikt in eiwitonderzoek: het verwijderen van DNA uit eiwitpreparaten. |
| Recombinant DNase I (RNase-vrij)(CAT-nr. 10325) | RNase-vrij, voor onderzoek | Ideaal voor uiteenlopende toepassingen: het verwijderen van DNA uit RNA- en eiwitpreparaten, zoals RNase-gevoelige cDNA-bibliotheken, of het voorbereiden van monsters voor RT-PCR-experimenten. |
| UCF.ME™Deoxyribonuclease I (DNase I) GMP-klasse(CAT-nr. 10611) | RNase-vrij, GMP farmaceutische kwaliteit. | Ideaal voor uiteenlopende toepassingen: het verwijderen van DNA uit RNA- en eiwitpreparaten, zoals RNase-gevoelige cDNA-bibliotheken, of het voorbereiden van monsters voor RT-PCR-experimenten. |
Over het lezen:
GMP-kwaliteit reagentia voor mRNA in vitro synthese
De principes van DNase I-footprinting en de biomedische toepassingen ervan
Referenties
1. Baldwin A, Morris AR, Mukherjee N. Een eenvoudige, kosteneffectieve en schaalbare methode om menselijk ribosomaal RNA uit te putten voor RNA-seq[J]. Current Protocols, 2021.
2. Song C, Zhang S, Huang H. Kiezen van een geschikte methode voor de identificatie van replicatieoorsprongen in microbiële genomen [J]. Frontiers in Microbiology, 2015, 6:1049.
