Wat moet je doen als de qPCR-resultaten vreemd zijn?
qPCR-experimenten kunnen gemakkelijk en soms moeilijk aanvoelen. Nadat het primerontwerp is voltooid, worden qPCR-monsters stap voor stap toegevoegd, net als PCR, en kan het experiment op de machine worden voltooid. Er zijn verschillende veelvoorkomende problemen bij qPCR-experimenten met behulp van de kleurstofmethode en gezien de mogelijke oorzaken en oplossingen hopen we dat u ze kunt gebruiken om de problemen in qPCR-experimenten op te lossen.
1. Wat is qPCR?
2. Abnormale qPCR-amplificatiecurve
3. Abnormale qPCR-smeltcurve
4. Bestelgegevens
5. Gerelateerde producten
6. Over het lezen
1. Wat is qPCR?
Sinds de polymerasekettingreactietechniek (PCR) is uitgevonden, is PCR waarschijnlijk de meest gebruikte techniek in de moleculaire biologie vanwege zijn eenvoud, goedkoopheid, betrouwbaarheid, snelheid en hoge gevoeligheid. qPCR is een technologie die is ontwikkeld door PCR-technologie. Tijdens het DNA-amplificatieproces heeft de methode voor het detecteren van de totale hoeveelheid producten na elke PCR-cyclus met fluorescerende kleurstoffen niet alleen de snelheid en gevoeligheid van PCR, maar heeft ook meer specificiteit en hoge, realtime monitoring, herhaalbare en nauwkeurige kwantificering en andere voordelen. qPCR is een technologie die gebruikmaakt van een kwantitatief realtime PCR-amplificatieapparaat, namelijk een qPCR-instrument, om nucleïnezuuramplificatieproducten in realtime te monitoren tijdens het PCR-proces. qPCR realiseert de kwantitatieve analyse van de starttemplate door de analyse van de Ct-waarde en standaardcurve. In 1992 stelde de Japanner Higuchi voor het eerst "realtime fluorescerende kwantitatieve PCR-technologie" voor. In 1996 lanceerde de American Biological Company 's werelds eerste fluorescerende kwantitatieve PCR-instrument, dat bestaat uit een PCR-amplificatie thermisch cyclussysteem, fluorescentiedetectie optisch systeem en computer- en applicatiesoftware. Het kan nucleïnezuuramplificatieproducten in realtime bewaken via fluorescerende kleurstoffen of fluorescerende probes. , door de wiskundige functierelatie, gecombineerd met de software voor resultaatanalyse, wordt de berekening van de initiële templatehoeveelheid van het te testen monster gerealiseerd. Daarom is realtime fluorescerende kwantitatieve PCR-technologie op grote schaal gebruikt. Fluorescerende labelmethoden voor qPCR worden onderverdeeld in fluorescerende kleurstofmozaïekmethode gebaseerd op SYBR Green I-kleurstofmethode, fluorescerende probemethode gebaseerd op Taqman-probemethode (Cycling Probe, Molecular Bracon, enz.), quencher-kleurstofprimermethode.
qPCR-experimenten kunnen gemakkelijk en soms moeilijk aanvoelen. Nadat het primerontwerp is voltooid, worden qPCR-monsters stapsgewijs toegevoegd, net als PCR, en kan het experiment op de machine worden voltooid. Er zijn echter veel kleine details die aandacht behoeven, zoals geen vloeistof die aan de pijpwand hangt, geen bellen in de pijp en de noodzaak om het directe contact tussen de pijpwand, pijpafdekking, enz. te minimaliseren, om experimentele fouten of vreemde experimentele resultaten te verminderen. Xiaoyi heeft verschillende veelvoorkomende problemen in qPCR-experimenten verzameld door middel van de kleurstofmethode en de mogelijke oorzaken en oplossingen gegeven, waarvan we hopen dat u ze kunt gebruiken om de problemen in qPCR-experimenten op te lossen.
2. Abnormale qPCR-amplificatiecurve
2.1 Afwijkingen in de basislijnfase van de logaritmische grafiek van de versterkingscurve
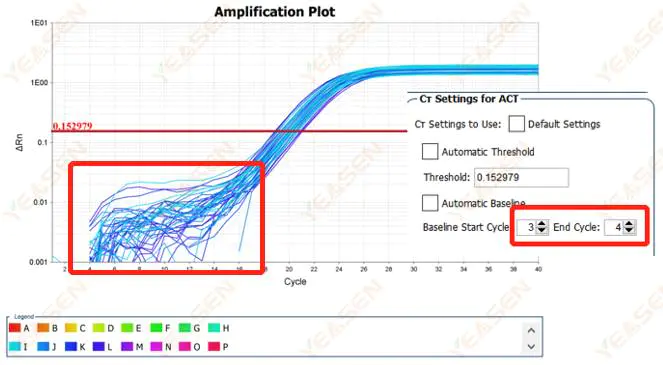
Figuur 1. Amplificatieplot
Mogelijke oorzaken:
Onjuiste basislijninstelling.
Oplossingen:
Het wordt aanbevolen om de eindpuntwaarde van de basislijn te verhogen.Het voorbeelddiagram van de aanpassing ziet er als volgt uit.

Figuur 2. Amplificatieplot
2.2 Versterkingscurve logplot curve segmentatie
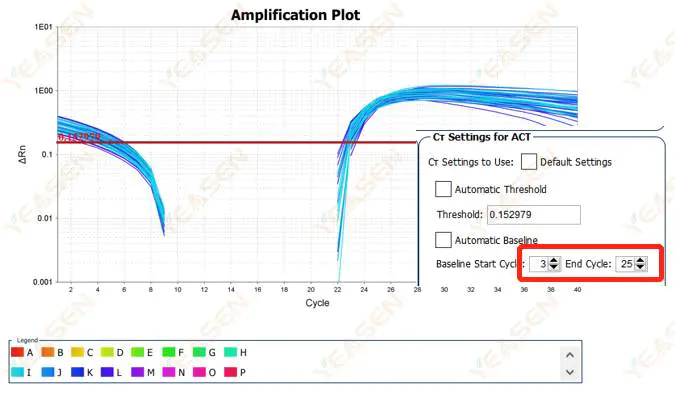
Figuur 3. Amplificatieplot
Mogelijke oorzaken:
De basislijn is te hoog ingesteld.
Oplossingen:
Het wordt aanbevolen om de eindpuntwaarde van de basislijn te verlagen. Het aanpassingsvoorbeelddiagram is als volgt.
2.3 Ongelijkmatige lineariteitsplot van de versterkingscurve
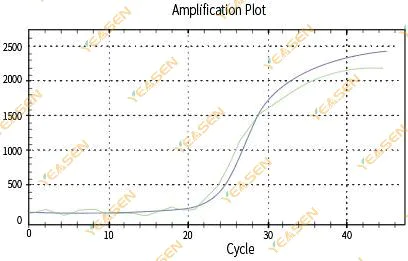
Figuur 4. Amplificatieplot
Mogelijke oorzaken:
1) De PCR-reageerbuis was niet goed afgesloten en de reactievloeistof lekte.
2) PCR-reactieoplossing met hangende wand.
3) Het instrument is niet gekalibreerd (inclusief automatische kalibratie of ROX-kalibratie).
4) Het systeem bevat veel remmers, wat leidt tot onstabiele fluorescentie.
5)Overmatig gebruik van het instrument resulteert in een onstabiele fluorescentieverzameling.
Oplossingen:
1) Druk de buisafdekking goed aan.
2) Meng het reagens grondig en doe het voorzichtig in het kwantitatieve instrument nadat het volledig is gecentrifugeerd.
3) Kalibreer het instrument.
4) Verbeter de zuiverheid van RNA en selecteer geschikte reagentia voor reverse transcriptie.
2.4 Versterkte kromme lineaire grafiek zigzag op een plateau
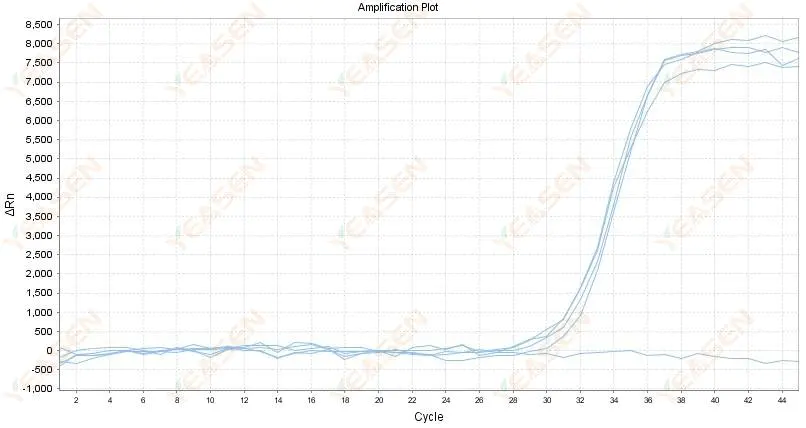
Figuur 5. Amplificatieplot
Mogelijke oorzaken:
1) De zuiverheid van RNA is slecht en er zitten veel onzuiverheden in.
2) Het instrument wordt te lang gebruikt.
Oplossingen:
1) Hoogwaardig RNA opnieuw extraheren.
2) Verdun het RNA-sjabloon om de concentratie onzuiverheden te verminderen.
3) Kalibreer het instrument.
2.5 Lineaire grafiek van de amplificatiecurve bereikt geen plateau
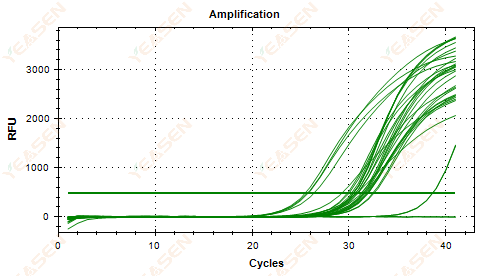
Figuur 6. Amplificatieplot
Mogelijke oorzaken:
1) Lage templateconcentratie (Ct-waarde rond de 35).
2) Te weinig versterkingscycli.
3) Lage efficiëntie van reagensversterking (kleine Ct, maar ook niet in staat om plateau te bereiken).
Oplossingen:
1) Verhoog de concentratie van het sjabloon.
2) Verhoog het aantal cycli.
3) Toenemende Mg2+ concentratie.
2.6 Versterkingscurve lineaire plot plateau doorzakken
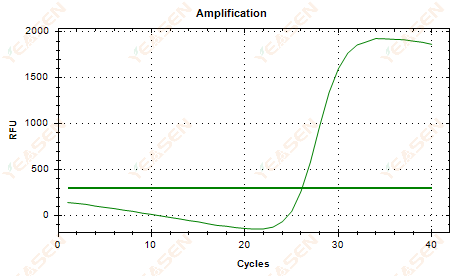
Figuur 7. Amplificatieplot
Mogelijke oorzaken:
1) Aanwezigheid van degradatie (degradatie van amplificatieproduct, SYBR-degradatie).
2) De dop van het buisje is niet goed afgesloten en het reagens is vervluchtigd.
3) De cDNA-concentratie is te hoog (als de Ct-waarde te laag is, wordt de fluorescentiedrempel verhoogd en is de uitzakking ernstiger).
4) Er komen luchtbelletjes in de buis, daarna verdwijnen ze.
Oplossingen:
1) Verbeter de zuiverheid van het systeem.
2) Verminder de hoeveelheid cDNA (verdun de template).
3) Verlaag de eindpuntwaarde van de basislijn.
2.7 Grote waarde van Ct in de amplificatiecurve
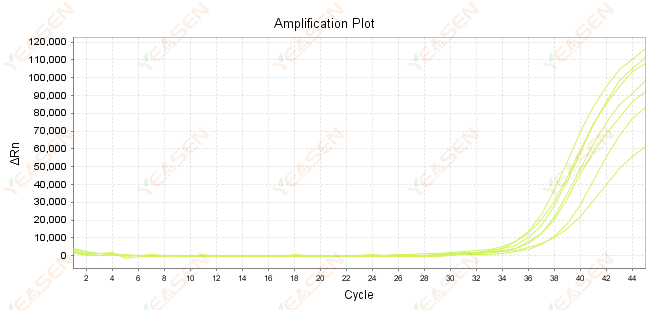
Figuur 8. Amplificatieplot
Mogelijke oorzaken:
1) Weinig sjablonen.
2) Lage versterkingsefficiëntie.
3) PCR-fragment is te lang.
4) Aanwezigheid van remmers in het reactiesysteem.
Oplossingen:
1) Verkort de verdunningstijden of vergroot de hoeveelheid template zodat de Ct-waarde zoveel mogelijk tussen de 15 en 30 ligt.
2) Optimaliseer de reactieomstandigheden, probeer een amplificatieprocedure in drie stappen of herontwerp primers.
3) De lengte van het PCR-product is ontworpen om binnen 100-150 bp te liggen. Het wordt niet aanbevolen om de lengte te overschrijden tot 300 bp.
4) Reproductie van sjablonen met een hogere zuiverheid.
2.8 Slechte herhaalbaarheid van amplificatiecurven
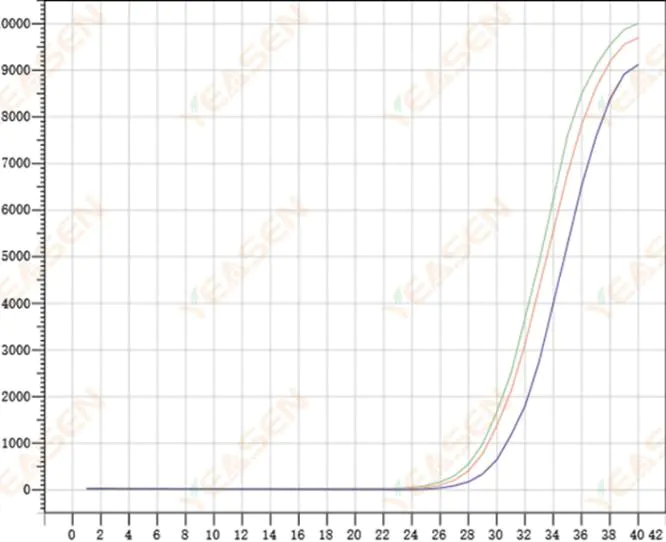
Figuur 9. Amplificatieplot
Mogelijke oorzaken:
1) Grote spikefouten.
2) Reagentia en systemen zijn niet goed gemengd.
3) Laag aantal kopieën van cDNA.
4) Er is geen Rox-kalibratie gebruikt.
Oplossingen:
1) Pipetten kalibreren.
2) Grondige menging van het reactiesysteem.
3) Een lage templateconcentratie, slechte herhaalbaarheid, 4-6 samengestelde gaten en 1-2 afwijkingen van grotere waarden kunnen op de juiste manier worden verworpen.
4) Het is het beste om Rox-kalibratie te gebruiken. Als het gebruikte reagens geen Rox bevat, moet de referentiekleurstof worden geselecteerd als Geen.
2.9 Willekeurige versterkingscurven
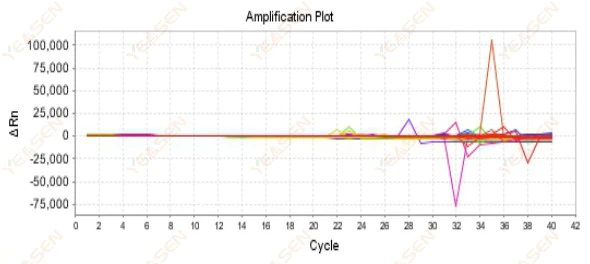
Figuur 10. Amplificatieplot
Mogelijke oorzaken:
De mismatch tussen de Rox-concentratie en het model.
Oplossingen:
Wijzig de referentiekleurstofinstelling op het instrument van ROX naar GEEN en kijk of de versterkingscurve weer normaal wordt.
2.10 NTC-piek
2.10.1 Ct>35, smeltcurve Tm-waarde <80℃
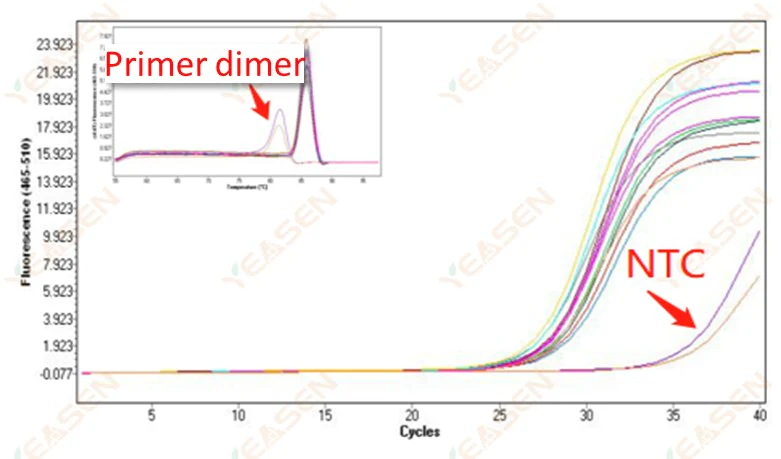
Figuur 11. Amplificatieplot
Mogelijke oorzaken:
Resultaten van primerdimerisatie.
Oplossingen:
Geoptimaliseerde primers.
2.10.2 Ct-waarde <35, NTC-smeltcurve en gen-smeltcurve piekvorm overlappen
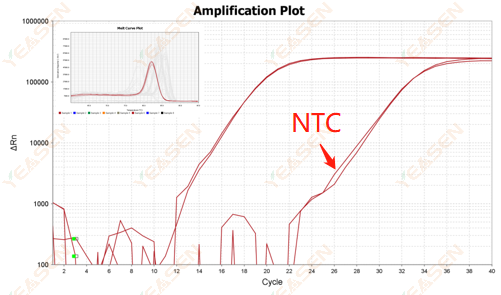
Figuur 12. Amplificatieplot
Mogelijke oorzaken:
Verontreiniging van het reactiesysteem.
Oplossingen:
Controleer de bronnen van vervuiling één voor één.
3. Abnormale qPCR-smeltcurve
3.1 Enkelvoudige pieksmeltcurve maar niet scherp
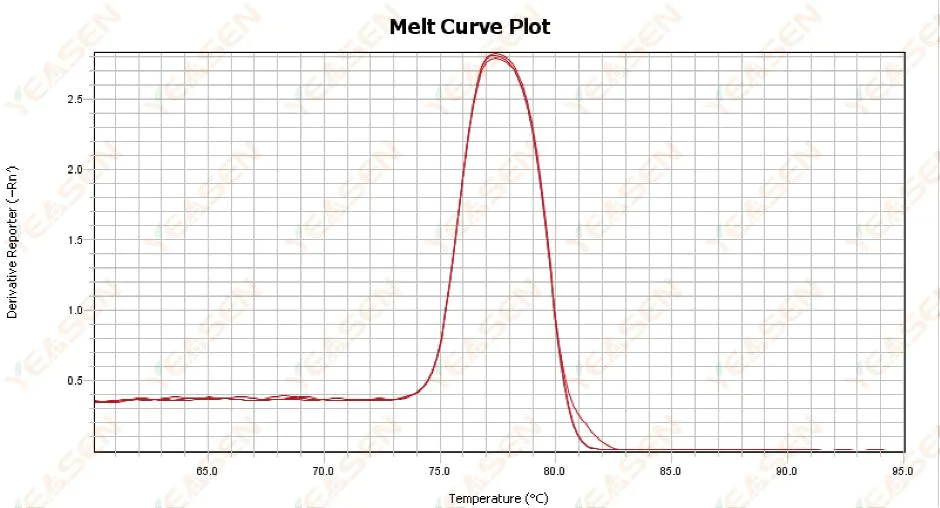
Figuur 13. Smeltcurveplot
Mogelijke oorzaken:
1) Het heeft te maken met de samenstelling van het reagens en het instrumentmodel.
2) Aanwezigheid van niet-specifieke amplificatie van fragmenten van vergelijkbare grootte.
Oplossingen:
1) De temperatuurspanne van piekbegin tot piekuit is niet hoger dan 7°C. De resultaten worden als bruikbaar beschouwd, d.w.z. enkele piek.
2) Om de bepaling te ondersteunen, werd agarose-elektroforese met een hoge concentratie (bijv. 3% agarose) uitgevoerd.
3.2 De smeltcurve is dubbel piekend en de onderste piek Tm ligt vóór 80°C
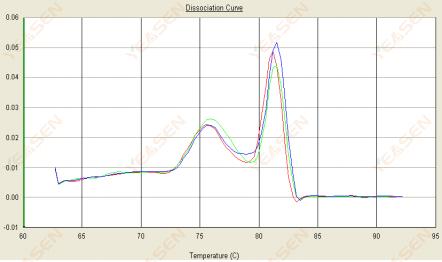
Figuur 14. Dissociatiecurve
Mogelijke oorzaken:
Aanwezigheid van primerdimeren.
Oplossingen:
Verhoog de gloeitemperatuur, verlaag de primerconcentratie of herontwerp de primers.
3.3 De smeltcurve heeft een dubbele piek en de onderste piek Tm is na 80°C
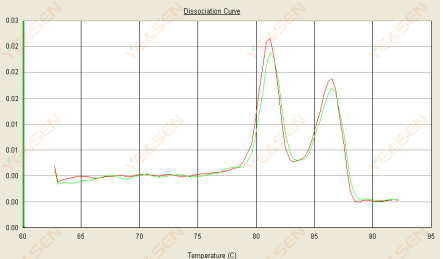
Figuur 15. Dissociatiecurve
Mogelijke oorzaken:
1) Een slechte primerspecificiteit leidt tot de amplificatie van niet-specifieke producten.
2) gDNA-verontreiniging.
Oplossingen:
1) Controleer de primerspecificiteit, herontwerp indien deze slecht is.
2) Bevestiging door NRC negatieve controle, indien van toepassing, moet opnieuw worden voorbereid in de template.
3.4 Rommelige smeltcurvepieken
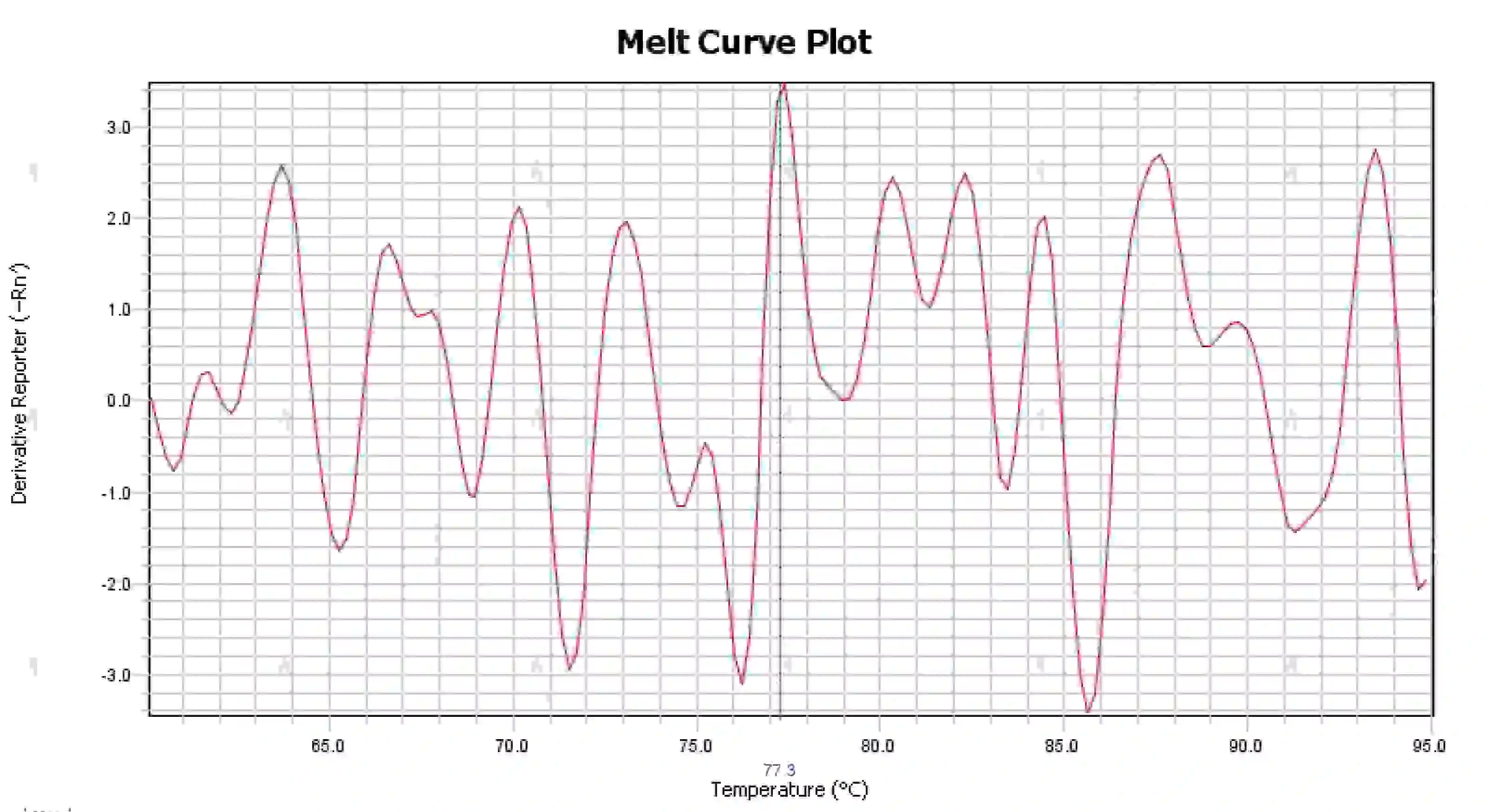
Figuur 16. Smeltcurveplot
Mogelijke oorzaken:
1) Verontreiniging van het reactiesysteem.
2) Blootstelling van reagentia aan fel licht of hoge temperaturen leidt tot reagensfalen.
3) Het instrument is al geruime tijd niet meer gekalibreerd.
4) Mismatch tussen verbruiksartikelen en instrumenten.
Oplossingen:
1) Door de NTC- en NRC-resultaten te combineren om contaminatie te bevestigen, wordt aanbevolen om contaminatie uit water, primers, enzymen en de omgeving één voor één uit te sluiten.
2) Het is aan te raden om vergelijkende experimenten uit te voeren met nieuwe reagentia.
3) Regelmatig onderhoud aan de instrumentkalibratie wordt aanbevolen.
4) Controleer of aan de vereisten voor verbruiksartikelen van het bijbehorende instrument is voldaan.
3.5 Onjuiste pieken aan de voorkant van de smeltcurve
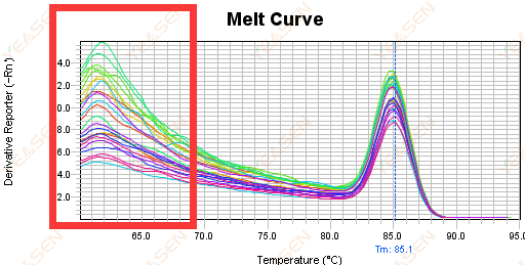
Figuur 17. Smeltcurveplot
Mogelijke oorzaken:
Rox-concentratie komt niet overeen met het model.
Oplossingen:
Het is raadzaam om de Rox-correctie te annuleren om te zien of de smeltcurve normaal is.
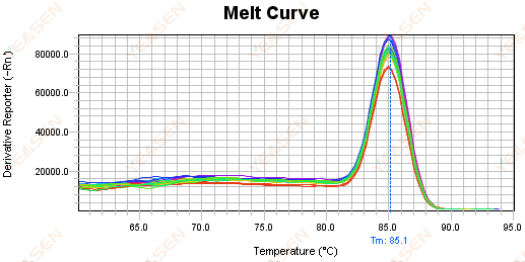
Figuur 18. Smeltcurveplot
4. Bestelgegevens
Tabel 1. Bestelgegevens
| Productnaam | Productcode | Specificatie |
| Hieff Unicon™ Universal Blue qPCR Master Mix (op basis van kleurstof) | 11184ES03 | 1 ml |
| 11184ES08 | 5×1 ml | |
| 11184ES50 | 50×1 ml | |
| 11184ES60 | 100×1 ml |
5. Gerelateerde producten
De producten die door
Tabel 2. Gerelateerde producten
| Productpositionering | Productnaam | Productcode |
| GDNA-verwijdering en reverse transcriptie in één stap | Hifair™V eenstaps RT-gDNA-digestie SuperMix voor qPCR (Navraag) | 11142ES |
| Mastermix met hoge gevoeligheid (met gDNA-verwijderingsstap) | Hifair™ III 1e streng cDNA-synthese SuperMix voor qPCR (gDNA-digestor plus) | 11141ES |
| Kit met hoge gevoeligheid (met gDNA-verwijderingsstap) | Hifair™ III 1e streng cDNA-synthesekit (gDNA-digestor plus) (Navraag) | 11139ES |
| Gewone kwantitatieve mastermix (probe-methode) | Hieff™ qPCR TaqMan Probe Master Mix (Navraag) | 11205ES |
6. Over het lezen
DNase I en hun toepassingen in de biomedische wetenschap
De richtlijn voor reverse transcriptaseselectie
