Hoe kiest u uw NGS-adapters?
NGS Adapter, een essentieel onderdeel van de next-generation sequencing bibliotheek, speelt een rol bij het verbinden van het geteste DNA fragment en de Flow cel (Sequencing chip). De efficiëntie van de verbinding is een belangrijke factor bij het bepalen van de kwaliteit en opbrengst van de bibliotheek. Dus wat is een NGS adapter? Wat zijn de meest voorkomende typen NGS adapters? En hoe kiest u de juiste NGS adapters voor uw sequencing platforms?
1. Wat is een NGS-adapter?
2. Met welke factoren moet u rekening houden bij het kiezen van de NGS-index?
3. Wat zijn de meest voorkomende typen indexen?
4. Wat zijn de meest voorkomende typen NGS-adapters?
- UMI-adapter
- Volledige adapters
- Onvolledige adapters
- Tn5-adapters
5. Hoe kiest u de juiste NGS-adapters voor uw sequencingplatforms?
6. Over Lezen
1. Wat is een NGS-adapter?
NGS Adapter, een serie adapters in sequencing, is een korte nucleotidesequentie met een bekende sequentie. Het is geligeerd aan beide uiteinden van het doelnucleïnezuurfragment. Tijdens sequencing start het met sequencing door te hybridiseren met de bekende sequentie op de Flow-cel om de bibliotheek te combineren met de chip. Dus wat is de structuur van de NGS-adapter?
Als we het Illumina-platform als voorbeeld nemen, kan een NGS-adapter in drie delen worden verdeeld:
P5 en P7: Sequentie gecombineerd met P5- en P7-einden op de Flow-cel om de bibliotheek op de sequentiechip te fixeren, waardoor clusterreactie via Bridge-PCR wordt vergemakkelijkt.
Rd1 SP en Rd2 SP (Read1/Read2 sequencing primer): Bindende regio's van sequentieprimers, die de positie aangeven waar de sequentie begint te worden gelezen.
Index (ook wel barcode genoemd): een bekende synthetische sequentie die wordt gebruikt om verschillende monsters te onderscheiden bij het sequencen van de gemengde bibliotheek.
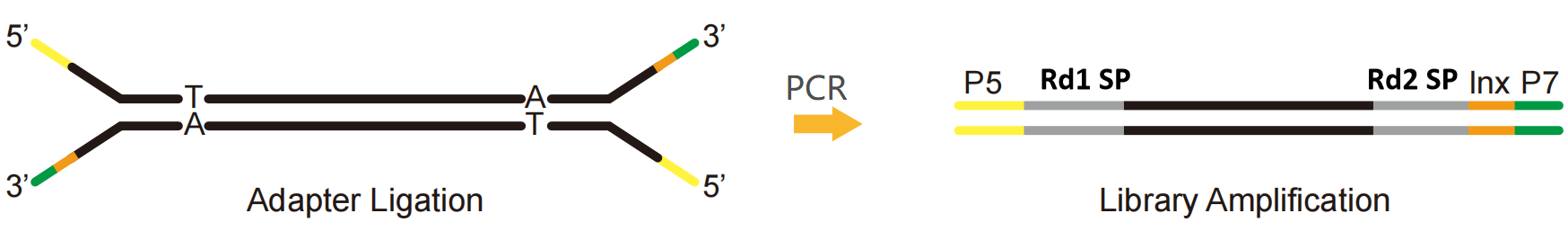
Afbeelding. 1 Illumina-platformbibliotheek met één index

Afbeelding. 2 MGI-platform single-end indexbibliotheek
Met de toename van de sequencing throughput kunnen meerdere samples tegelijkertijd worden gesequenced. Het is dus vooral belangrijk om verschillende samples te onderscheiden. Zoals eerder vermeld, worden indexsequenties van NGS-adapters gebruikt om verschillende samples te onderscheiden in next-generation sequencing (NGS). Met welke factoren moet u rekening houden bij het kiezen van de index? Lees verder...
2. Met welke factoren moet u rekening houden bij het kiezen van een index?
De index is over het algemeen 6nt-18nt lang en is verdeeld in een enkele index en een dubbele index op basis van het aantal indexen. De dubbele index bevindt zich aan beide uiteinden van het te testen fragment. Basebalans en fluorescentiebalans moeten in overweging worden genomen bij het selecteren van een indexcombinatie.
Basisbalans verwijst naar de balans tussen meerdere indexen, in plaats van de basisbalans in een enkele index. Het moet worden beschouwd vanuit zowel basistypen als basisdistributie. Het combinatieprincipe houdt in dat de vier bases A/T/C/G in dezelfde groep indexen moeten worden opgenomen en dat het aandeel van deze vier bases dicht bij elkaar ligt, namelijk respectievelijk ongeveer 25%.
Fluorescentiesignaalbalans verwijst naar de keuze om de balans van fluorescentiesignalen te verzekeren wanneer de basisbalans niet gegarandeerd kan worden. In de 4-kanaals sequencer in het Illumina-platform is dG/dT gelabeld met groene fluorescentie en dC/dA is gelabeld met rode fluorescentie. Tijdens het sequensen moeten er in elke cyclus zowel groene als rode fluorescentiesignalen aanwezig zijn om een succesvolle sequens te garanderen. Daarom moet bij het selecteren van de index rekening worden gehouden met de balans tussen het groene en het rode signaal.
3. Wat zijn de meest voorkomende typen indexen?
De meest voorkomende dubbele indexen zijn onder andere Unique Dual Index (UDI), Unique Dual Barcode (UDB) en Combined Dual Index (CDI), die indexhopping en verkeerde toewijzingen aanzienlijk verminderen.
UDI&UDB: de indexen aan beide uiteinden corresponderen één-op-één, zijn in groepen ontworpen en kunnen aan beide uiteinden kruisverifieerbaar zijn;
Een Stubby UDI Primer Kit voor Illumina geleverd door
CDI: de indexen aan beide uiteinden kunnen volgens bepaalde vereisten worden gecombineerd om een dubbelzijdige indexbibliotheek te vormen;
Een 384 CDI Primer voor Illumina, Set1-Set2 geleverd door
Om de doorvoer en de versterkingsefficiëntie te verbeteren en de sequentiekosten te verlagen, introduceerde Illumina de Array Flow Cell (PFCT) en de exclusieve versterkingstechnologie (ExAmp) voor Novaseq en andere sequencers met een hoge doorvoer, maar versterkte onbedoeld het fenomeen van mismatch tussen monsterlabels en indexhopping.

Figuur 3 Verschillende instrumentmodellen van Illumina maken gebruik van niet-gepatroneerde stroomcellen of gepatroneerde stroomcellen
Om het index hooping-probleem te compenseren dat werd benadrukt door de sequencingplatforms zoals HiSeq3000/4000, HiSeq X Series en NovaSeq, stelde Illumina de strategie voor om de index aan beide uiteinden van de bibliotheek te plaatsen, wat bilaterale verificatie kan uitvoeren en de niet-overeenkomende adapters kan elimineren. Bij gebruik van de unieke indexen aan beide uiteinden, zal de index error allocation rate worden verlaagd tot 0,01%. Vergeleken met de vorige conventionele index permutatiegroepcombinatiemethode, zal index hopping met twee ordes van grootte worden verminderd.
Bij de constructie van een PCR-vrije bibliotheek is een single-end indexadapter beschikbaar. De labelmismatch wordt voornamelijk veroorzaakt door sequentiefouten. Over het algemeen is de labelmismatchratio laag (gemiddeld 0,0004%, tot 0,001%). Bij de constructie van een gerichte capturebibliotheek wordt het crosstalkprobleem echter versterkt omdat meerdere stappen leiden tot labelmismatch en de UDI/UDB/CDI-adapters meestal worden gebruikt.
4.Wat zijn de meest voorkomende typen NGS-adapters?
Met de ontwikkeling van sequencingtechnologie zijn er steeds meer soorten adapters, zoals single index/double index adapters (zoals genoemd in sectie 3), UMI adapters, transposase adapters, complete/incomplete adapters, etc., die geschikt zijn voor verschillende toepassingsscenario's. Dit deel sorteert deze adapters systematisch om u de basis te geven voor adapterselectie.
4.1 UMI-adapter
Unique molecular identifier (UMI) adapter is een edge tool voor low-frequency mutation detection en absolute kwantificering. UMI is een willekeurige synthetische sequentie met een bekende sequentie. Het kan worden ontworpen als een volledig willekeurige nucleotideketen, gedeeltelijk gedegenereerde nucleotideketen of vaste nucleotideketen.De lengte is meestal 10nt (single-ended UMI) of 5-8nt (double-ended UMI). De functie ervan is om de toestand van DNA-fragmenten te bevriezen vóór amplificatie, en elk DNA-molecuul komt overeen met een UMI. Daarom kan het tijdens de analyse van bioinformatica DNA-sjablonen van verschillende bronnen onderscheiden, onderscheiden welke vals-positieve mutaties zijn veroorzaakt door willekeurige fouten in het proces van PCR-amplificatie en -sequencing, en welke daadwerkelijk door patiënten worden gedragen, om de achtergrondruis eruit te filteren, de nauwkeurige detectie van laagfrequente en extreem laagfrequente mutaties te realiseren en absolute kwantificering van verschillende DNA-moleculen uit te voeren. Het wordt veel gebruikt bij de detectie van laagfrequente mutaties, met name op het gebied van tumoronderzoek.

Figuur 4 Schematisch diagram van UMI-adapter structuur van het Illumina-platform
4.2 Complete adapters
Complete adapters, een noodzakelijk product voor een PCR-vrije bibliotheek, bevatten alle sequenties die nodig zijn voor sequenties, zoals P5, P7, RdS1 en RdS2 in het Illumina-platform, en indexeren ook sequenties en UMI-sequenties volgens de vereisten voor sequenties. Met de complete adapters kan het direct worden gesequenced zonder andere adapters via PCR te introduceren. Complete adapters kunnen dus worden gebruikt om een PCR-vrije bibliotheek te bouwen. De PCR-vrije bibliotheken kunnen PCR-amplificatiebias, foutpercentage en sequentieduplicatie verminderen, waardoor de dekking van sommige regio's met een hoge GC of hoge AT die veel worden gebruikt in populatiegenoomonderzoek, wordt vergroot.
Een compleet adapterproduct voor het Illumina-platform geleverd door
Een compleet adapterproduct voor het MGI-platform geleverd door
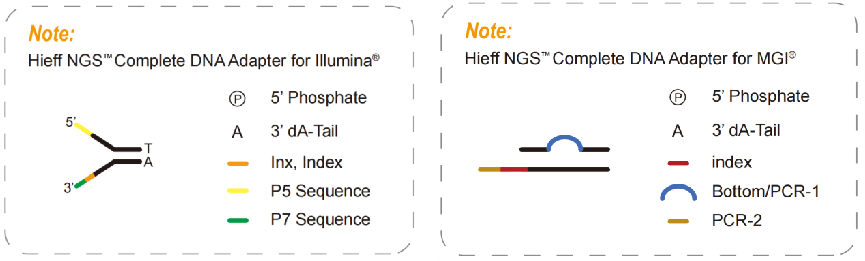
Figuur 5 Compleet adapterdiagram
4.3 Onvolledige adapters
Incomplete adapters moeten andere sequenties introduceren door PCR na de adapterligatie om een complete adapter te vormen. Ze worden gekenmerkt door een hoge verbindingsefficiëntie en een hoge effectieve bibliotheeksnelheid. Het PCR-proces is een verrijkingseffect voor de complete bibliotheek om de concentratie van de effectieve bibliotheek te garanderen, en kan ook double-ended indexen en UMI-sequenties introduceren.
4.4 Tn5-adapters
Tn5-adapters verbinden een deel van de adaptersequentie met beide uiteinden van de DNA-fragmenten via de restrictie-endonuclease-activiteit van Tn5. Ze zorgen ervoor dat fragmentatie en adapterligatie gelijktijdig worden uitgevoerd om tijd en monsters te besparen. Ten slotte worden de rest van de linkersequentie, index, UMI en andere sequenties geïntroduceerd door PCR om een complete bibliotheek te vormen. Het kan worden gebruikt om een Cut&tag-bibliotheek te bouwen.
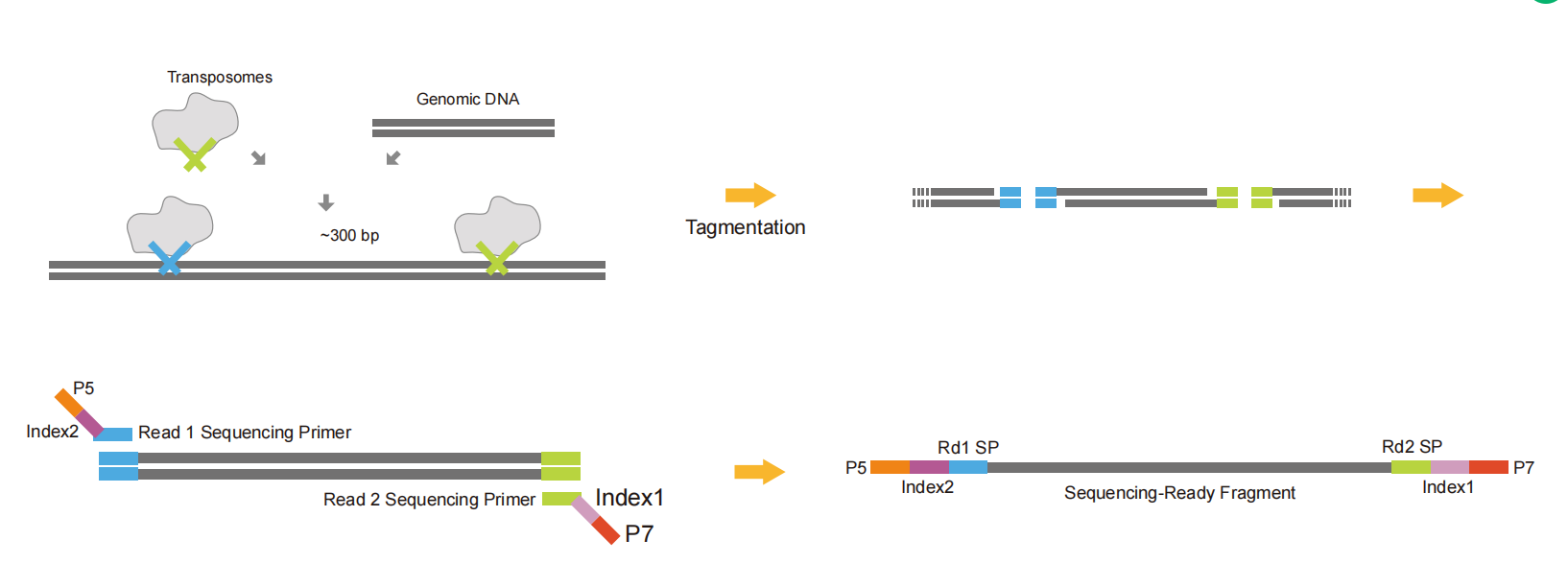
Figuur 6 Schematisch diagram van de constructie van de Tn5-adapterbibliotheek
5. Hoe kiest u de juiste NGS-adapters voor uw sequencingplatforms?
Momenteel zijn er twee gangbare sequencingplatforms: Illumina en MGI.
Wat het Illumina-platform betreft, worden de Illumina NGS-adapters geleverd door
Complete en UDI NGS-adapters hoeven zich geen zorgen te maken over koppelingsproblemen, geschikt voor klanten die eenvoudig te gebruiken willen zijn; CDI NGS-adapters hebben minder buizen en een klein formaat, wat geschikt is voor klanten die gemakkelijk willen opbergen en meenemen. PCR-vrij vereist het gebruik van complete NGS-adapters.
Voor Illumina
| InTube | Hoog NGS® DNA Lib-voorbereiding 384 CDI Primer voor Illumina, Set 1 (8*12, 96 index) | 12412ES |
| Hoog NGS® DNA Lib-voorbereiding 384 CDI Primer voor Illumina, Set 2 (8*12, 96 index) | 12413ES | |
| Hieff NGS® RNA Lib Prep 384 CDI Primer voor Illumina, Set 1 (96 index) | 12414ES | |
| Hieff NGS® RNA Lib Prep 384 CDI Primer voor Illumina, Set 1 (96 index) | 12415ES | |
| In plaat | Hieff NGS® Stubby UDI Primer Kit voor Illumina (1-384 Index) Set 1-4 | 12407ES |
| Hieff NGS® Stubby UDI Primer Kit voor Illumina Set1(96 wells plaat, 1-96 index) Reeks 1 | 12327ES | |
| Hieff NGS® Stubby UDI Primer Kit voor Illumina Set2(96-wells plaat, 97-192 index) Setje 2 | 12328ES | |
| Hieff NGS® Stubby UDI Primer Kit voor Illumina Set3(96-wells plaat, 193-288 index) Reeks 3 | 12329ES | |
| Hieff NGS® Stubby UDI Primer Kit voor Illumina Set4(96-wells plaat, 289-384 index) Setje 4 | 12330ES |
Voor MGI
| Hieff NGS™ Dubbele UMI UDB-adapterkit voor MGI, Set1/Set2 | 96 soorten index | |
| Hoog NGS™ Complete adapterkit voor MGI, Set1/Set2/Set3 (Vraag) | 13360ES | 8 soorten index, 41-48 |
| 13361ES | 16 soorten index, 57-72 | |
| 13362ES | 96 soorten index, 1-96 |
Over Lezen
Belangrijke enzymen betrokken bij de constructie van de NGS-bibliotheek
Hoeveel weet u over NGS-gerelateerde technologie?
Verschillende soorten magnetische kralen in NGS: DNA\RNA\mRNA magnetische kralen
NGS-bibliotheekkwantificering: snelle en nauwkeurige qubit of precieze qPCR?Alles wat u nodig hebt!
