Você aprendeu como construir uma biblioteca livre de PCR?
No sequenciamento de alto rendimento, a construção convencional de bibliotecas requer amplificação por PCR. Por um lado, é para amplificar amostras de DNA de rastreamento e aumenta o rendimento da biblioteca. Por outro lado, pode amplificar sinais fluorescentes, tornando mais fácil para os sequenciadores capturarem e identificarem sinais fluorescentes para melhorar a precisão do sequenciamento. No entanto, a PCR é como uma "espada de dois gumes". Ao resolver o problema do baixo volume inicial da amostra e amplificar o sinal fluorescente, também introduz erros e vieses de amplificação e não pode apresentar perfeitamente a sequência do genoma "Verdadeira face". Além disso, a polimerase de PCR tem um certo viés de amplificação. Algumas regiões, especialmente GC alto ou GC baixo, estrutura secundária e outras regiões, têm baixa eficiência de amplificação e são difíceis de cobrir. Também introduzirá um grande número de InDels errados e trará um grande número de Duplicação. , causando um desperdício de volume de dados de DNA e aumentando o custo do sequenciamento. Portanto, a introdução da tecnologia PCR-Free não só tem as vantagens da PCR, mas também supera as deficiências da PCR. Ela não só pode trazer bibliotecas de alta qualidade com alta sensibilidade, e realizar a detecção de amostras de traços, mas também ter alta precisão e reduzir a carga de trabalho de Validação de acompanhamento para estudos de variantes. Então, o que exatamente é a biblioteca PCR-Free e quais são suas vantagens?
1. O que é a biblioteca PCR-Free?
2. Quais são as vantagens da tecnologia PCR-Free?
3. Exibição de dados de PCR-Free
4. Perguntas frequentes
5. Informações do produto
6. Sobre a leitura
1. O que é a biblioteca PCR-Free?
A tecnologia de sequenciamento de próxima geração é a tecnologia mais comumente usada em pesquisas de sequenciamento de alto rendimento na biologia molecular moderna. O sequenciamento tradicional de primeira geração não pode mais atender totalmente às necessidades dos pesquisadores. O resequenciamento do genoma de organismos modelo e o sequenciamento do genoma de organismos não modelo exigem mais custos. Tecnologia de sequenciamento de baixo, alto rendimento e mais rápida, dando origem à tecnologia de sequenciamento de segunda geração. O princípio central da tecnologia de sequenciamento de segunda geração é o sequenciamento por síntese, ou seja, determinar a sequência de DNA capturando as informações da base recém-sintetizada com rótulos finais. O princípio principal da tecnologia de sequenciamento de segunda geração para sequenciamento de DNA é primeiro fragmentar o DNA, reparar a extremidade do DNA fragmentado e, em seguida, adicionar adaptadores específicos em ambos os lados e, em seguida, usar métodos diferentes para gerar milhões de PCRs espacialmente fixados. Para matrizes clonais, os dados de sequenciamento são obtidos usando hibridização de primers e reações de extensão enzimática para criar imagens dos rótulos fluorescentes incorporados por cada reação de extensão.
O sequenciamento de DNA por sequenciamento de próxima geração inclui principalmente dois processos: preparação da biblioteca e sequenciamento na máquina. Durante a preparação da biblioteca, fragmentos genômicos interrompidos aleatoriamente são tipicamente amplificados por PCR padrão. No entanto, para alguns modelos especiais, há fatores como estrutura secundária complexa ou baixa estabilidade térmica, que afetam a preferência de amplificação do modelo de PCR, portanto, nem todas as sequências genômicas podem ser igualmente refletidas na biblioteca de amplificação de PCR. Especialmente para alguns modelos com alto teor de GC ou alto teor de AT, às vezes é difícil usar o método de PCR para amplificar para construção de biblioteca. Não há diferença entre a biblioteca sem PCR e a biblioteca de PCR convencional ao sequenciar na máquina, exceto que a PCR não é realizada durante o processo de construção da biblioteca. A biblioteca sem PCR pode teoricamente melhorar a distribuição de leituras de dados e gerar uma cobertura genômica mais uniforme.A biblioteca livre de PCR é um suplemento ao método de construção de biblioteca. Como é preparada diretamente em uma biblioteca onboard sem amplificação de PCR, ela pode melhorar a cobertura de algumas regiões de GC alto ou AT alto, reduzindo assim as bases de erro introduzidas por PCR, viés de dados e repetição de sequência.
As principais etapas da construção convencional da biblioteca NGS são fragmentação do genoma, reparo final, adição de adaptador, PCR e amplificação do sinal antes do sequenciamento. Então, o que dizer da construção da biblioteca PCR-Free? Como o nome sugere, é um processo de construção de biblioteca que não requer PCR. As principais etapas da construção da biblioteca são fragmentação do genoma, modificação final, adição de linker e controle de qualidade da biblioteca. Mas, na verdade, isso só pode ser considerado PCR-Free no processo de construção da biblioteca. O PCR-free real deve ser um processo da construção da biblioteca ao sequenciamento sem amplificação da biblioteca (como a tecnologia de sequenciamento de molécula única) ou uma tecnologia de sequenciamento sem acúmulo de erros de amplificação de PCR (como o DNBseq da MGI).

Figura 1. Fluxograma da construção de biblioteca convencional e construção de biblioteca PCR-Free
2. Quais são as vantagens da tecnologia PCR-Free?
No processo de construção de biblioteca de rotina, a enzima de amplificação pode introduzir erros. Por meio da amplificação cíclica, esses erros serão acumulados e amplificados, reduzindo a fidelidade da replicação da sequência de DNA. Além disso, a DNA polimerase também tem um certo viés de amplificação, especialmente para regiões com grandes diferenças no conteúdo de GC ou estruturas secundárias, resultando em baixa eficiência de amplificação e baixa uniformidade de cobertura; ao introduzir InDels errados, também trará um grande número de Duplicação causa desperdício de volume de dados de DNA e aumenta os custos de sequenciamento. A tecnologia PCR-Free pode evitar bem os problemas mencionados acima introduzidos pela amplificação por PCR na construção de biblioteca convencional. No processo de construção e sequenciamento de biblioteca, se houver um processo de amplificação por PCR, ele terá um impacto maior na uniformidade da cobertura do genoma. Para modelos de DNA, alguns têm estruturas secundárias complexas e alguns têm grandes diferenças na estabilidade térmica. Esses fatores afetarão a eficiência da amplificação por PCR. Portanto, no processo de amplificação por PCR, não é possível garantir que todos os fragmentos genômicos possam obter a mesma eficiência de amplificação, mas há um viés de amplificação óbvio, como baixa cobertura em regiões de GC alto ou GC baixo do genoma. No entanto, não há PCR em todo o processo de sequenciamento PCR-Free. Comparado com a construção da biblioteca de PCR, a cobertura de regiões de GC alto e regiões de repetição AT/TA do genoma é melhorada. As vantagens específicas são as seguintes.
2.1 Processo de preparação de biblioteca simplificado, economizando tempo
A preparação da biblioteca sem PCR elimina a necessidade de amplificação por PCR e outras etapas, agilizando o processo de preparação da biblioteca e economizando tempo.
2.2 Redução do custo de preparação da biblioteca
No processo convencional de preparação de biblioteca, enzimas caras de alta fidelidade são usadas para amplificação de PCR para garantir a autenticidade da sequência. Ao mesmo tempo, o PCR-Free elimina a etapa de amplificação de PCR, reduzindo os custos de preparação da biblioteca.
2.3 Reduzir o viés de amplificação
As enzimas de amplificação de DNA têm certos vieses de amplificação, especialmente para moldes com diferenças significativas no conteúdo de GC ou estrutura secundária. Portanto, o PCR-Free pode efetivamente evitar o viés de amplificação causado pela polimerase.
2.4 Sem duplicação de PCR
O PCR-Free pode reduzir leituras duplicadas e aumentar as taxas de mapeamento exclusivo e a utilização efetiva de dados.
2.5 Parâmetro de erro reduzido
A amplificação por PCR tem maior probabilidade de introduzir erros de replicação de inserção e exclusão, resultando em uma maior taxa de erro de sequenciamento Indel, enquanto a PCR-Free pode efetivamente evitar a geração e o acúmulo de tais erros.
3. Exibição de dados de PCR-Free
Kit de preparação de biblioteca de DNA livre para PCR Hieff NGS™ Ultima Pro V2 (Cat# 12196ES) tem uma taxa de conversão de biblioteca melhor do que produtos concorrentes.
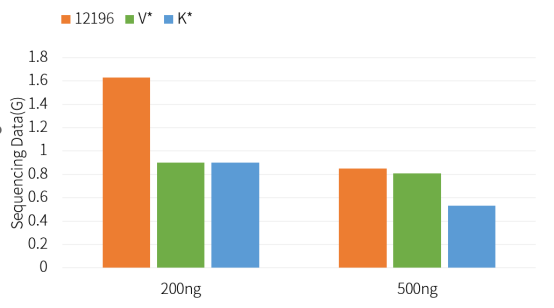
Figura 2. Quantidade de dados de sequenciamento off-machine para construção de biblioteca PCR-Free
4. Perguntas frequentes
P: Que tipos de amostras são adequadas para bibliotecas sem PCR?
R: Algumas amostras com estruturas secundárias complexas, alto GC e preferência por PCR podem escolher uma biblioteca sem PCR para construir.
P: Qual é o tamanho geral do fragmento da biblioteca livre de PCR?
R: A biblioteca livre de PCR em si não tem requisitos especiais quanto ao tamanho do fragmento. A biblioteca de produtos de PCR é construída de acordo com o tamanho do produto de PCR. Para a biblioteca genômica, é recomendado que seja em torno de 350 pb, para que a qualidade dos dados da biblioteca seja relativamente boa.
5. Informações do produto
O produto que a
Tabela 1. Informações do produto
| Produto Nome | Cat. Não. | Tamanho |
| Kit de preparação de biblioteca de DNA livre para PCR Hieff NGS™ Ultima Pro V2 | 12196ES24/96 | 24/96T |
6. Sobre a leitura
A solução para a preparação da biblioteca NGS a partir de amostras de DNA
O quanto você sabe sobre tecnologia relacionada a NGS?
